Face aux arguments commerciaux développés en faveur de tels ou tels séquenceurs de paillasse à haut débit, l’étude menée par Loman et al., Nature Biotechnology propose une comparaison des performances du 454 GS Junior (Roche), du MiSeq (Illumina) et du PGM Ion torrent (Life Technologies), à partir du séquençage de novo d’un isolat d’E.coli O104 :H4 responsable de l’intoxication alimentaire en Allemagne (Mai 2011). Pour cela, le génome de référence a été généré à l’aide du GS FLX (Roche) et permettra d’évaluer l’efficacité des séquenceurs et algorithmes d’assemblages associés.
Sur la base des caractéristiques techniques établies en 2011, les trois plateformes ont permis de générer un draft exploitable du génome bactérien sans pour autant parvenir à le reconstituer à 100%. Par conséquent, il est impossible de désigner un séquenceur comme étant celui de choix, chaque plateforme étant un compromis entre avantages et inconvénients.
Deux « runs » de 454 GS Junior (associés au bénéfice de reads de 600 bases) ont permis d’atteindre 96,28% de couverture par rapport au génome de référence (Contre 96,05% pour le MiSeq et 95,4% pour le PGM Ion torrent).
Malgré une capacité de séquençage du PGM Ion torrent supérieure à celle du 454 GS Junior (100Mb contre 70Mb) et une précision de séquençage voisine (Q20, soit 1 erreur toutes les 100 bases), la technologie reposant sur les semiconducteurs fait face à ses limites au niveau de la gestion des homopolymères et accessoirement d’une longueur de reads de 100 bases.
Vingt fois supérieur au 454 GS Junior en terme de quantité de bases décodées, un « run » de MiSeq ne parvient qu’à talonner les performances d’un double « run » de la technologie Roche. Bien qu’étant la seule à gérer les homopolymères et la seule doter d’une précision à Q30 (soit 1 erreur toutes les 1000 bases), la technologie Illumina faillit sur ses tailles de « reads » qui ne permettent pas une gestion optimum des séquences répétées lors de l’assemblage.
L’approche analytique est également évoquée et selon le système d’assemblage utilisé (Velvet, CLC, MIRA), de nombreuses différences sont observées.
Enfin, ce document est l’occasion de rassembler des notions assez pragmatiques comme le tarif des plateformes (et annexes associées), la durée d’un « run » de séquençage ou encore la quantité de données générées.
Ceci étant, on est en droit d’émettre quelques réticences quant à la pertinence de cette étude qui base son comparatif sur une application (séquençage de novo) qui n’est pas la vocation première de cette catégorie de séquenceurs. Par ailleurs, dans le laps de temps où cette étude a été réalisée et la parution des résultats, la technologique du PGM Ion torrent a évolué. Testé sur les bases d’une puce 316 (100Mb) et d’une technologie de 100 bases en longueur de « reads », le PGM bénéficie désormais de la puce 318 (1 Gb) associée à une technologie de reads de 200 bases. Sans tarder, Life technologies réagit (“Loman et al reflects the past, not the present”) et propose une mise à jour au travers de ce document.
Le séquençage massif parallélisé palliant les limites des anciennes méthodes, a depuis ces dernières années amorcé de nouvelles perspectives, comme l’étude d’altérations génomiques pour la compréhension de nombreuses maladies rares (Plan National Maladies Rares). De plus, la mise sur le marché des derniers séquenceurs de paillasse aux visées hospitalières (positionnement de marché de Ion torrent avec son PGM) contribue également au développement de nombreux diagnostics cliniques.
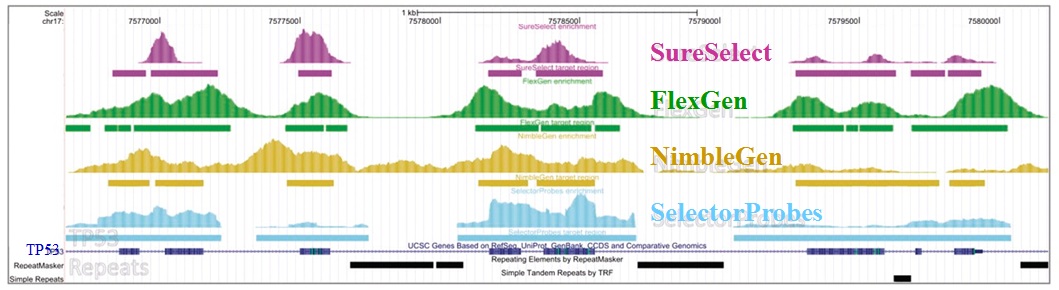
Face à cette révolution, il convient de faire face au goulot d’étranglement de l’analyse et de l’interprétation, en faisant appel à une stratégie plus appropriée que le séquençage de génome complet et qui consiste à focaliser les efforts de décodage sur les régions codantes enrichies (Exome, gènes cibles, etc…) par une étape préliminaire de d’enrichissement d’exons. Tout en contribuant à une diminution des coûts (on ne séquence que ce qui semble pertinent…), elle permet entre autre d’augmenter la profondeur de séquençage des régions d’intérêts, paramètre indispensable pour l’identification d’altérations génomiques.
Succinctement, les techniques d’enrichissement sont multiples et peuvent être regroupées en trois catégories sur la base de leur principe technique:
– Enrichissement par capture d’hybrides : La capture par hybridation des régions cibles est initiée à partir de la librairie de séquençage. Elle peut être effectuée soit en solution (billes magnétiques) soit sur support solide (microarrays). Récemment, de nombreuses solutions (commerciales ou « à façon ») ont été développées: SureSelect, Agilent – SeqCap EZ, Roche NimbleGen – Sequence Capture Arrays, Roche Nimblegen – FleXelect, FlexGen – MYselect, MYcroarray – TargetSeq, Life Technologies .
– Enrichissement par circularisation de fragments d’ADN : Cette méthode est supérieure à la précédente en terme de spécificité. En amont de la préparation de la librairie, les fragments d’ADNg (fragmentation enzymatique ou mécanique selon la méthode) sont enrichis via une sonde constituée d’une séquence universelle flanquée aux extrémités de séquences spécifiques de la région cible. HaloPlex, Agilent.
– Enrichissement par PCR : Cette approche intervient avant la préparation de la librairie et consiste grossièrement en une PCR multipléxée ciblant les régions d’intérêts. Une étape préliminaire de design des amorces est requise. SequalPrep, Life technologies. Tout juste disponible, Ion AmpliSeq Designer, Life Technologies propose un PCR Ultra-Plex (jusqu’à 1536 amplicons) avec un étape de digestion des amorces permettant de ne conserver que les régions d’intérêts lors du séquençage.
Une technologie récente et élégante faisant appel à la microfluidique et l’émulsion PCR permet l’amplification multipléxée (jusqu’à 20000) en micro-gouttelettes (une paire d’amorces par microréacteur) en un seul tube. Multiplicom, Fluidigm – RainDance.
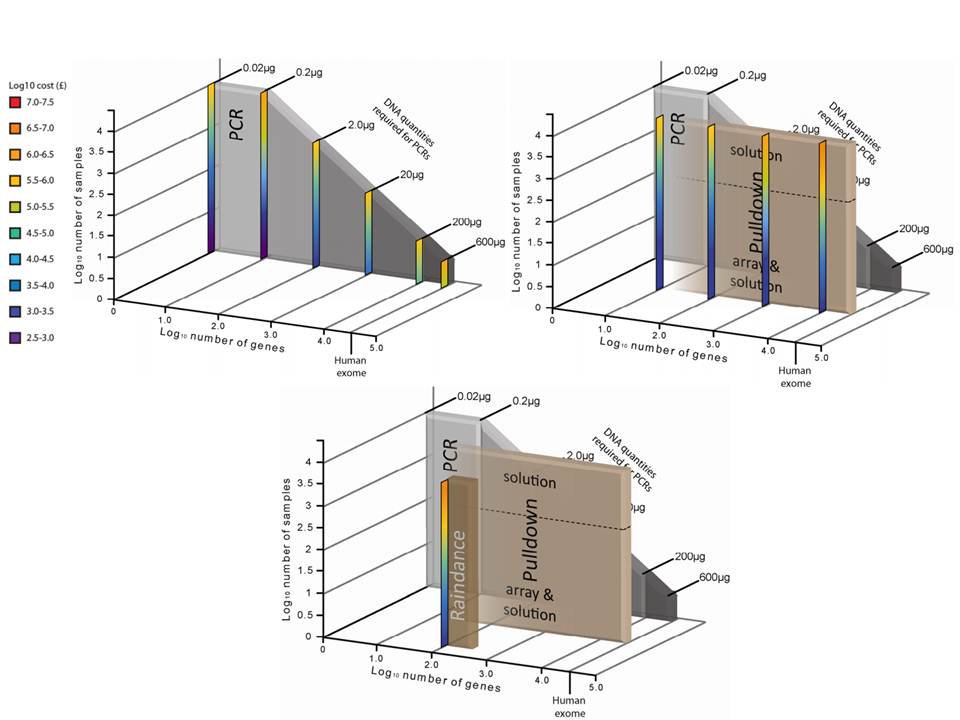
Ceci étant, les considérations fondamentales dans l’analyse de variants résident notamment dans:
– la profondeur de séquençage
– l’homogénéité de couverture des régions d’intérêt
– la reproductibilité de la méthode
– la quantité d’ADN « Input » requis
– le nombre d’échantillons à traiter
– le coût global
L’étude récemment menée par Florian Mertes et al. (nov. 2011) souligne l’importance de ces notions et fait le postulat de performances de captures de régions cibles significativement différentes selon la méthode d’enrichissement employée.
Il est impossible d’identifier une méthode d’enrichissement comme étant la meilleure notamment parce que ces approches sont en continuelle évolution et amélioration. Chacune d’elles répondent à des caractéristiques particulières et à des applications distinctes.
Si l’enrichissement par circularisation offre davantage de spécificité mais moins d’uniformité, l’inverse est aussi valable pour l’enrichissement par capture d’hybrides. Parmi les considérations dans le choix d’une méthode, le nombre d’échantillons et la taille des régions cibles sont essentiels. Si la capture d’hybrides est privilégiée pour l’analyse de mégabases (étude de l’exome) avec un nombre limité de cas, cette méthode sera délaissée au profit d’une approche PCR multipléxée pour l’étude d’un nombre restreint de régions cibles de petite taille appliquée à un grand nombre d’échantillons (Diagnostic).
L’association des méthodes d’enrichissement au séquençage haut débit offre des capacités technologiques ouvrant la voie vers de nombreuses perspectives. Toutefois, si l’émergence des séquenceurs de troisième génération s’accompagne à nouveau d’une diminution des coûts de séquençage, on est en droit de se demander si cette option restera aussi attractive.
Le réseau régional d’ingénieurs en bioinformatique de Lille et le PPF bioinformatique organisent lundi 14 Mai 2012 une conférence sur l’utilisation des outils GMOD (Generic Model Organism Database). Cette conférence se déroulera de 13h30 à 17h30 dans l’amphithéâtre de l’Institut de Biologie de Lille.
« Le projet GMOD a pour objectif de fournir une série d’outils génériques, clé en main, pour gérer et visualiser différents types de données biologiques. »
Deux interventions sont prévues :
Olivier Arnaiz du CGM Centre de Génétique Moléculaire (UPR 3404 Gif-sur-Yvette) introduira GMOD tout en se focalisant plus particulierement sur deux outils : Chado et Gbrowse. Chado est un schéma de base de données relationnelles qui permet d’intégrer des données « omiques » (génomiques, transcriptomices, séquençages…), tandis que GBrowse est un outil permettant de visualiser des annotations sur un génome.
Joelle Amselem de l’INRA – URGI (BIOGER Versailles) présentera un outil d’annotation développé à l’URGI à partir de modules GMOD. Deux exemples concrets d’utilisation seront développés.
Pour en savoir plus, vous pouvez télécharger le programme ici.
Si vous souhaitez des informations sur GMOD, je vous invite à consulter la présentation de GMOD.
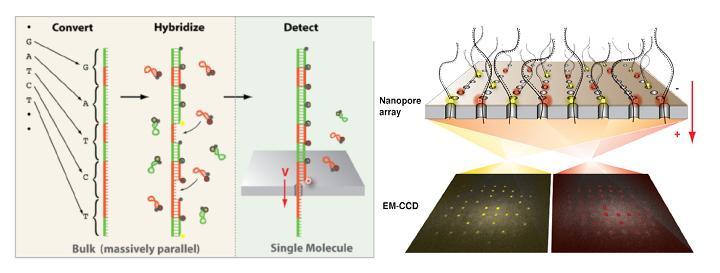
 Noblegen Biosciences, start-up localisée dans le Massachusetts, ambitionne de commercialiser pour 2014 « optipore » (pour « optical detection » et « nanopore ») , un séquenceur de paillasse de troisième génération combinant nanotechnologies et un système de lecture optique permettant de réduire drastiquement les coûts de séquençage. L’objectif est de conquérir le marché des laboratoires cliniques dans la perspective de l’émergence d’une médecine personnalisée et de proposer ainsi un séquençage de génome humain complet à faible coût et dans un temps record. Dans cette course engagée, la société américaine se trouve en bonne position.
Noblegen Biosciences, start-up localisée dans le Massachusetts, ambitionne de commercialiser pour 2014 « optipore » (pour « optical detection » et « nanopore ») , un séquenceur de paillasse de troisième génération combinant nanotechnologies et un système de lecture optique permettant de réduire drastiquement les coûts de séquençage. L’objectif est de conquérir le marché des laboratoires cliniques dans la perspective de l’émergence d’une médecine personnalisée et de proposer ainsi un séquençage de génome humain complet à faible coût et dans un temps record. Dans cette course engagée, la société américaine se trouve en bonne position.
A l’instar du PGM Ion torrent, une puce en silicium constitue le coeur de la machine et renferme des centaines de nanopores.  La molécule d’ADN unique native destinée à être séquencée est tout d’abord convertie en une nouvelle molécule synthétique transformant chaque base par une séquence nucléotidique spécifique correspondante. A chacune des quatre signatures nucléotidiques correspond un « molecular beacon » complémentaire fluorescent initialement inactivé par un système de « quencher ». Leurs hybridations au brin néoformé aboutit à un ADN double brin. la molécule est dirigé vers les nanopores par des échanges ioniques et en raison de la taille des orifices, les « molecular beacon » sont contraints à se deshybrider libérant cette fois une fluorescence, lue par un capteur photographique de type CMOS, qui est traduite en séquence.
La molécule d’ADN unique native destinée à être séquencée est tout d’abord convertie en une nouvelle molécule synthétique transformant chaque base par une séquence nucléotidique spécifique correspondante. A chacune des quatre signatures nucléotidiques correspond un « molecular beacon » complémentaire fluorescent initialement inactivé par un système de « quencher ». Leurs hybridations au brin néoformé aboutit à un ADN double brin. la molécule est dirigé vers les nanopores par des échanges ioniques et en raison de la taille des orifices, les « molecular beacon » sont contraints à se deshybrider libérant cette fois une fluorescence, lue par un capteur photographique de type CMOS, qui est traduite en séquence.
L’ensemble des études et preuves de faisabilité sur lesquelles repose la technologie de séquençage « optipore » sont décrites et détaillées au travers de l’article libre d’accès ci dessous: A ce jour, peu de caractéristiques techniques liées à cette plateforme filtrent mais Frank Feist, cofondateur de la société, annonce une capacité de séquençage de 500 Gb/heure. De plus, en raison de l’étape de conversion initiale, la taille des fragments serait limitée à 200 bases.
A ce jour, peu de caractéristiques techniques liées à cette plateforme filtrent mais Frank Feist, cofondateur de la société, annonce une capacité de séquençage de 500 Gb/heure. De plus, en raison de l’étape de conversion initiale, la taille des fragments serait limitée à 200 bases.
Un soutien de 4,2 millions de dollars en septembre dernier de la part du National Human Genome Research Institute devrait permettre à la société américaine de conforter cette avancée prometteuse.
La confusion entre mate-pair et paired-end, tant au niveau technologique (selon qu’on lise les notes techniques d’Illumina, de Roche ou de Life) que logiciel nous a mené à rédiger, en collaboration avec Ségolène Caboche, Bioinformaticienne à l’université de Lille2, une note technique dont le contenu est résumé ci-dessous :
– Genèse de la confusion entre mate-pair et paired-end
– Descriptions les deux approches, avec un focus sur les principales technologies de seconde génération de séquenceurs
– Traitement au niveau logiciel et conseils généralistes pour l’utilisation
Le document est consultable dans son intégralité sur notre blog :
Télécharger Paired-end versus mate-pair
Bonne lecture!
Ce post fait naturellement suite à celui dédié à la seconde génération de séquenceurs multi-parallélisés, et conserve la même approche, à savoir un tour d’horizon des technologies et une évocations des informations générales sur le sujet.
A l’instar du PGM de Ion torrent mis sur le marché depuis un an (10Mb – reads 100b – 06.2011 / 100Mb – reads 200b -11.2011 / 1Gb – reads 400b – prévu début 2012), la seconde génération de séquenceurs haut débit tend vers une production de reads de plus en plus longs et de moins en moins chère. Toutefois, on est en droit de se demander quelle sera leur pérennité face à la 3éme génération répondant à un cahier des charges assez similaire et la possibilité de bénéficier de nouvelles applications.
Le principe de la 3ème génération peut être symbolisé par le séquençage d’une molécule d’ADN sans étape de pré-amplification (contrairement à la génération actuelle type 454 Roche, SOLiD Life technologie, Ion Proton, PGM Ion torrent, HiSeq Illumina, …) en conservant l’incorporation de nucléotides, par cycles ou non ( dans ce dernier cas, le terme de « Séquençage d’ADN simple molécule en temps réel » est approprié).
Les technologies « SMS » pour « Single Molecule Sequencing » peuvent être regroupées selon trois catégories:
– Technologies de séquençage en temps réel impliquant la synthèse du brin d’ADN complémentaire via une ADN polymérase.
– Technologies de séquençage par détection des bases successives d’une molécule d’ADN au travers de nanopores.
– Technologies de séquençage basées sur des techniques de microscopie.
En combinant les dernières avancées dans la nanofabrication, la chimie de surface et l’optique, Pacific Biosciences (Pacbio RS) a lancé une plateforme technologique puissante appelée technologie de molécule unique en temps réel, ou « SMRT » pour « Single Molecule Real-time sequencing ». Parmi ses concurrents directs, Helicos Biosciences (Helicos) qualifié « tSMS » pour « True Single Molecule Sequencing ». Malgré le recours à une technologie analogue, la mention « Temps réel » auquel il échappe est simplement liée à une incorporation cyclique des nucléotides fluorescents.
D’autres technologies, à des degrés de développement plus ou moins avancé, sont dans les tuyaux et qui sait de Noblegen, Starlight, Cracker Bio, NABSys, Halcyon, ou autres… révolutionnera encore un peu plus cet univers du haut débit et suivra le chemin emprunté dernièrement par Oxford Nanopore …
 Depuis la publication de 2006, d’Astier and al., J. Am. Chem. Soc., la communauté scientifique attendait la révolution du séquençage 3ème génération que constituait la géniale idée d’Oxford Nanopore : à savoir l’alliance des nanotechnologies, du génie génétique le tout à des fins de séquençage (et plus si affinité). Ainsi, un pore d’alpha-hémolysine (d’environ 1 nanomètre) ou la porine A de Mycobacterium smegmatis ou bien encore du graphène ont été investigués pour devenir le pore idéal dans lequel, après clivage par une exonucléase des liaisons phosphodiester, les bases azotées dont l’enchaînement constitue le séquençage passe afin d’être analysées (par analysées on entend lues, la lecture étant basée sur la différence de conductance électrique liée à la nature chimique des bases azotées). Le pari, pour Oxford Nanopore réside dans le fait de trouver un mode de détection suffisamment sensible pour analyser puis par puis un signal généré par le passage d’une base au travers de pores (autrement appelés trous). La résolution de la technologie constituait un défit, sa faiblesse provenait (espérons que ce verbe se conjugue aujourd’hui au passé) de la rapidité de passage des bases à travers le pore (1 à 5 µs / base)…
Depuis la publication de 2006, d’Astier and al., J. Am. Chem. Soc., la communauté scientifique attendait la révolution du séquençage 3ème génération que constituait la géniale idée d’Oxford Nanopore : à savoir l’alliance des nanotechnologies, du génie génétique le tout à des fins de séquençage (et plus si affinité). Ainsi, un pore d’alpha-hémolysine (d’environ 1 nanomètre) ou la porine A de Mycobacterium smegmatis ou bien encore du graphène ont été investigués pour devenir le pore idéal dans lequel, après clivage par une exonucléase des liaisons phosphodiester, les bases azotées dont l’enchaînement constitue le séquençage passe afin d’être analysées (par analysées on entend lues, la lecture étant basée sur la différence de conductance électrique liée à la nature chimique des bases azotées). Le pari, pour Oxford Nanopore réside dans le fait de trouver un mode de détection suffisamment sensible pour analyser puis par puis un signal généré par le passage d’une base au travers de pores (autrement appelés trous). La résolution de la technologie constituait un défit, sa faiblesse provenait (espérons que ce verbe se conjugue aujourd’hui au passé) de la rapidité de passage des bases à travers le pore (1 à 5 µs / base)…
Une attente à émouvoir un James Dewey Watson, plusieurs publications en guise de démonstration de la faisabilité d’un tel séquençage, des appels de fonds ayant abouti à l’intégration d’Illumina au niveau du capital d’Oxford Nanopore (investissement en deux fois d’abord de 28 millions $ puis de 41 millions) ont maintenu en haleine les scientifiques avides de séquençage 3ème génération.
Oxford Nanopore est l’un des seuls acteurs européens, dans le monde du séquençage haut-débit où l’hégémonie californienne est écrasante… Alors que les Californiens (PacBio, Life, Illumina) communiquent à grand renfort d’annonces choc, Oxford Nanopore garde une part de mystère (aucune spécification réellement disponible à ce jour), nourrissant notre curiosité avec quelques publications scientifiques et quelques animations dont certaines vous sont présentées ci-dessous.
http://vimeo.com/18630569
Cette première animation reprend les différentes modalités de séquençage
http://vimeo.com/20289048
Avec la vidéo ci-dessus et celle ci-après, Oxford Nanopore réinvente le magnétoscope VHS à visée séquençage haut-débit…
http://vimeo.com/19288315
Finalement, le message à peine subliminal de ce court article : Oxford Nanopore est une société à surveiller de près… de plus en plus près.
Note : si les liens vers les vidéos ne fonctionnent pas essayer toujours en suivant celui-ci…
Dernier né de la gamme des séquenceurs de paillasse, le « Ion Proton Sequencer » (149.000 $) est le premier de sa catégorie à permettre le séquençage en quelques heures du génome humain et à faible coût (1.000 $). A l’instar du PGM Ion Torrent, cette nouvelle plateforme est basée sur la technologie des semi-conducteurs (et la détection de la libération d’ions H+ suite à la polymérisation de dNTP).
La puce « Ion Proton ™ I »,disponible mi-2012, aura une capacité de séquençage 1000 fois supérieure au premier format de puce 314 du PGM Ion torrent soit 10 Gb (davantage orientée pour le séquençage d’exome humain). Le second format (puce « Ion Proton ™ II ») , disponible environ six mois plus tard aura une capacité de 100 Gb (Séquençage Génome humain).
Compte tenu de l’apparente similarité entre les deux plateformes de chez Life technologies, l’évolution réside essentiellement dans l’architecture de la puce avec une augmentation du nombre de puits ( 165 millions de puits pour la puce « Ion Proton ™ I » et 660 millions pour la puce « Ion Proton ™ II » soit respectivement 100 et 1000 fois plus que la puce 314 du PGM ), l’une des bases de la capacité de séquençage (en complément de la longueur des reads générés).
Pour continuer dans notre tour d’horizon technologique des différentes méthodes de séquençage voici une revue faisant le point sur les différentes plateformes (plutôt dans le gros calibre). Certes, cette revue date de presque deux ans et écarte d’emblée les séquenceurs de paillasse, néanmoins les schémas, plutôt bien faits, permettent d’entrevoir les subtilités des diverses modalités de séquençage. En outre, cette revue propose une base comparative argumentée permettant de se faire une opinion éclairée des capacités des diverses plateformes (comme d’habitude vous avez accès à la publication en cliquant sur l’image ci-dessous)… bonne lecture.
Voici pour continuer sur les développements qui ont lieu sur la plateforme de séquençage de Ion Torrent, le PGM, une nouvelle note d’application qui ouvre de nouvelles perspectives dans le champ du séquençage haut-débit : le fait de pouvoir très bientôt séquencer des fragments de 400 bases dans les deux sens. Ainsi, le PGM de Ion Torrent sera la seule technologie capable de fournir sur des reads plutôt longs un séquençage paired end (sachant que le kit et protocole associé « mate paire » est d’ores et déjà disponible). Evidemment, ce développement vient contrecarrer une critique qui peut être portée sur cette plateforme de séquençage : sa « justesse » de séquençage sur séquences brutes générées à Q20 (une erreur générée pour 100 bases séquencées, qui dépasse en général Q50 sur séquence consensus, soit 1 erreur pour 100 000 bases), comparable cependant au taux d’erreur d’un 454 de chez Roche.
La note d’application, plutôt enthousiasmante, est très didactique (notamment pour évoquer les subtilités des différences entre séquençage piared end et mate pair) laisse entrevoir de grandes perspectives… seul bémol, le kit paired end ne serait disponible que dans le premier trimestre de 2012.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages