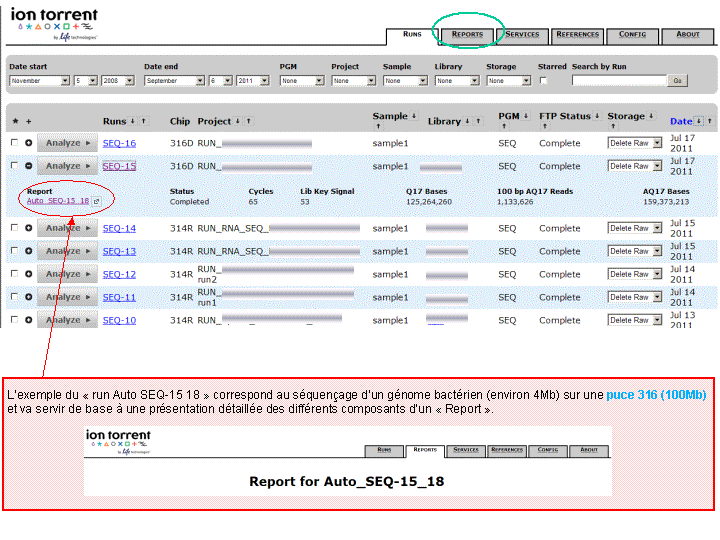
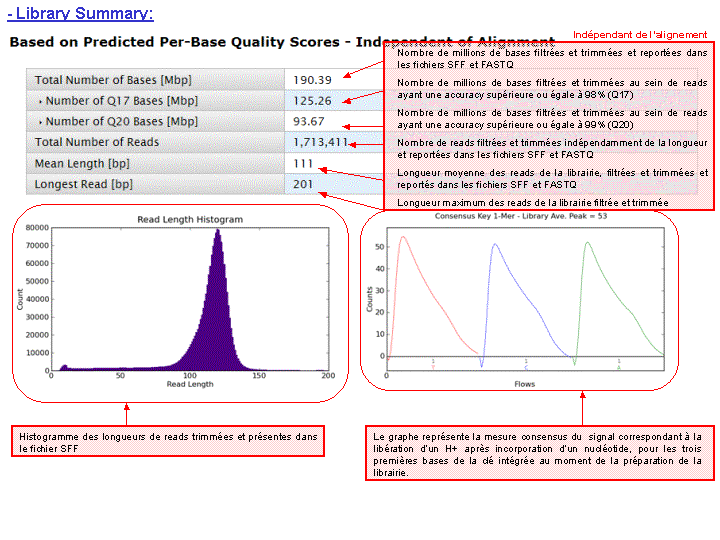
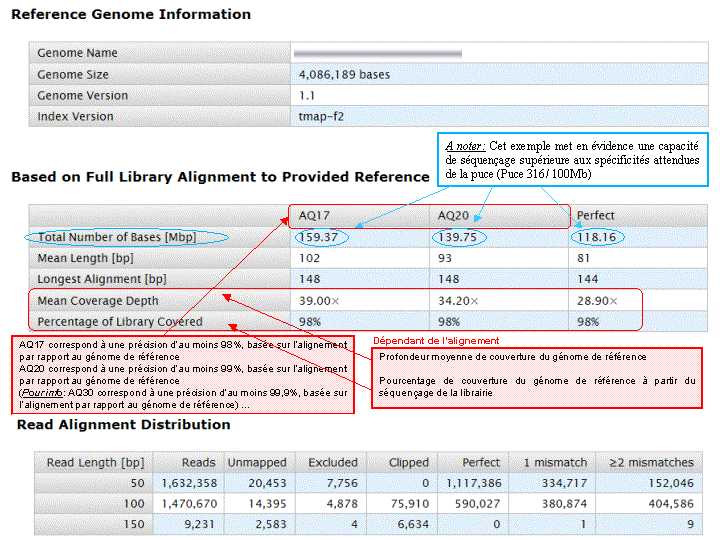
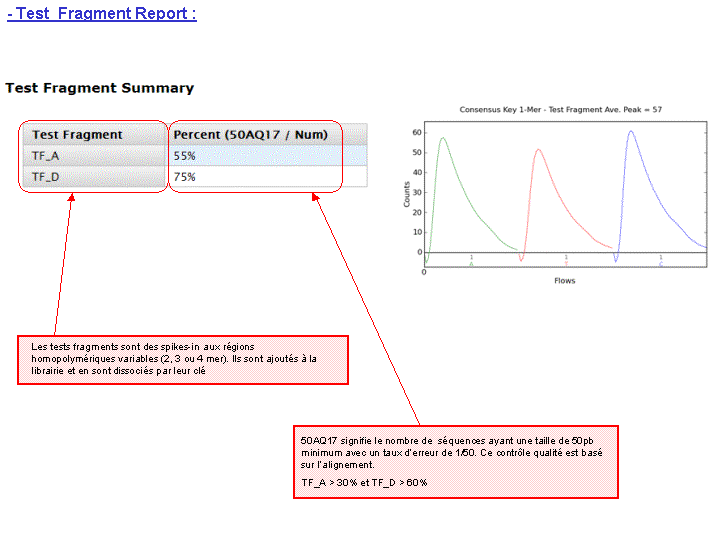
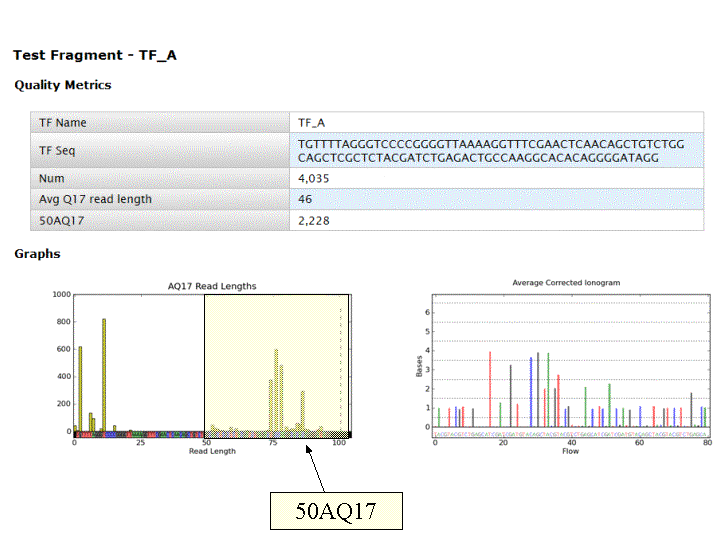
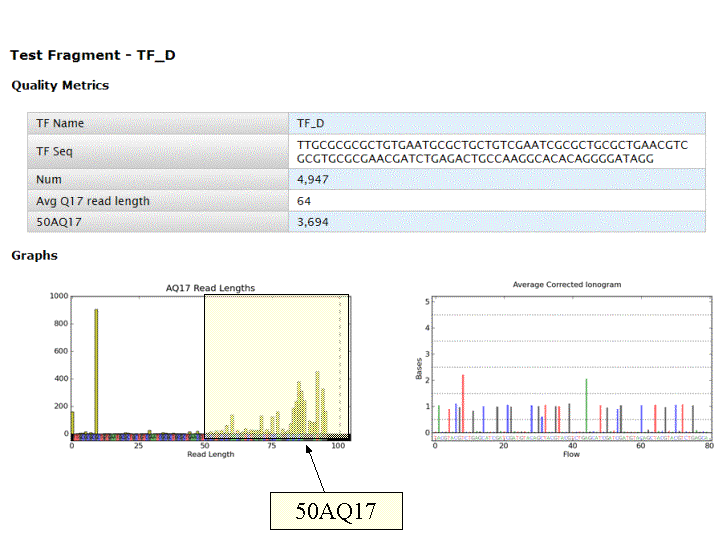
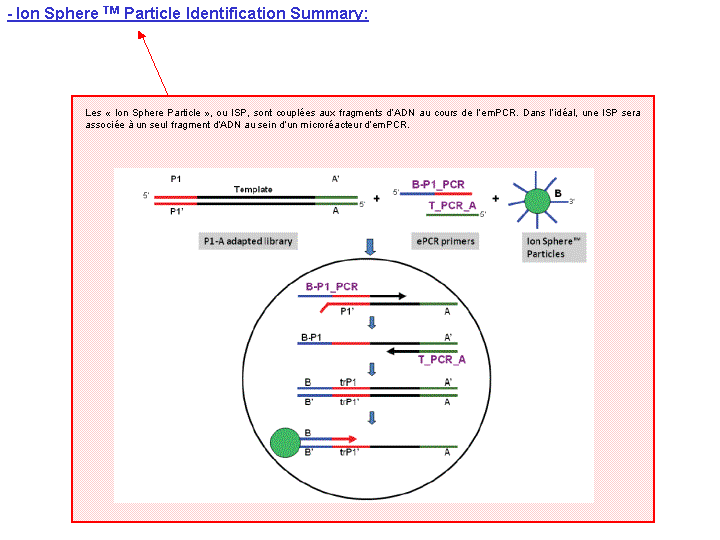
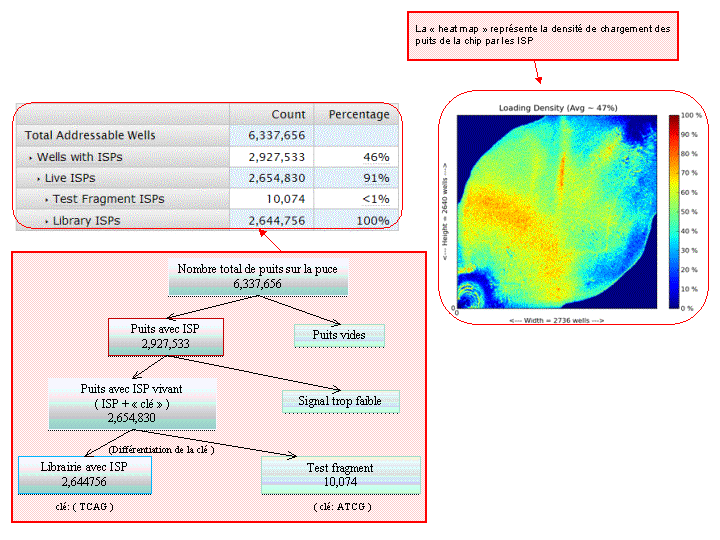
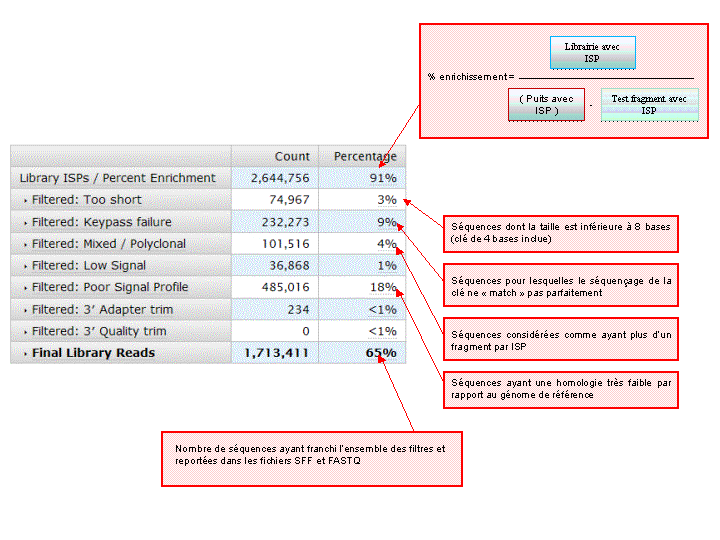
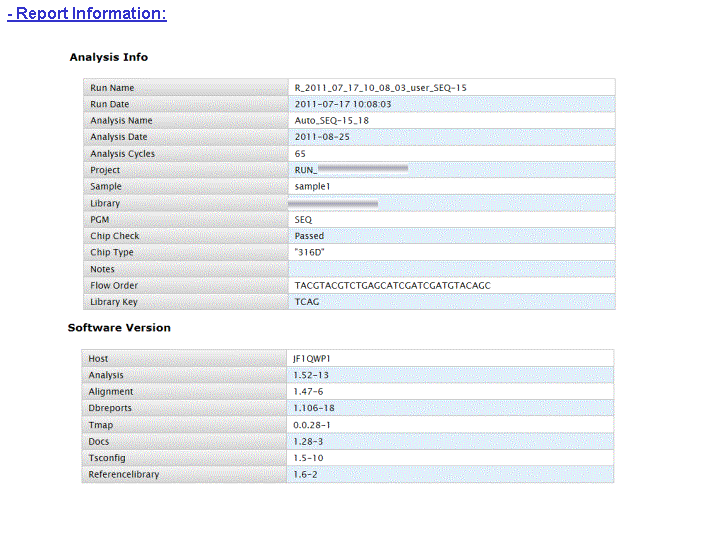
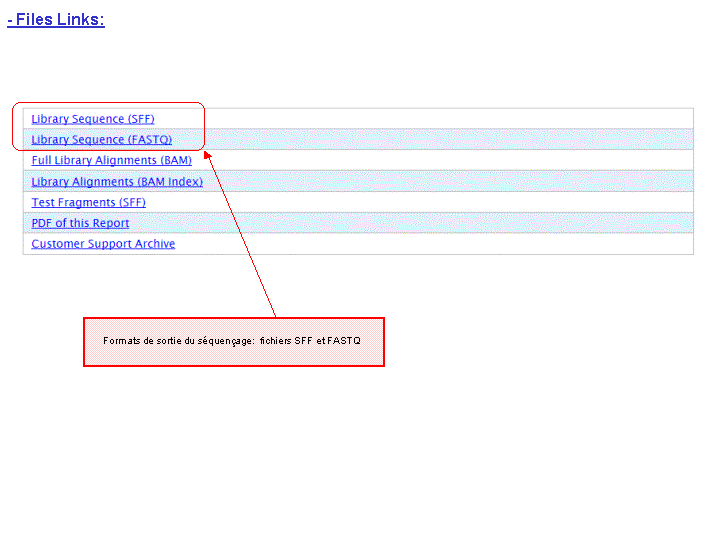
A l’issue d’un « run » de séquençage Ion Torrent (PGM), l’ensemble du signal brut (ionogramme) est converti en séquences et stocké au niveau du serveur. Pour chaque « run de re-séquençage», un alignement préliminaire est réalisé sur la base du génome de référence mentionné lors de l’initialisation du PGM. Cette information est reprise au travers d’un rapport qui comporte également un ensemble de paramètres, que l’on se propose de détailler ci-dessous :
Le rapport se divise en 5 sections:
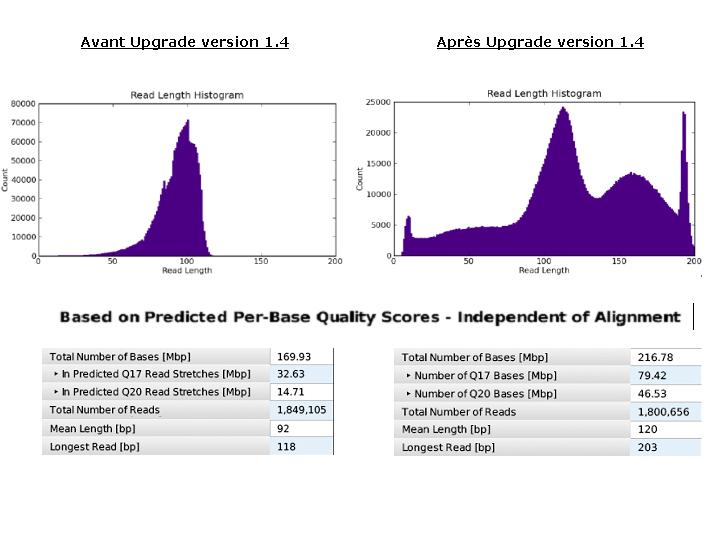
L ’évolution de la technologie Ion torrent permet depuis ces derniers jours, d’accéder au format de puce « 316 » succédant à la « 314 ». Ces puces multiplient par 10 la quantité de données générées à l’issue d’un « run » de séquençage aboutissant ainsi à 100 Mb.
De manière à bénéficier de la capacité totale de ces puces, une mise à jour du serveur, ainsi qu’une configuration du PGM en version 1.4 sont nécessaires et ont été réalisées ce jour. Ces modifications se traduisent par une amélioration de l’algorithme d’analyse (basecalling, alignement, …) optimisant le nombre de reads considérées et traitées. Le développement apporté au niveau des « runs » permet désormais d’intégrer des « reads » de plus grande taille (jusqu’à 200b environ) directement lié à une amélioration de la précision de séquençage.
Techniquement, quelques modifications au niveau du PGM ont conduit au remplacement de certaines tubulures (diamètre supérieur aux précédentes) : Cet épisode fut l’occasion de découvrir les entrailles de la machine…
Notre laboratoire possède un PGM IonTorrent de Life Technologies. Cet outil haut-débit est un séquenceur de paillasse permettant de réaliser des runs à prix relativement accessible, inférieur à 800€/run (cf. notre historique du séquençage proposé dans un post précédent).
Nous avons pu réaliser, en avant-première, un séquençage complet d’une souche d’Escherichia coli sur une puce 316 ,qui permet d’obtenir 100Mb / run, soit l’équivalent d’un peu plus d’une couverture de 20X d’une Escherichia coli.
Pour plus d’informations sur ce séquençage, vous pouvez lire:
– le communiqué de presse associé : CP_Escherichia_coli
-L’article de Futura Sciences sur le sujet : http://www.futura-sciences.com/fr/news/t/genetique-1/d/sequencage-haut-debit-de-coli-vers-une-medecine-personnalisee_31316/
– Des articles plus généralistes sortis dans différents journaux :
Ou regarder ces reportages (datés du 06/07/2011) :
Reportage-LCI-séquençage-coli-06072011
Reportage-BFMTV-séquençage-coli-06072011
Le procédé de séquençage se déroule sur 3 jours :
– préparation de la librairie : 1 jour (fragmentation de l’ADN génomique…)
– préparation de la matrice : 1 jour (PCR en émulsion + enrichissement)
-séquençage de la matrice enrichie : 1/2 jour
Et on obtient le report suivant : Report Run IonTorrent
(*) Sequencing data were generated using system software and protocols bothnon released and non supported by Ion TorrentTM (part of Life TechnologiesTM) and may not reflect actual Ion Torrent PGMTM performance in term of throughput and/or accuracy.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages