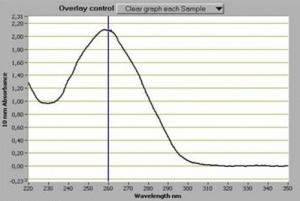
Souvent perçu comme une évidence, le dosage d’acides nucléiques est un point fondamental. Il faut pourtant le considérer comme le premier « monitoring » important d’une expérience de biologie moléculaire. Que vous soyez équipés d’un NanoDrop™, d’un plus antique spectrophotomètre, d’un fluorimètre… les outils sont parfois complémentaires et ont des spécificités propres qu’il vous faudra évidemment connaître (au moins approximativement).
Souvent perçu comme une évidence, le dosage d’acides nucléiques est un point fondamental. Il faut pourtant le considérer comme le premier « monitoring » important d’une expérience de biologie moléculaire. Que vous soyez équipés d’un NanoDrop™, d’un plus antique spectrophotomètre, d’un fluorimètre… les outils sont parfois complémentaires et ont des spécificités propres qu’il vous faudra évidemment connaître (au moins approximativement).
En matière de dosage des acides nucléiques, il apparaît fondamental (si vous utilisez un spectrophotomètre) de connaître la fameuse loi de Beer où A=Ɛ.l.C
A étant l’absorbance (sans unité) et Ɛ est le coefficient d’absorption moléculaire (unité : L.mol -1 .cm-1)
l : la longueur du trajet optique (valeur fixe, donnée par l’épaisseur d’une cuve ou d’un ménisque sur un Nanodrop) (unité : cm)
C, souvent ce que l’on souhaite déterminer: la concentration d’acides nucléiques (unité : mol.L-1)
Cette formule permet d’estimer assez aisément une concentration en acides nucléiques, elle est vérifiée lorsque la solution est de concentration inférieure à : c < 0,1 mol.L-1
| A260 nm | ||
| acides nucléiques | Ɛ (M-1.cm-1) | pour 1 unité absorbance (µg.mL-1) |
| ADN db | 6200 | 50 |
| ADN ss | 8350 | 37 |
| ARN | 7700 | 40 |
| standard : température ambiante, l=1cm, A260/A280 =1,80 | ||
Assez rapidement, la concentration, le plus souvent en ng.µL-1, est déduite. Le point fort de la méthode spectrophotométrique (comparée à la méthode fluorométrique, plus sensible, quant à elle) est fournie par les informations que l’on tire des ratios suivants:
Ratio A260/A280 : un ratio de 1,80 (+/- 0,1) permet de qualifier une extraction d’ADN comme pure, ce ratio se situe autour de 2,0 pour une extraction d’ARN (+/- 0,1). Si ce ratio est inférieur cela signifie que l’extraction est polluée par des protéines et/ou des composés phénoliques
Ratio A260/A230 : la valeur attendue de ce ratio se situe entre 2,0 et 2,20. Si ce ratio est plus faible cela indique nécessairement qu’un composé absorbe à 230 nm. Ces composés peuvent le plus souvent être de l’EDTA, des sucres, et encore une fois le phénol et particulièrement le TRIzol reagent.
Ces valeurs de ratios peuvent parfois être négligées, cependant elles donnent une indication quant à la présence d’éventuels inhibiteurs qui viendraient perturber le devenir de vos acides nucléiques (PCR et autres réactions enzymatiques). Il est important de réaliser une valeur de blanc sur la solution qui vous aura servi à éluer vos acides nucléiques (car comme il a été précédemment mentionné l’EDTA absorbe à 230 nm… et bien souvent le TE 1X sert à reprendre et conserver des acides nucléiques)
vous trouverez ci-contre le mode d’emploi du Nanodrop 1000.
Une autre méthode alternative, permettant une meilleure sensibilité : méthode picogreen. Cette méthode, bien que très sensible, donne des résultats non valides (car très largement sous-estimés) dès lors que votre solution d’acides nucléiques est polluée par des traces d’agents détergents (type SDS). En effet, ces « adjuvants » quenchent la fluorescence émise par l’agent qui s’intercale au sein des acides nucléiques que l’on souhaite doser.
Note : bien souvent il vous est utile de calculer la quantité de molécules (quantité de copies).
Sachant que la masse molaire moyenne d’une base azotée => Masse Molaire (MM) : 309 g.mol-1
&
avec le nombre d’Avogadro 6,02214129.1023 molécules.mol-1
La formule devient : nombre de copies pour 1 µL = [C (concentration mesurée en ng/µL) x V (volume, ici 1 µL) x 10-9 ]/ [(MM environ 309 g.mol-1) x la taille de votre génome en pb x 2] le tout que l’on multiplie par le nombre d’Avogadro… le tour est joué !
plus simplement : 978 Mb pèsent 1 pg (en avant le produit en croix).
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages