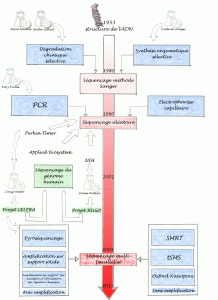
Un bref historique des évolutions du séquençage de l’ADN permet de comprendre ce que certains nomment révolution technologique… certainement concernant l’univers de la biologie moléculaire une innovation comparable à celle la PCR.
Allan Maxam , Walter Gilbert (USA) et Frederick Sanger (Royaume-Uni), les pionniers de la conquête de la séquence, ont mis au point deux méthodes très différentes permettant d’accéder à la lecture de la séquence. Maxam, Gilbert ont exploité des stratégies de dégradation chimique sélective quand Sanger choisit une stratégie de synthèse enzymatique sélective. Pour ces découvertes, Gilbert et Sanger ont été récompensés par le prix Nobel de chimie en 1980. L’histoire ne retient effectivement que Sanger, à juste titre, puisque cette stratégie bénéficiant de l’invention de laPCR (invention de Kary Mullis publiée en 1986, lui aussi nobélisé par le prix de chimie en 1993) et du développement de l’électrophorèse capillaire, permettant de simplifier la partie séparative et analytique. C’est ainsi que des sociétés comme Perkin Elmer, Beckman Coulter et Applied Biosystem ont investi le marché sur le principe même du séquençage Sanger sans le révolutionner mais en permettant son automatisation et l’augmentation du nombre de réactions de séquences analysables par jour. Une fois amorti le coût des séquenceurs, le coût de revient d’une séquence n’a cessé de baisser permettant de banaliser l’accès à la séquence.
Bien que se montrant de plus en plus performant, des applications comme le séquençage complet de génomes eucaryotes supérieurs, les approches métagénomiques (nécessité de cloner), les études de modulation de transcrits (la méthode SAGE les permettait sur la base du séquençage Sanger, cette méthode est lourde et nécessite toujours une phase de clonage) ont connu des limites quasi infranchissables (méthodes nécessitant trop de temps et de capitaux). Prenons à titre d’exemple les projets de séquençage du génome humain. Ces projets auront nécessité plus de 10 ans de travail et de 300 millions de USD pour le projet privé de Celera et un peu moins de 3 milliards de USD pour le projet HUGO (HUman Genome Organisation), le match entre Craig Venter (fondateur de Celera avec le soutien de Perkin Elmer) et James Watson (premier directeur du NIH coordinateur du consortium international) se solde par un match nul entaché de polémiques avec les publications en 2001 des premières séquences du génome humain.
L’année 2005 a connu à grands bruits, l’arrivée de nouvelles méthodes de séquençage à haut débit. Ces méthodes de séquençage massif font appel aux techniques de clonage et d’amplification moléculaire, leur spécificité relevait de leur stratégie de lecture.
En effet, la société 454 (rachetée par Roche en 2007), utilise des méthodes de pyroséquençage (luminescence par libération de pyrophosphate) de fragments d’ADN isolés dans des micro-gouttes comme micro-réacteur de PCR isolés au sein d’une émulsion, la société Solexa (rachetée par Illumina en 2007), utilise des méthodes d’amplification sur support solide permettant l’incorporation de bases terminateurs de chaîne réversibles marqués par des fluorochromes. La société Agencourt (rachetée par Applied Biosystem en 2006), a quant à elle basé son système de détection sur le principe de l’amplification par émulsion et hybridation-ligation chimique.
Depuis 2007, une certaine frénésie s’empare du monde de la biologie moléculaire, un très grand nombre de machines que l’on qualifie tour à tour de NGS (Next Generation Sequencing), HTS (High Throughput Sequencing), ou encore de manière plus appropriée sequençage multi-parallélisé se trouvent disponibles et évoluent vers le plus de profondeur, ou vers le moindre coût, rendant obsolètes les versions précédentes (la société Illumina à titre d’exemple depuis le rachat de Solexa a lancé un modèle de séquenceur tous les ans).
Cette révolution technologique est sortie des laboratoires pour attirer l’attention d’investisseurs et autres banquiers conscients qu’une ruée vers l’or accompagnant la ruée vers la séquence pouvait s’engager. Dans ce sens nous mettons à disposition un document datant de 2007 réalisé pour le compte de la Deutsche Bank (ce document très complet a été co-réalisé par nombre de consultants en biotechnologies, il permet de décortiquer assez finement les 3 technologies présentes sur le marché à partir de 2005 et de montrer les attentes des mondes scientifique et financier)
Afin de terminer ce bref tour d’horizon il semble nécessaire de hiérarchiser ces nouvelles technologies. Il est possible de distinguer tout d’abord deux grands groupes : les technologies sans amplification (permettant de séquencer une seule molécule d’adn matriciel, citons exemple de la technologie SMRT de Pacific Biosciences, la technologie tSMS de Helicos Biosciences ou encore celle toujours en développement mais très attendue développée par Oxford Nanopore) et avec amplification (toutes les technologies amplifiant une matrice clonale d’adn issu d’une librairie)
Parmi cette seconde classe, il est possible de distinguer trois grandes classes de matériel :
– les usines à produire de la séquence (type Hiseq 2000 de Illumina permettant de séquencer jusqu’à 200 Gb par run de 8 jours
– les séquenceur de paillasse (type PGM de IonTorrent de taille et prix beaucoup plus modeste permettant de réaliser des run autour de 100 Mb avec une perspective de 1 Gb pour début 2012)
– les séquenceurs haut débit en voie d’obsolescence (454 de Roche, les versions des séquenceurs Illumina antérieures au HiSeq 1000 etc.)
Cet historique est bien entendu un prétexte pour planter le décor et présenter les forces en présence. Après l’âge des pionniers (Maxam et Sanger), celui de la douce exploitation (séquenceur capillaire) vient celui de l’accélération que certains nomment révolution… ces technologies marquent quoi qu’il en soit un tournant pour la génomique et pour les applications actuelles et en devenir qui y sont liées. Enfin la diffusion de ces technologies va de paire avec un changement de physionomie des laboratoires : la biotechnologie est une science composite où bio-informatique, biologie cellulaire et moléculaire mais aussi physique et statistique deviennent de plus en plus interdépendantes.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages