Société Française de Microbiologie, le Vendredi 17 Mai 2013, vous propose un colloque ayant pour thème central : les nouvelles méthodes de détection des micro -‐organismes : PCR/ESI-‐TOF MS, séquençage de nouvelle génération.
Société Française de Microbiologie, le Vendredi 17 Mai 2013, vous propose un colloque ayant pour thème central : les nouvelles méthodes de détection des micro -‐organismes : PCR/ESI-‐TOF MS, séquençage de nouvelle génération.
ASIEM, 6 Rue Albert de Lapparent, 75007 Paris (Métro Ségur, ligne 10)
L’intégralité du programme est disponible (ici, au format pdf) : les représentants des 3 fournisseurs de séquenceurs de paillasse de 2ème génération (Roche, Life et Illumina) y seront présents avec des interventions regroupées en deuxième partie de matinée. Outre le séquençage haut-débit, ce colloque est l’occasion d’entendre parler de PCRs couplées à de la spectrométrie de masse comme outil de détection moléculaire.
Des informations complémentaires sont disponibles sur le site de la SFM.
 Le séquençage haut-débit utilisant des analogues de nucléotides « portant des groupes bloquants en 3′ qui sont utilisés pour produire une terminaison de chaîne réversible appliqués pour le séquençage par synthèse nucléotidique » (je sais cette phrase est « un peu » lourde…) est un brevet de Caliper Technologies -autrement dit de Perkin Elmer : la firme qui n’a pas su saisir sa chance lors de la révolution du séquençage haut-débit. Le séquençage par synthèse (avec réversion du blocage des nucléotides fluorescents polymérisés -un peu moins lourde cette phrase là…) est le cœur de la technologie faisant une partie du chiffre d’affaire d’Illumina (l’intégralité de sa gamme de séquenceurs fonctionne sur ce principe).
Le séquençage haut-débit utilisant des analogues de nucléotides « portant des groupes bloquants en 3′ qui sont utilisés pour produire une terminaison de chaîne réversible appliqués pour le séquençage par synthèse nucléotidique » (je sais cette phrase est « un peu » lourde…) est un brevet de Caliper Technologies -autrement dit de Perkin Elmer : la firme qui n’a pas su saisir sa chance lors de la révolution du séquençage haut-débit. Le séquençage par synthèse (avec réversion du blocage des nucléotides fluorescents polymérisés -un peu moins lourde cette phrase là…) est le cœur de la technologie faisant une partie du chiffre d’affaire d’Illumina (l’intégralité de sa gamme de séquenceurs fonctionne sur ce principe).
Le principe assez simple du séquençage SBS (Sequencing By Synthesis) est en train de se décliner au sein de machines haut-débit plus accessibles en terme de coût… il s’agit là de l’arrivée sur le marché d’une nouvelle concurrence : des séquenceurs génériques tels que ceux que proposent le distributeur Azco Biotec, Inc. Ces séquenceurs viennent directement concurrencer ceux d’Illumina accompagnés de leurs réactifs « génériques » tels que ceux distribués par la société Proteigene…
Ces séquenceurs « génériques » ne révolutionnent rien, leur part de marché est actuellement marginale… les laboratoires académiques préfèrent souvent l’original à la copie, malgré tout la concrétisation de la « promesse » –qui l’a formulée au juste ?– d’un génome humain pour 100 $ d’ici 2015 passe par la large diffusion de machines dont la technologie a fait ses preuves… Le retard pris par les séquenceurs de 3ème génération laisse le champ libre à la 2ème. Le marché risque de se saturer en séquenceurs de paillasse (séquenceur de 2ème génération tels que le MiSeq d’Illumina et les PGM et Ion Proton de Life Technologies).
Face aux arguments commerciaux développés en faveur de tels ou tels séquenceurs de paillasse à haut débit, l’étude menée par Loman et al., Nature Biotechnology propose une comparaison des performances du 454 GS Junior (Roche), du MiSeq (Illumina) et du PGM Ion torrent (Life Technologies), à partir du séquençage de novo d’un isolat d’E.coli O104 :H4 responsable de l’intoxication alimentaire en Allemagne (Mai 2011). Pour cela, le génome de référence a été généré à l’aide du GS FLX (Roche) et permettra d’évaluer l’efficacité des séquenceurs et algorithmes d’assemblages associés.
Sur la base des caractéristiques techniques établies en 2011, les trois plateformes ont permis de générer un draft exploitable du génome bactérien sans pour autant parvenir à le reconstituer à 100%. Par conséquent, il est impossible de désigner un séquenceur comme étant celui de choix, chaque plateforme étant un compromis entre avantages et inconvénients.
Deux « runs » de 454 GS Junior (associés au bénéfice de reads de 600 bases) ont permis d’atteindre 96,28% de couverture par rapport au génome de référence (Contre 96,05% pour le MiSeq et 95,4% pour le PGM Ion torrent).
Malgré une capacité de séquençage du PGM Ion torrent supérieure à celle du 454 GS Junior (100Mb contre 70Mb) et une précision de séquençage voisine (Q20, soit 1 erreur toutes les 100 bases), la technologie reposant sur les semiconducteurs fait face à ses limites au niveau de la gestion des homopolymères et accessoirement d’une longueur de reads de 100 bases.
Vingt fois supérieur au 454 GS Junior en terme de quantité de bases décodées, un « run » de MiSeq ne parvient qu’à talonner les performances d’un double « run » de la technologie Roche. Bien qu’étant la seule à gérer les homopolymères et la seule doter d’une précision à Q30 (soit 1 erreur toutes les 1000 bases), la technologie Illumina faillit sur ses tailles de « reads » qui ne permettent pas une gestion optimum des séquences répétées lors de l’assemblage.
L’approche analytique est également évoquée et selon le système d’assemblage utilisé (Velvet, CLC, MIRA), de nombreuses différences sont observées.
Enfin, ce document est l’occasion de rassembler des notions assez pragmatiques comme le tarif des plateformes (et annexes associées), la durée d’un « run » de séquençage ou encore la quantité de données générées.
Ceci étant, on est en droit d’émettre quelques réticences quant à la pertinence de cette étude qui base son comparatif sur une application (séquençage de novo) qui n’est pas la vocation première de cette catégorie de séquenceurs. Par ailleurs, dans le laps de temps où cette étude a été réalisée et la parution des résultats, la technologique du PGM Ion torrent a évolué. Testé sur les bases d’une puce 316 (100Mb) et d’une technologie de 100 bases en longueur de « reads », le PGM bénéficie désormais de la puce 318 (1 Gb) associée à une technologie de reads de 200 bases. Sans tarder, Life technologies réagit (“Loman et al reflects the past, not the present”) et propose une mise à jour au travers de ce document.
En pleine guerre froide des compagnies de biotechnologies, développeuses et vendeuses de matériel de séquençage… une fuite de documents est organisée. Le tout pour fournir aux personnes prescriptrices ou décideuses d’un futur achat de séquenceur, les arguments pour orienter ou justifier leur choix… parfois même leur mauvaise foi.

En cliquant sur la volute nucléique (depuis le 30/08/2012 – ce lien est censuré par Illumina- cf. commentaires de ce même article) ci-dessus vous aurez accès à un vrai-faux document confidentiel fourni par Illumina. Ce document assez bien réalisé a devancé celui de Life Technologies présenté dans notre article précédent. Il met l’accent sur le bénéfice, au niveau de la qualité des données générées, que procure un MiSeq (Illumina) par rapport à un PGM (Ion Torrent, Life Technologies)
Un document paru dans la Ion Community (site de partage autour de la technologie Ion Torrent et Ion Proton de Life Technologies) commence à faire parler de lui. En effet, des scientifiques de Life Technologies montrent et démontrent que le MiSeq d’Illumina produit (aussi) des erreurs au niveau des homopolymères.
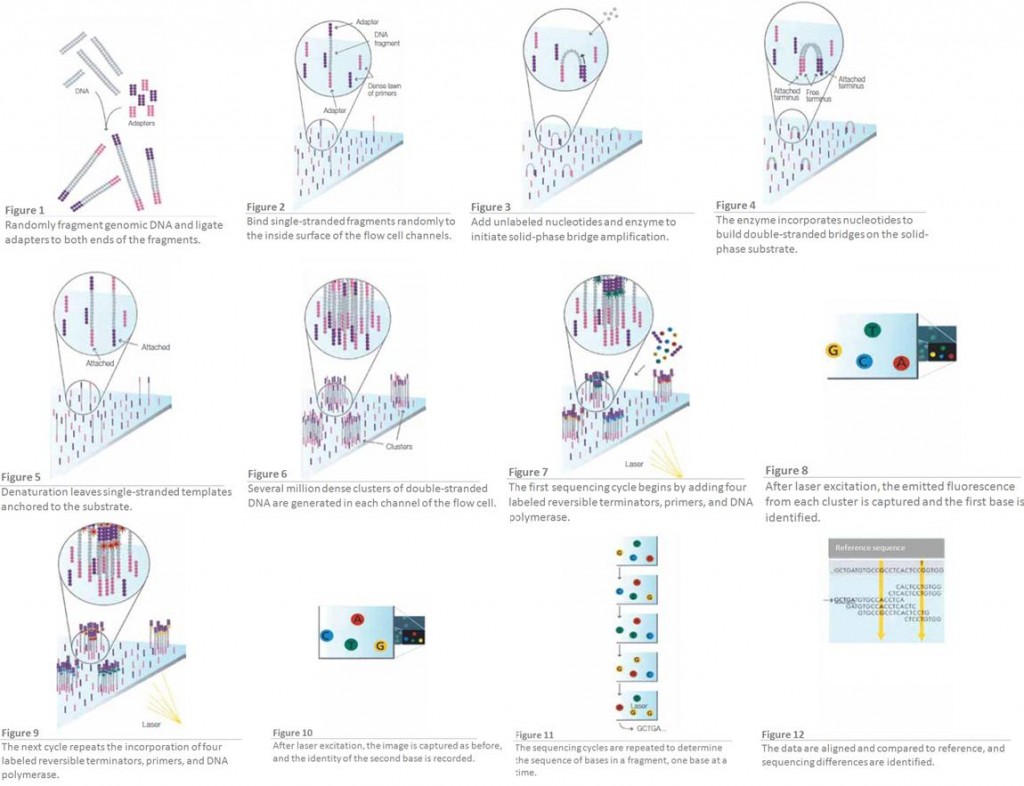
Nous savions depuis longtemps que les séquenceurs de type 454 ou Ion Torrent montraient des biais quant à détermination correcte de ce type de séquences… Ici, pour la première fois les méthodes de SBS (Sequencing By Syntesis) dont le principe pour la technologie Illumina est rappelée par le schéma disponible ci-dessous, montrent des biais étonnement importants pour la juste détermination de séquences adjacentes d’homopolymères (particulièrement concernant les régions GC % riches). La note qui met le feu dans l’argumentaire commercial d’Illumina est disponible ici-même. Une hypothèse quant à la génération de ce type d’erreurs est formulée.
Nous vous laissons le soin de lire la note, intéressante malgré la partialité de la source. La démonstration quoique scientifique, montre l’âpreté de la bataille qui se joue entre les acteurs majeurs du séquençage de paillasse de deuxième génération.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages