Ce poste a vocation de passer en revue les différentes étapes de la technologie de séquençage à haut-débit du PGM Ion Torrent, depuis la préparation de la librairie jusqu’à l’obtention des données brutes. Ce survol technologique permet de rassembler un maximum d’explications techniques et de termes clefs associés à cette technologie. L’intérêt est de répondre essentiellement à une attente de néophytes ou futurs utilisateurs du PGM friands de retours d’expériences.
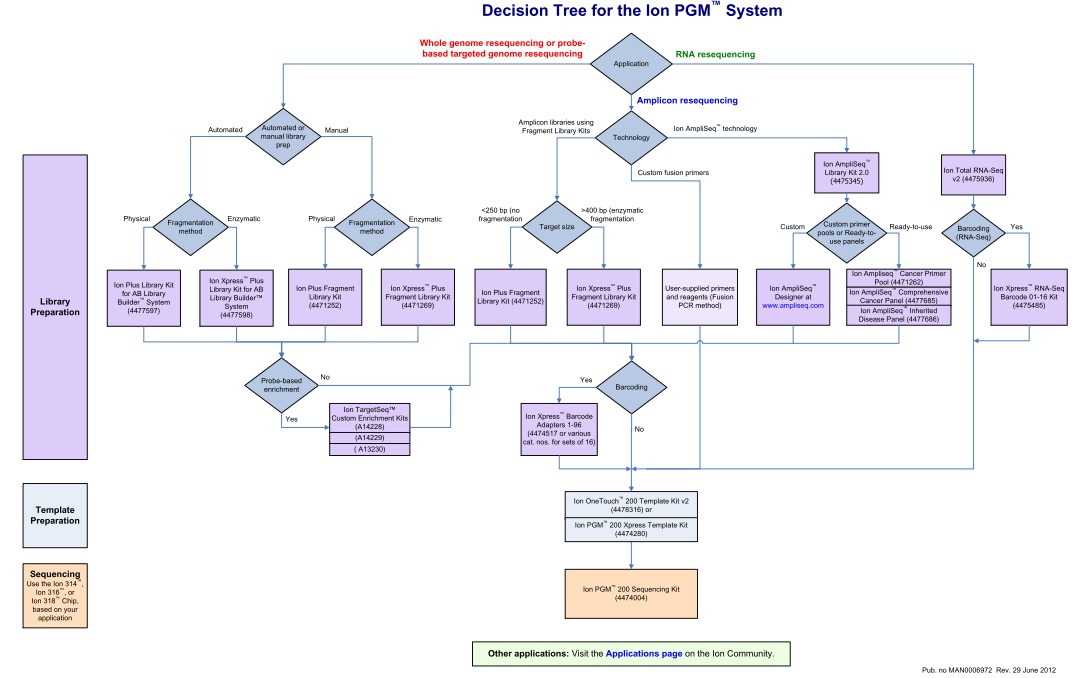
L’arbre de décision Ion Torrent s’étant considérablement développé, seuls les axes « Whole genome » et « Amplicon » serviront de support à l’ensemble de la présentation.
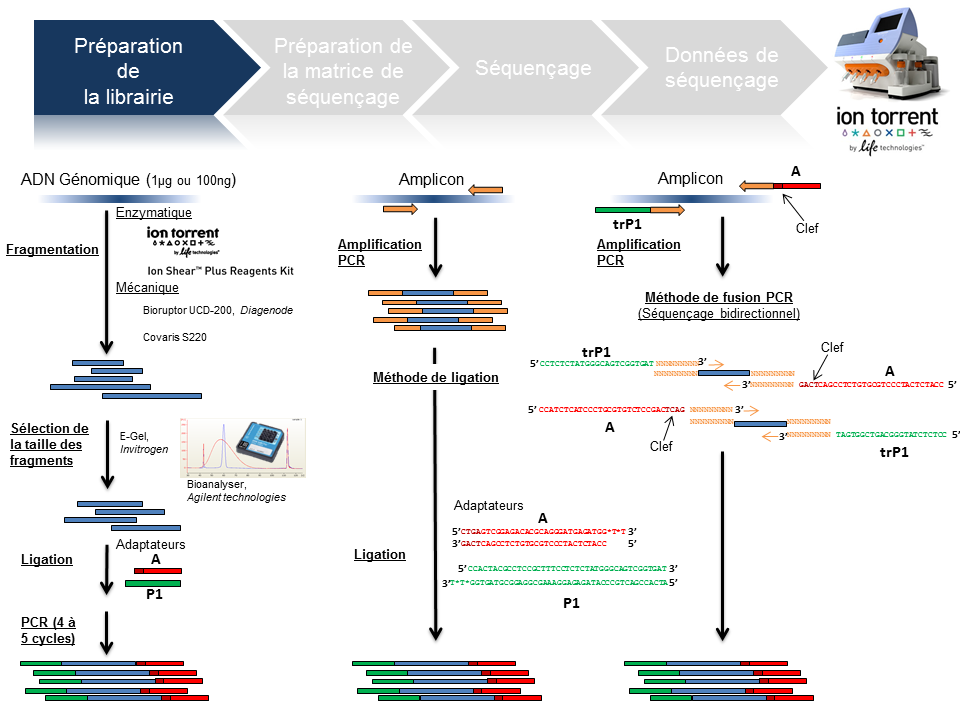
Préparation de la librairie
La finalité d’une préparation de librairie pour le PGM Ion Torrent est de lier aux fragments d’ADN à séquencer le couple d’adaptateurs A et P1. La taille médiane des fragments est variable et définie selon la chimie employée: 100, 200 , 300 ou 400 bases (Cf tableau ci-dessous).
Le traitement d’un échantillon d’ADN génomique débute par une étape de fragmentation mécanique ou enzymatique. Cette dernière présente l’avantage d’être considérablement plus rapide.
En amplicon-seq, la méthode pour flanquer les adaptateurs est double, par ligation ou par fusion PCR.
Par ailleurs, il est envisageable de traiter plusieurs échantillons en parallèle en utilisant des adaptateurs avec code barre ( En standard chez Life technologies: Au nombre de 96 pour les échantillons ADN et 16 pour les échantillons ARN).
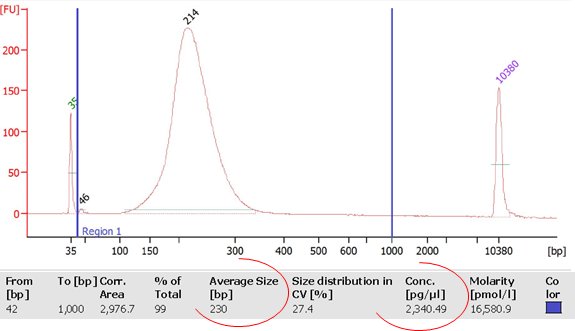
La librairie est monitorée sur puce DNA High sensitivity (Bioanalyzer, Agilent) avec comme objectifs de calculer sa concentration et d’identifier la taille moyenne des fragments qui la composent. Ces valeurs permettront ainsi de déterminer la concentration molaire de la libairie et d’y appliquer le facteur de dilution nécessaire pour favoriser le ratio idéal 1/1 (Fragment ADN de la librairie/Ion Sphere Particle) pour l’étape suivante de PCR en émulsion.
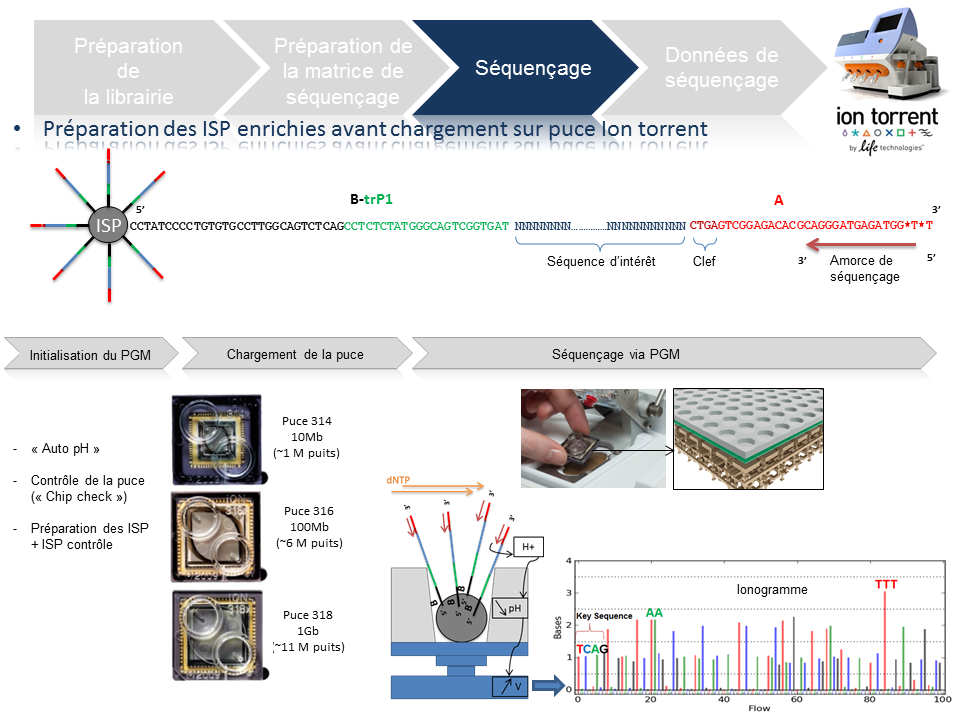
Préparation de la matrice de séquençage
Cette étape automatisée permet l’amplification clonale (OneTouch2) suivi d’un enrichissement en « Ion Sphere Particles » (ISPs) à la surface desquelles un fragment de librairie est amplifié (OneTouch ES).
L’amplification clonale est réalisée au cours de la PCR en émulsion (emPCR) et contribuera à atteindre un seuil de détection du signal nécessaire et suffisant au moment du séquençage. Malgré une optimisation du ratio 1/1 (ISP / Fragment ADN), plusieurs configurations de microréacteurs sont envisageables. Seule la configuration de monoclonalité est souhaitée car elle seule, est source de données de séquençage. Les autres configurations généreront des données qui seront filtrées lors de la primo-analyse par la « Torrent suite ».
L’amorce ePCR-A couplée à la biotine permettra l’enrichissement ultérieure par un système de capture sur billes liées à la streptavidine.
Séquençage
En amont de l’étape de séquençage, une initialisation du PGM est requise et permet notamment une homogénéisation des valeurs de pH ~ 7,8 au sein des différents réactifs de l’appareil (« Auto pH« ).
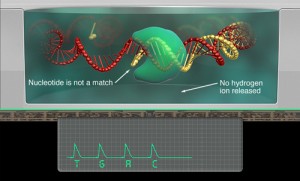
La matrice de séquençage couplée aux amorces de séquençage et à la polymérase est chargée sur la puce Ion Torrent selon un protocole bien spécifique. Les puces se déclinent selon 3 capacités de séquençage ( Chip 314 >10Mb, Chip 316 >100Mb, Chip 318 >1Gb). A noter qu’une version « v2 » pour chacune des puces précitées existe et est indispensable pour toute application de séquençage nécessitant la chimie 400. Le séquençage multiparallélisé revient donc au décryptage simultané des fragments ADN couplés aux ISP. A chaque polymérisation de nucléotides non modifiés, la libération d’ ions H+ entraine une variation de pH, elle même détectée au niveau de la couche mince (technologie des semi-conducteurs) située au fond de chaque puits. L’ensemble des données brutes générées est transcrits sous forme de ionogrammes.
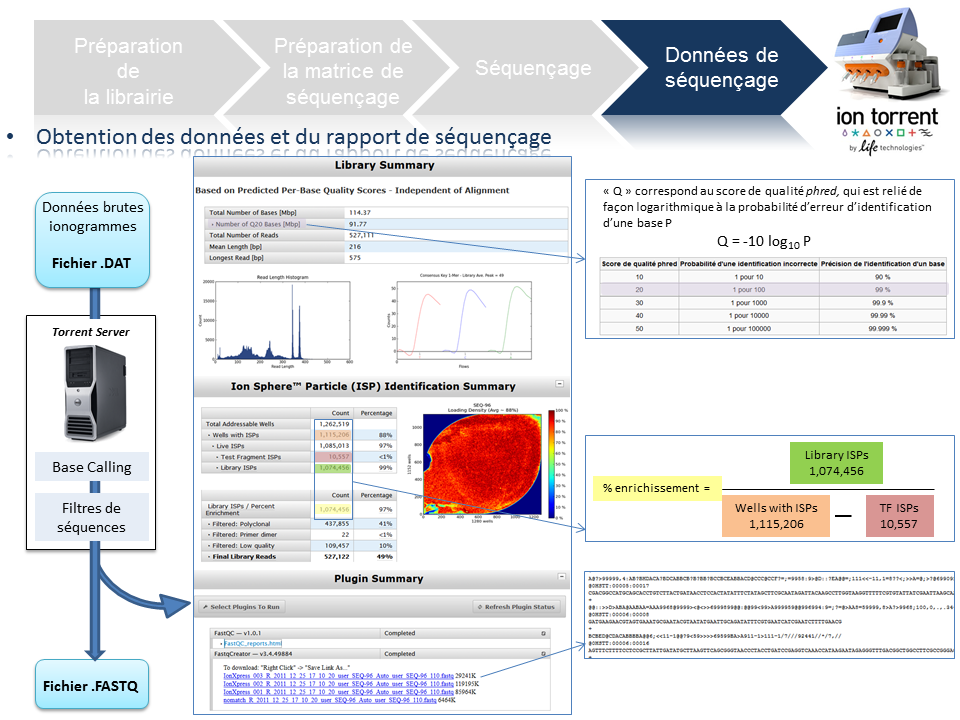
Données de séquençage
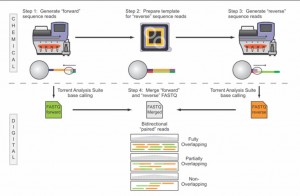
A l’issue du « run » de séquençage, le fichier .DAT regroupe l’ensemble des données brutes (ionogrammes). Ces fichiers sont transférés du PGM vers le Torrent Server. L’algorithme de « base calling » permet la conversion des données sous forme de lettres en séquences (A,T,C,G) formant le read (séquence au format fasta) associé à un score de qualité (Phred Score codé en ASCII), les deux types de données étant associés dans un fichier .FASTQ (qui tend à devenir le format de référence).
Un prétraitement est également appliqué sur la base des reads générés et qui équivaut au nombre d’ISPs vivantes ou « Live ISPs » (On parle d’ISPs vivantes pour les ISPs associées à la clef):
– trimming : élimination des adaptateurs et/ou portions de reads de mauvaise qualité
– filtres : élimination des « reads » de petites tailles, de mauvaise qualité, des polyclonaux
L’ensemble de ces informations est repris au travers du « report » généré à l’issue du séquençage et du prétraitement. Y sont également renseignés, le nombre de reads générés ainsi que leur taille moyenne.

L’Ion Torrent™ Mini Lab sera à Lille
le 20 Juin 2013
14h00-16h00
Parking du Campus Institut Pasteur de Lille
1 rue du Pr. Calmette
59000 Lille
Métro Ligne 2, Station Grand Palais
Flyer : une mini, un Ion Torrent dedans…
 Le mois de décembre 2012 a connu le lancement du kit 400 bases (
Le mois de décembre 2012 a connu le lancement du kit 400 bases (
Ion PGM™ Template OT2 400 Kit) qui est principalement attendu pour les applications de séquençage de novo et de séquençage d’amplicons ciblant le 16S, 18S ou encore ITS2 (respectivement pour l’étude de la diversité bactérienne, eucaryote ou fongique) pour les études métagénomiques. Comme vous pouvez le constater cette application est uniquement supportée par le One Touch 2 devenu nécessaire pour accéder aux reads de 400 bases (c’est en partie pour cette raison que notre plateforme PEGASE-Biosciences a décidé de se doter de cette automate).
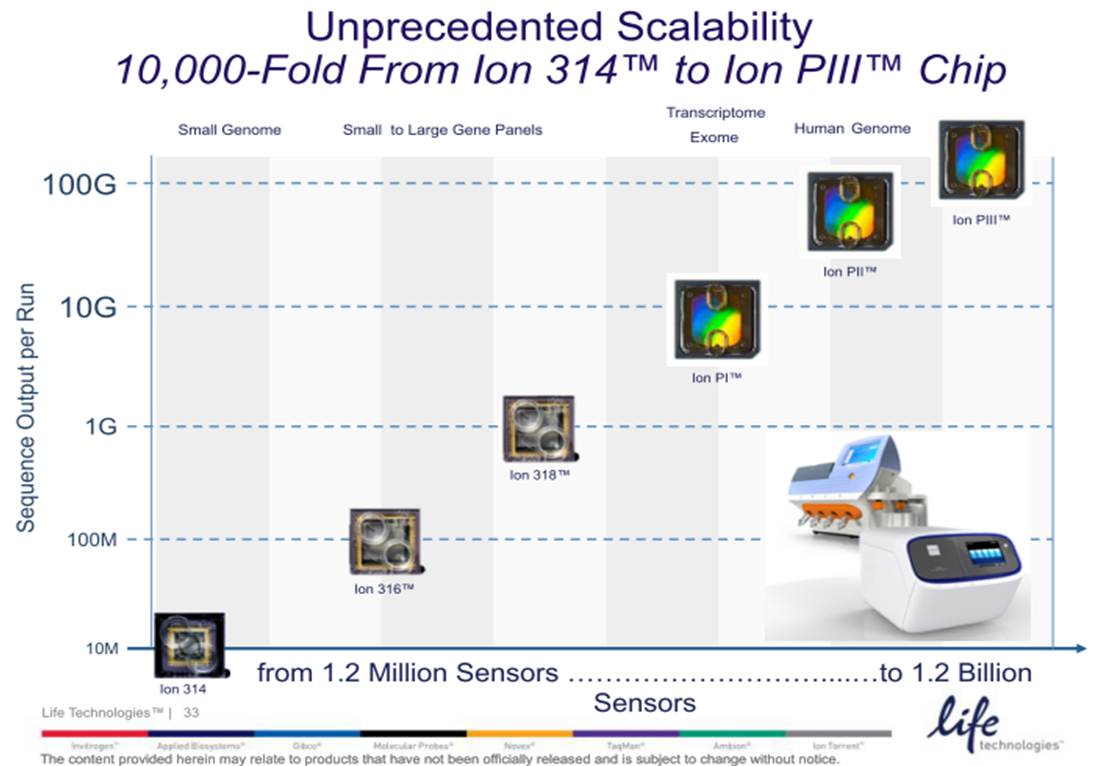
L’autre nouveauté est portée par les consommables de séquençage associés au Ion Proton (le grand frère du PGM). En effet, si la Puce Ion PIII est encore virtuelle, la PI et PII, permettent de délivrer : 10 Gb et 70 millions de reads filtrés environ pour la première, contre plus de 280 de millions de reads et plus de 70 Gb pour la seconde (de quoi séquencer un génome humain à 20 X).
Du côté de Life Technologies et de sa technologie Ion Torrent, sans aucun doute, la principale nouveauté de l’année 2013 touche l’amplification clonale (phase où, à partir de la librairie préparée, est réalisée la matrice de séquençage fixée de manière covalente sur des billes qui se logeront dans les puits présents sur la puce). Ainsi, la fameuse (et pour certains la fâcheuse) phase de PCR en émulsion trouvera une alternative avec la chimie isothermique dite « avalanche » (dont vous pouvez voir l’aperçu d’un run ci-dessous). A en croire Life Technologies, cette chimie ouvre la voie à des tailles de séquences générées encore plus longues.
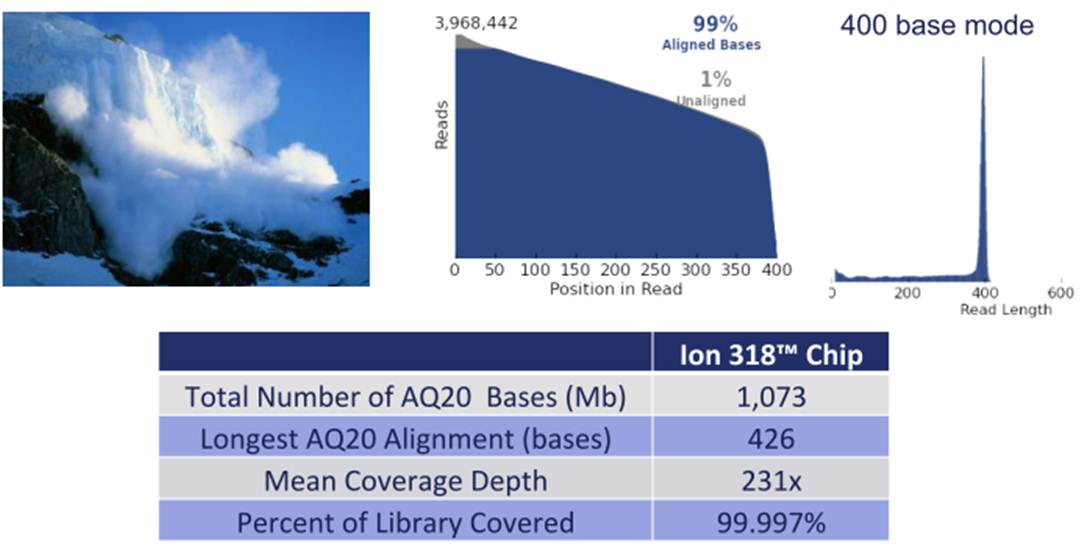
Bien entendu, Life Tech annonce l’arrivée d’un nouvel automate permettant d’intégrer la nouvelle chimie mais aussi de charger les puces (et pour quelques dizaines de k€, d’épargner au manipulateur les deux phases délicates du séquençage).
Pour conclure brièvement, l’une des nouveautés et non des moindres : l’acceptation du PGM comme diagnostic moléculaire CE-IVD (un parcours pas vraiment de tout repos) a n’en pas douter une conformité qui ne devrait pas laisser insensible les chercheurs, praticiens hospitaliers.
Jeudi 13 Septembre 2012 de 9h00 à 12h30 dans l’amphi Buttiaux de l’Institut Pasteur de Lille
Après plus d’un an d’utilisation du séquenceur Ion PGM ™ , la plateforme PEGASE-Biosciences organise une demi-journée de retour d’expérience afin de dresser un bilan des possibilités offertes par cette technologie et d’échanger sur les applications du séquençage à haut-débit.
Vous pouvez télécharger le flyer en suivant ce lien.
Agenda :
9h00 – 9h10 Mot d’accueil – Introduction.
9h10 – 9h30 Stéphane Jankowski (Life Technologies) : Technologie Ion Torrent ™
9h30 – 10h00 Valérie Leclère (Laboratoire ProBioGEM, Univ Lille1) : « Séquençage de génomes entiers pour caractériser de nouvelles souches de Bacillus ».
10h00 – 10h30 Magali Chabé (BDPEE, CIIL – Institut Pasteur de Lille) : « Etude du transcriptome de deux espèces de Pneumocystis par RNA-sequencing de novo ».
10h30 – 10h50 Pause café.
10h50 – 11h20 Christophe Audebert (Gènes Diffusion) & Ségolène Caboche (IFR142, Lille2) : « Le séquençage haut-débit appliqué à la caractérisation d’un agent infectieux : exemple du séquençage d’Escherichia coli à l’origine d’une infection alimentaire ».
11h20 – 11h50 Charles Hébert (Pathoquest) : « Détection et identification de micro-organismes dans le plasma humain par approche métagénomique ».
11h50 – 12h20 David Hot (TAG, CIIL – Institut Pasteur de Lille) : « Identification de nouveaux transcrits (small RNAs) et étude des départs de transcription chez Bordetella pertussis par ‘differential RNA-seq’ ».
12h20 – 12h30 Conclusion – modalités du concours PEGASE.
Pour célébrer la création de “PEGASE-Biosciences” entre le laboratoire de Transcriptomique et Génomique Appliquée de l’IPL et la société Gènes Diffusion, la plateforme organise un concours* vous permettant de gagner un séquençage Ion Torrent ™ sur puce 318 (~1Gb de séquences).
Les places étant limitées, nous vous remercions de vous inscrire par email à l’adresse suivante :
stephane.jankowski@lifetech.com ou admin@pegase-biosciences.com
*Les modalités de ce concours vous seront présentées lors de cette journée.
Lien vers l’annonce du séminaire sur : http://www.pegase-biosciences.com/?p=464
Vous pouvez confirmer votre participation en scannant le QR Code suivant :
Un document qui n’est pas sans rappeler celui publié en novembre 2011, en intégrant les dernières mises à jour.
Cet arbre de décision est l’occasion de retrouver l’ensemble des modalités de séquençage qu’offre la technologie Ion Torrent via le PGM. En attendant l’arrivée du kit permettant de séquencer 400b, l’essentiel des modifications résident dans la préparation des échantillons avec notamment de nouvelles indexations (évolution du kit à 96 barcodes en DNA-seq et apparition du kit à 16 barcodes en RNA-seq), un élargissement du panel de primers dédiés à la recherche sur le cancer, etc…
Pour les privilégiées de la Ion Community, ce document est également l’occasion de retrouver aisément la liste des protocoles Ion Torrent selon l’ application d’intérêt en cliquant sur le lien en bas de page.
En pleine guerre froide des compagnies de biotechnologies, développeuses et vendeuses de matériel de séquençage… une fuite de documents est organisée. Le tout pour fournir aux personnes prescriptrices ou décideuses d’un futur achat de séquenceur, les arguments pour orienter ou justifier leur choix… parfois même leur mauvaise foi.

En cliquant sur la volute nucléique (depuis le 30/08/2012 – ce lien est censuré par Illumina- cf. commentaires de ce même article) ci-dessus vous aurez accès à un vrai-faux document confidentiel fourni par Illumina. Ce document assez bien réalisé a devancé celui de Life Technologies présenté dans notre article précédent. Il met l’accent sur le bénéfice, au niveau de la qualité des données générées, que procure un MiSeq (Illumina) par rapport à un PGM (Ion Torrent, Life Technologies)
Dernier né de la gamme des séquenceurs de paillasse, le « Ion Proton Sequencer » (149.000 $) est le premier de sa catégorie à permettre le séquençage en quelques heures du génome humain et à faible coût (1.000 $). A l’instar du PGM Ion Torrent, cette nouvelle plateforme est basée sur la technologie des semi-conducteurs (et la détection de la libération d’ions H+ suite à la polymérisation de dNTP).
La puce « Ion Proton ™ I »,disponible mi-2012, aura une capacité de séquençage 1000 fois supérieure au premier format de puce 314 du PGM Ion torrent soit 10 Gb (davantage orientée pour le séquençage d’exome humain). Le second format (puce « Ion Proton ™ II ») , disponible environ six mois plus tard aura une capacité de 100 Gb (Séquençage Génome humain).
Compte tenu de l’apparente similarité entre les deux plateformes de chez Life technologies, l’évolution réside essentiellement dans l’architecture de la puce avec une augmentation du nombre de puits ( 165 millions de puits pour la puce « Ion Proton ™ I » et 660 millions pour la puce « Ion Proton ™ II » soit respectivement 100 et 1000 fois plus que la puce 314 du PGM ), l’une des bases de la capacité de séquençage (en complément de la longueur des reads générés).
Voici pour continuer sur les développements qui ont lieu sur la plateforme de séquençage de Ion Torrent, le PGM, une nouvelle note d’application qui ouvre de nouvelles perspectives dans le champ du séquençage haut-débit : le fait de pouvoir très bientôt séquencer des fragments de 400 bases dans les deux sens. Ainsi, le PGM de Ion Torrent sera la seule technologie capable de fournir sur des reads plutôt longs un séquençage paired end (sachant que le kit et protocole associé « mate paire » est d’ores et déjà disponible). Evidemment, ce développement vient contrecarrer une critique qui peut être portée sur cette plateforme de séquençage : sa « justesse » de séquençage sur séquences brutes générées à Q20 (une erreur générée pour 100 bases séquencées, qui dépasse en général Q50 sur séquence consensus, soit 1 erreur pour 100 000 bases), comparable cependant au taux d’erreur d’un 454 de chez Roche.
La note d’application, plutôt enthousiasmante, est très didactique (notamment pour évoquer les subtilités des différences entre séquençage piared end et mate pair) laisse entrevoir de grandes perspectives… seul bémol, le kit paired end ne serait disponible que dans le premier trimestre de 2012.
Le bien beau document que voilà ! Se repérer dans toutes les options de préparation des échantillons à séquencer, options qui sont fonctions de l’origine et de l’application que l’on souhaite mettre en oeuvre par l’intermédiaire du séquençage, peut être un réel casse-tête que ce document souhaite simplifier ici.
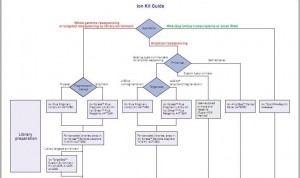 Cet arbre de décision s’étoffera très rapidement : d’autres branches pousseront avec les protocoles « mate pair » & « paired end », les nouvelles indexations… et surtout l’arrivée du kit permettant de séquencer 400 b… Peut être que les branches portant les options 100 b seront, quant à elles, pour l’occasion élaguées.
Cet arbre de décision s’étoffera très rapidement : d’autres branches pousseront avec les protocoles « mate pair » & « paired end », les nouvelles indexations… et surtout l’arrivée du kit permettant de séquencer 400 b… Peut être que les branches portant les options 100 b seront, quant à elles, pour l’occasion élaguées.
Ce document, et beaucoup d’autres, est disponible sur le site de la communauté Ion Torrent.
Pour donner suite aux demandes assez nombreuses de détails concernant le principe de fonctionnement du séquençage multiparallélisé PGM (Ion Torrent / Life Technologies).
Sur le lien suivant vous trouverez une vidéo de principe de la technologie Ion Torrent (Life). En supplément, la publication de juillet 2011 de Rothberg et al. dans Nature (pour quelques informations supplémentaires). Il est à noter pour les connaisseurs que Jonathan Rothberg est l’un des fondateurs de la la société 454 Life Sciences (ayant donné naissance au séquenceur haut-débit le plus utilisé dans un passé proche, utilisant le principe du pyroséquençage), technologie vendue à Roche et le créateur de la société Ion Torrent avant que celle-ci ne soit rachetée 350 millions de dollars par Life Technologies.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages