La confusion entre mate-pair et paired-end, tant au niveau technologique (selon qu’on lise les notes techniques d’Illumina, de Roche ou de Life) que logiciel nous a mené à rédiger, en collaboration avec Ségolène Caboche, Bioinformaticienne à l’université de Lille2, une note technique dont le contenu est résumé ci-dessous :
– Genèse de la confusion entre mate-pair et paired-end
– Descriptions les deux approches, avec un focus sur les principales technologies de seconde génération de séquenceurs
– Traitement au niveau logiciel et conseils généralistes pour l’utilisation
Le document est consultable dans son intégralité sur notre blog :
Télécharger Paired-end versus mate-pair
Bonne lecture!
Dernier né de la gamme des séquenceurs de paillasse, le « Ion Proton Sequencer » (149.000 $) est le premier de sa catégorie à permettre le séquençage en quelques heures du génome humain et à faible coût (1.000 $). A l’instar du PGM Ion Torrent, cette nouvelle plateforme est basée sur la technologie des semi-conducteurs (et la détection de la libération d’ions H+ suite à la polymérisation de dNTP).
La puce « Ion Proton ™ I »,disponible mi-2012, aura une capacité de séquençage 1000 fois supérieure au premier format de puce 314 du PGM Ion torrent soit 10 Gb (davantage orientée pour le séquençage d’exome humain). Le second format (puce « Ion Proton ™ II ») , disponible environ six mois plus tard aura une capacité de 100 Gb (Séquençage Génome humain).
Compte tenu de l’apparente similarité entre les deux plateformes de chez Life technologies, l’évolution réside essentiellement dans l’architecture de la puce avec une augmentation du nombre de puits ( 165 millions de puits pour la puce « Ion Proton ™ I » et 660 millions pour la puce « Ion Proton ™ II » soit respectivement 100 et 1000 fois plus que la puce 314 du PGM ), l’une des bases de la capacité de séquençage (en complément de la longueur des reads générés).
Le terme « Nouvelle génération de séquençage à haut-débit » ( ou « Next generation sequencing » ) regroupe l’ensemble des technologies ou plateformes de séquençage développées depuis 2005 par quelques sociétés de biotechnologies.
L’objectif de cet article est de proposer de manière synthétique, un tour d’horizon des différents principes et caractéristiques de ces nouveaux outils et ainsi fournir quelques orientations et solutions techniques en réponses à des questions biologiques.
La position actuelle dans laquelle nous nous trouvons, entre la commercialisation de certains séquenceurs et ceux en cours de développement, est caractéristique d’une période charnière dissociant les technologies à haut débit dites de 2ème génération qui requièrent une étape d’amplification des molécules d’ADN en amont du décodage, de celles dites de 3ème génération permettant le décryptage direct d’une seule molécule d’ADN. Cette dernière catégorie fera à elle seule l’objet d’un prochain article.
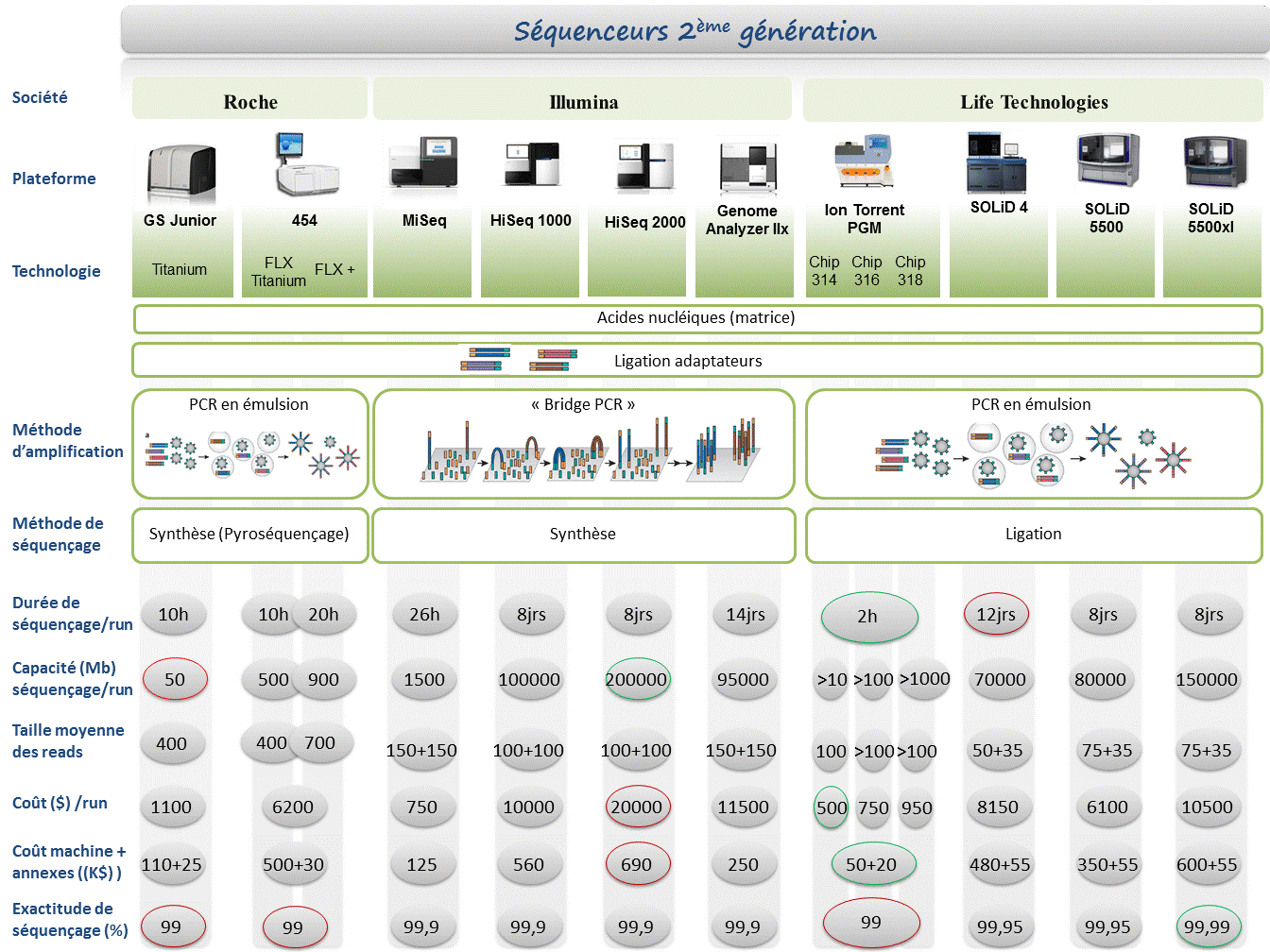
Le marché des séquenceurs de 2éme génération est couvert par 3 grand groupes que sont Roche, Illumina et Life Technologies ayant respectivement proposés de manière successive, leur première plateforme à savoir le 454, le Genome Analyser et enfin le SOLiD. Depuis, le marché s’est étoffé proposant un panel de technologies au principe et caractéristiques propres telles qu’elles sont mentionnées ci dessous. A noter que parmi ce panel, le PGM, Ion torrent est le seul a connaitre une évolution constante en terme de capacités de séquençage (10Mb – reads de 100b – Juin 2011 / 100Mb – reads de 100b – Sept 2011 / 100Mb – reads 200b – Nov 2011 / 1Gb – Jan 2012 )
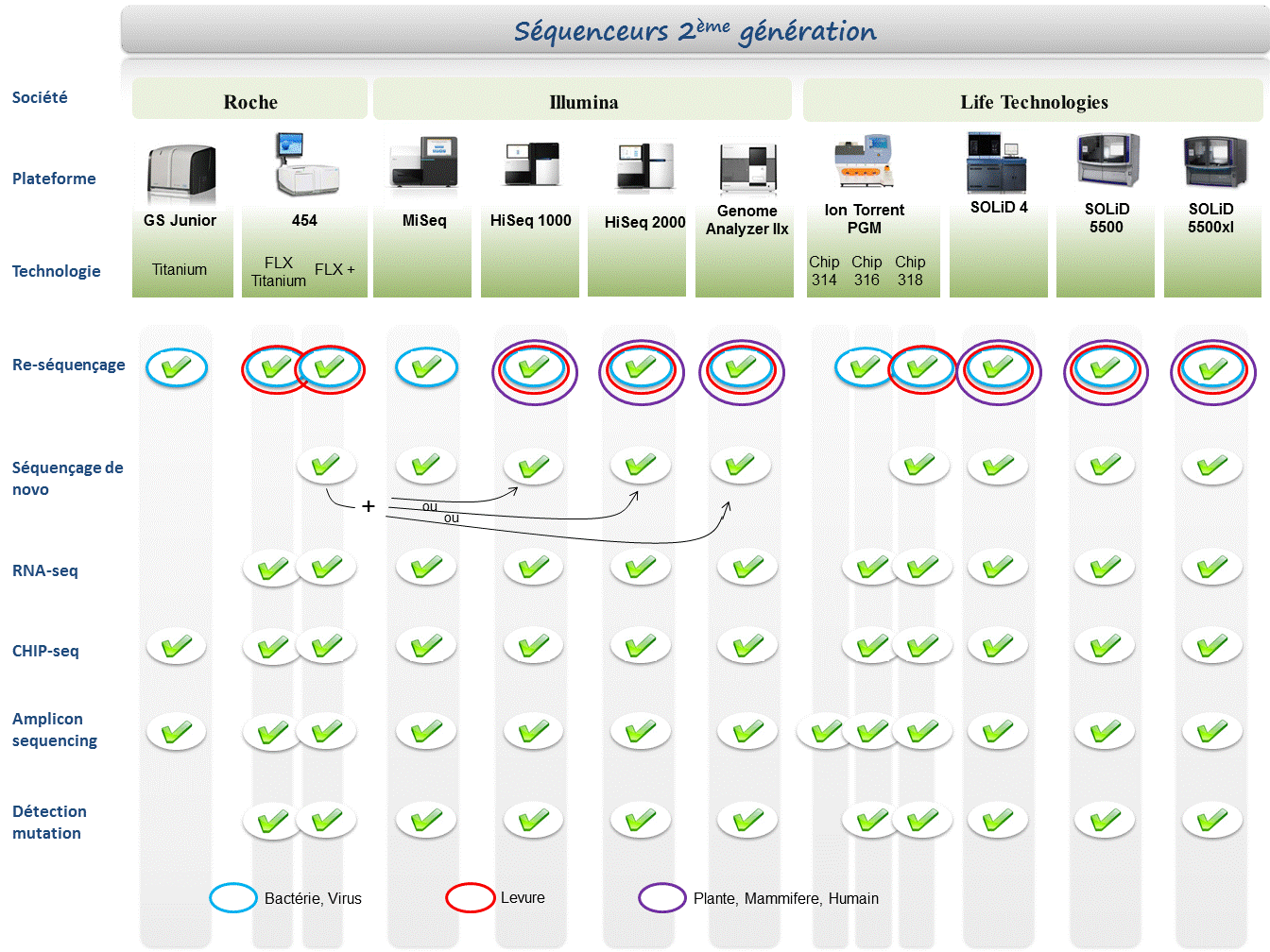
Chaque plateforme possède ses avantages et inconvénients et nombreuses sont celles configurées pour répondre à de nombreuses approches « omics », dans certaines limites. Il s’agira de faire un choix technologique selon les champs d’applications souhaités.
De manière générale, le type d’organisme étudié prédéterminera la technologie à employer. La notion de profondeur est récurrente à chaque application et dans l’objectif d’un reséquençage, le choix de la plateforme peut être identifié, de manière simplifiée, sur la base d’un calcul rapide ( P = N / L où P: Profondeur, N: Nombre des nucléotides totaux des reads, L: Taille du génome étudié).
Concernant les séquenceurs de 2ème génération, le séquençage de novo est une application mentionnée chez de nombreux fournisseurs (cf le tableau ci-dessous). Toutefois, l’association de deux technologies générant à la fois des reads longs (type 454, Roche) et une profondeur conséquente (type GAIIx, Illumina) palliant aux problèmes liés aux homopolymères et erreurs de séquençage, est préconisée (Au cours de l’article à venir sur les séquenceurs de 3ème génération, nous aborderons les plateformes davantage configurées pour cette application).
Ce paramètre de profondeur sera également à prendre en considération pour les champs d’applications incluant la notion d’analyse quantitative (RNAseq, ChIPseq, …). Si la profondeur permet d’atténuer les erreurs de séquençage, il reste néanmoins préférable de s’orienter vers des technologies à Q30 minimum (1 erreur sur 1000) pour la détection de SNPs.
Selon les technologies évoquées ci-dessus, les caractéristiques et champs d’applications ont évolués. Aussi, je vous propose de retrouver l’ensemble de ces informations actualisées en cliquant sur ce lien.
L’ensemble des informations sont détaillées dans l’article mentionné ci-dessous:
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages