La confusion entre mate-pair et paired-end, tant au niveau technologique (selon qu’on lise les notes techniques d’Illumina, de Roche ou de Life) que logiciel nous a mené à rédiger, en collaboration avec Ségolène Caboche, Bioinformaticienne à l’université de Lille2, une note technique dont le contenu est résumé ci-dessous :
– Genèse de la confusion entre mate-pair et paired-end
– Descriptions les deux approches, avec un focus sur les principales technologies de seconde génération de séquenceurs
– Traitement au niveau logiciel et conseils généralistes pour l’utilisation
Le document est consultable dans son intégralité sur notre blog :
Télécharger Paired-end versus mate-pair
Bonne lecture!
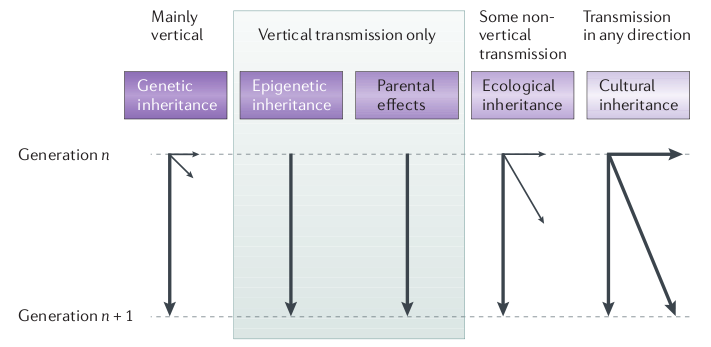
Pierre Barthélémy, journaliste scientifique au monde, a publié hier sur son blog (passeurdesciences.blog.lemonde.fr) une interview de Etienne Danchin (directeur de recherche CNRS et directeur de l’EDB) qui nous parle de la nécessité de s’extraire du dogme du « tout-génétique » pour s’intéresser plus précisément à d’autres mécanismes qui régissent l’hérédité, tel que l’épigénétique, l’hérédité culturelle et environnementale.
Vous pouvez retrouver l’interview dans son intégralité ici :
Cette interview fait suite à la publication d’une synthèse sur l’hérédité non génétique et son rôle dans l’évolution parue dans Nature Reviews Genetics en Juillet 2011 que vous pouvez consulter en intégralité ici.
Etienne Danchin n’est pas le seul à s’interroger sur la part réelle de la génétique dans les mécanismes de l’hérédité, nous vous conseillons une nouvelle fois l’excellente émission de Jean Claude Ameisen (à écouter dans ce post) qui aborde ces aspects de la transmission génétique et non génétique, entre générations.
L’hérédité culturelle est une composante en général peu discutée (notamment dans le domaine animal): quelle est la part de variation phénotypique qui peut être expliquée par une transmission sociale (ou transmission par apprentissage)? A noter que le rôle de la composante culturelle dans l’hérédité a été formalisé dans les années 1970, ça ne date donc pas d’hier, mais comme toutes les composantes non génétiques impliquées dans l’hérédité elle s’est retrouvée minimisée, voire ignorée, au profit de la composante génétique.
Quoi qu’il en soit, à l’heure de l’arrivée de la 3eme génération de séquenceur haut-débit et de la génération toujours plus astronomique de données génomiques, il est toujours bon de rappeler que le seul transfert de gènes n’explique qu’une proportion des mécanismes étudiés.
Pierre Barthélémy, journaliste scientifique à publié hier sur son blog (passeurdesciences.blog.lemonde.fr) une interview de Etienne Danchin (directeur de recherche CNRS) qui nous parle de la nécéssité de sortir du dogme du « tout-génétique » pour s’intérésser plus précisement à d’autre mécanisme qui régisse l’hériédité, l’épigénétique, l’hérédité génétique et environementale.
Vous pouvez retrouver l’interview dans son intégralité ici :
Cet interview est le résultats d’une synthèse sur l’hérédité non génétique et son rôle dans l’évolution publié dans Nature Reviews Genetics en Juillet 2011 que vous pouvez consulter en intégralité ici.
Etienne Danchin n’est pas le seul à s’intéroger sur la place réelle de la génétique dans les mécanisme de l’hérédité, nous vous conseillons une nouvelle fois l’excellente émission de Jean Claude Ameisen (à écouter dans ce post).
Une revue intéressante et qui se veut exhaustive sur les conséquences de la généralisation des technologies de séquençage et les solutions/adaptations possibles, on y retrouve pèle-mêle :
– Un listing à jour (2011) des différentes plateformes dédiées à la génération de données de séquençage (Illumina, Roche, Life Technologie pour ne citer qu’eux…) et leurs spécificités;
– La description de quelques stratégies de NGS : identification de variants, séquençage d’éxome, séquençage sur des régions précises…
– Les problématiques en bioinformatiques : stockage et analyse de données, développement de solutions logicielles adaptées…
– Les différentes analyses ainsi que des listes de logiciels pour répondre aux besoins: assemblage denovo et sur génome de référence, annotation et prédiction fonctionnelle, autant open-source que sous licence payante.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages