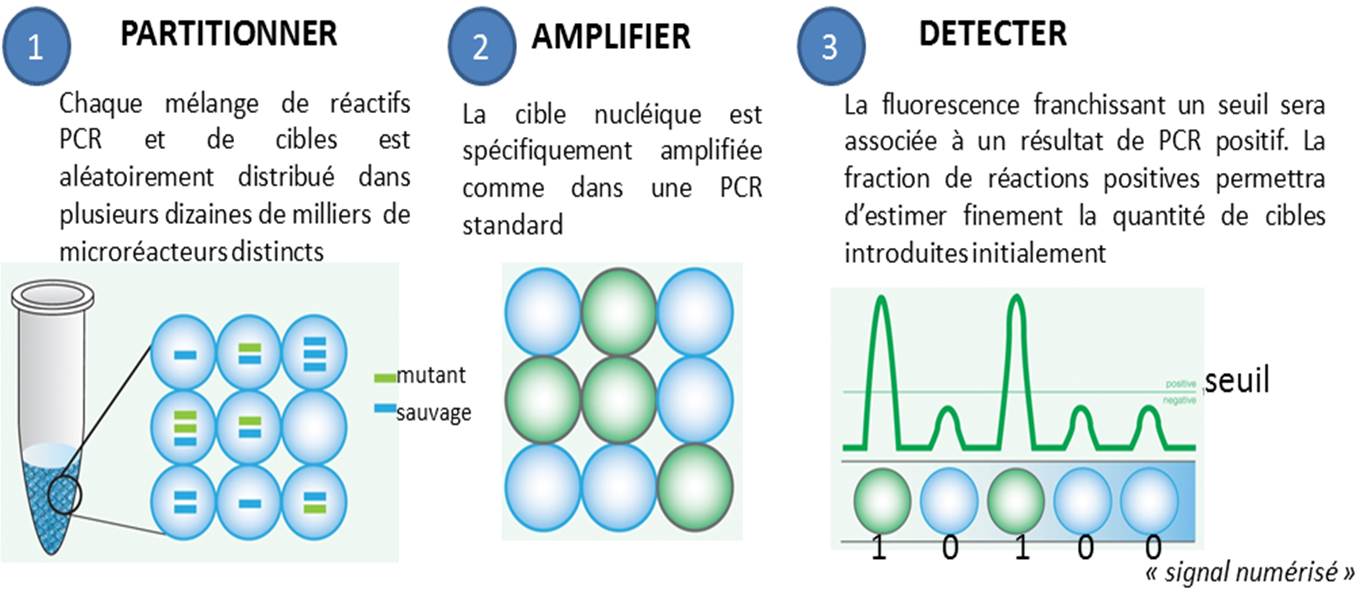
La PCR en point final, celle qui se termine souvent par un dépôt sur gel d’agarose, ainsi que la PCR quantitative, dans laquelle on suit l’évolution de la libération de molécules fluorescentes dont le décollage plus ou moins précoce dépend de la quantité de matrice initialement introduite, se voient déclinées en versions numériques. Par numérique est entendu ici, qu’une PCR peut prendre deux valeurs : 0 ou 1 (idéalement, 0 quand aucune cible n’est introduite au départ d’une PCR et que par voie de conséquence aucun signal issu d’amplification n’est observé, et 1 pour l’exact inverse).
Le principe de la PCR numérique est simple au niveau du concept mais techniquement beaucoup plus difficile à mettre en application. Il s’agit de multiplier le nombre de bio-réacteurs disponibles, ainsi, un échantillon sera partitionné en des milliers de compartiments distincts. Ensuite chaque compartiment sera considéré comme autant de réacteurs indépendants… donc en fin de PCR autant de réactions montrant oui ou non une amplification (autant de 0 et de 1), renvoyant à un système binaire. L’application de la Loi de Poisson permettra ensuite d’estimer très finement la quantité initiale de cibles présentes dans l’échantillon de départ.
Les avantages de la PCR numérique par rapport à la qPCR classique sont assez évidents :
– elle permet une quantification absolue sans établir une courbe standard préalable
– elle est plus sensible voire beaucoup plus sensible. Raindance annonce ainsi détecter un mutant parmi 250 000 molécules sauvages avec une limite inférieure de détection de 1 parmi 1.000.000
– elle est moins sensible aux inhibiteurs. Le fait de diluer la matrice ADN complexe de départ pour la répartir dans un nombre important de réacteurs permet de favoriser le départ de PCR
– elle est nettement plus précise. La technologie sera d’autant plus précise que le nombre de compartiments, le nombre de micro-réacteurs sera important
L’article « Digital PCR hits its stride » (La PCR numérique franchit un nouveau cap) de Monya Baker dans le Nature Methods (juin 2012) permet un aperçu de la technologie qui est passée du concept à des solutions commerciales qui se veulent de plus en plus accessibles.
Actuellement, plusieurs fournisseurs proposent des plateformes aux spécifications très différentes. Les quatre fournisseurs ci-dessous proposent des systèmes de PCR numérique basés sur de la microfluidique pour Fluidigm, des interfaces solides (type OpenArray) de Life Technologies et des micro-gouttes pour Raindance et Biorad.
Ainsi, que le mentionne Jim Huggett (chef d’équipe au LGC), cité dans la publication de Monya Baker (Digital PCR hits its stride), la PCR numérique est réservée à des utilisateurs experts et se trouve encore allouée à des applications spécialisées car encore beaucoup plus chère que la PCR quantitative (traditionnelle) qui reste adaptée à la majorité des applications.

A la croisée de la biologie, de la biochimie, de l’électronique, de la mécanique des fluidiques et de la thermodynamique, de nouveaux outils se préparent. Ceux-ci viendront peut être trouver des applications dans les laboratoires de génomique mais au-delà de cette perspective, ils pourraient trouver des applications au niveau des professionnels de santé eux-mêmes avant d’être accessibles aux clients-patients-citoyens directement.
Les Lab-on-Chips (ou Laboratoire sur puce) sont un concept qui a émergé dans les années 90 et qui au sein des laboratoires de biologie moléculaire a trouvé une application largement répandue sous la forme des puces Agilent consommables pour BioAnalyzer. La technologie d’Agilent, basée sur une électrophorèse ou une cytométrie de flux microfluidique, offre un outil de contrôle de la quantité, qualité (distribution de taille, intégrité des ARNs extraits etc.) des molécules considérées. Même si les puces Agilent sont affublées du qualificatif de « Lab-on-Chip« , elles nécessitent un appareillage dédié et bien entendu un PC relié. L’outil est encore loin du modèle rêvé, c’est-à-dire complètement autonome (en une version « stand alone »).
L’émergence de ce type de laboratoires a été, en partie, permise grâce à des budgets alloués à la lutte contre le bioterrorisme. En effet, des laboratoires qui tiennent dans une poche de treillis, qui peuvent être employés sur n’importe quel théâtre d’opération, peuvent venir parfaire le paquetage des experts en sécurité intérieure. Les laboratoires Sandia (Sandia National Laboratories) font partie des principaux laboratoires nationaux du département de l’Énergie des États-Unis et sont des pionniers de ces applications avec les développements réalisés par MicroChemLab qui est actif depuis 1988 sur ce type de développements.
Aujourd’hui les laboratoires unipersonnels de diagnostic embarqués sont devenus très populaires, pour des applications liées à des analyses biochimiques. L’un des exemples les plus fameux concerne le BG-Star (Sanofi Diabète) permettant de réaliser en quelques secondes, très aisément, des lectures de glycémie. L’outil est basé sur le principe de l’électrochimie dynamique où une bandelette jetable accueille une goutte de sang qui vient activer une glucose oxydase.
Les avantages de ce type de technologies :
– Une faible consommation de volume de fluide, en raison des volumes internes faibles des puces, ce qui minimise la quantité de déchets, baisse des coûts des réactifs onéreux et surtout minimise la quantité nécessaire d’échantillon de départ
– Une rapidité d’exécution où l’efficacité des réactions biochimiques est permise par la miniaturisation de l’outil, en effet la taille du dispositif ne nécessite qu’une faible quantité d’énergie pour fonctionner
– Un meilleur contrôle du processus en raison d’une réponse rapide du système (par exemple, le contrôle thermique pour des réactions chimiques exothermiques)
– Une parallélisation massive due à la compacité, qui permet l’analyse à haut-débit
– Une réduction des coûts de fabrication, permettant une utilisation de puces jetables, un processus qui deviendra rentable s’il est fabriqué en masse
– Une plateforme plus sûre pour les produits chimiques, les études radioactives ou biologiques en raison de la grande intégration du système (souvent en système clos), des faibles quantités de réactifs et d’échantillons nécessaires
Les vertus de la miniaturisation ne nécessitent pas plus amples apologies ! Depuis quelques années l’idée d’un laboratoire qui tient au creux de la main a réellement commencé à voir le jour.
Des exemples de ces laboratoires portatifs commencent à poindre dans des versions de plus en plus intégrées.
Ainsi la technologie Mondrian développée par NuGEN, technologie basée sur de la microfluidique digitale, permet de simplifier les étapes de constitution de librairie de séquences haut-débit.
Destinagenomics propose un système analytique ne faisant pas intervenir d’enzymes. Ce système fait intervenir des sondes interrogatives de mutations que la société souhaite embarquer dans des puces : la preuve du concept a été publiée par Pernagallo et al. (Sensors (Basel). 2012).
http://youtu.be/k-NnBS2xDLo
Panasonic, le groupe japonais, numéro 4 mondial de l’électronique, en partenariat avec la société belge, Imec, propose d’embarquer dans une puce d’ 1 cm2 : une pompe permettant aux fluides de circuler, une unité d’extraction d’ADN à partir de la goutte de sang prélevée, un thermocycleur, une unité de purification de produits de PCR, et un système de détection de SNPs.

Le concept est séduisant, mais pour être viable et concurrencer les coûts de génotypages basse densité qui sont relativement bas (quelques euros / marqueur), les concepteurs des Laboratoires sur Puces misent donc sur une large diffusion de leur produit. Ainsi la production de masse impacte drastiquement le coût de production qui lui-même est impacté sur le prix vente… un prix de vente attractif et une simplicité d’utilisation qui dope les ventes… et donc fait baisser les coûts de production…
Le pari réside dans le fait d’amorcer ce cercle vertueux, en quelque sorte chaque opérateur du marché – au niveau d’une application donnée- souhaite assommer la concurrence ! Une course au développement est donc lancée.
Revenons à la science ! Vous trouverez ici le document pdf. présentant la technologie de Panasonic/Imec,
Concernant l’application de séquençage, Oxford Nanopore proposera une « clé USB » capable de séquencer, le Minion.
Dans le futur il est envisageable, d’imaginer un outil combinant la technologie de séquençage de 3ème génération, d’un Oxford Nanopore, par exemple, avec en amont un dispositif intégrant (à l’instar du projet de Panasonic), une micro-pompe, un réseau microfluidique permettant d’alimenter en échantillon et réactifs les différents éléments permettant une extraction d’ADN/ARN, la constitution d’une librairie, une purification…
Parle-t’on ici de science fiction ? Pour un début de réponse, il faut comprendre que séparément toutes ces solutions technologiques existent déjà.
Pour les plus férus de laboratoires miniaturisés, un journal scientifique est là pour eux, explicitement nommé : Lab on a Chip.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages