La qPCR est une méthode permettant de doser la quantité d’acides nucléiques ciblés introduits dans une réaction de PCR. Pour des raisons de rapidité, de sensibilité et de coût, souvent l’option de travailler avec un agent intercalant (sans sonde) est choisie. La bonne vieille qPCR SybGreen nécessitant le seul design d’une paire d’amorces…
Simple ? Pas nécessairement, car cette approche, certainement plus que la version qPCR Taqman nécessite un travail in silico et de validation/optimisation expérimentales comme passages obligés. C’est ce que montre la publication de Stephen Bustin et Jim Huggett dans Biomolecular Detection and Quantification. Cette publication incontournable pour les férus de qPCR SybrGreen est un beau travail pour lequel la publication vous est mise à disposition en cliquant ci-dessous. On attend ardemment une déclinaison Taqman, HRM, MolecularBeacon de ce type de revues permettant de formaliser des procédures visant à optimiser l’approche d’optimisation.
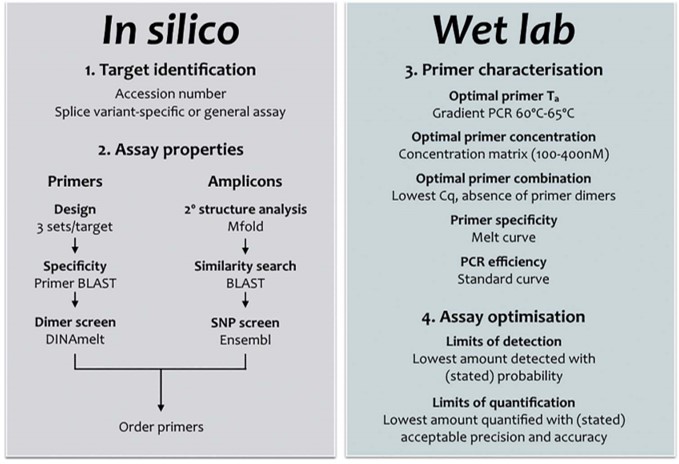
La quantification par qPCR SybrGreen suppose une relation linéaire entre le logarithme de la quantité initiale introduite en PCR et la valeur Cq obtenue lors de l’amplification. Ceci permet de calculer l’efficacité d’amplification d’un test et de borner ses limites de détection et de quantification. Les caractéristiques d’un test qPCR (bien) optimisé sont les suivantes:
• Une excellentissime spécificité révélée par un pic unique lors de l’établissement de la courbe de fusion
• Une efficacité d’amplification élevée (95-105%)
• Une courbe étalon linéaire (R2 > 0,980)
• Une bonne répétabilité
• Peu ou prou de dimères d’amorces
Pour paraphraser la conclusion de l’article, afin de finir par convaincre de lire cet « essentiel » de la qPCR :
La conception, le design d’une PCR est souvent au cœur de tout projet de recherche visant à quantifier les acides nucléiques. Il doit être réalisé avec soin, mais peut être simplifié en suivant un flux de travail simple, comme décrit ci-dessus (cf. diagramme workflow design qPCR).
Cela signifie généralement une spécificité absolue, l’absence de structures en épingle à cheveux ou de potentielles dimérisations croisées. Une bonne conception des essais doit tenir compte de la structure de l’amplicon (paramètre souvent négligé) et veiller à ce que les cibles de l’amorce soient exempts de structure secondaire. Il existe de nombreuses opinions et lignes directrices; une recherche sur Internet pour les termes « qPCR Assay Design » renvoie 695.000 pages. Cependant, bon nombre de ceux-ci sont basés sur des mythes ou peuvent être appropriés pour la PCR mais nécessitent des modifications subtiles (ou moins subtiles) pour être utilisés pour développer une qPCR. Chaque « nouveau » dosage doit être correctement validé, la validation in silico servant de filtre initial pour éliminer des designs ne permettant pas d’aboutir à une bonne qPCR. L’optimisation et la validation empirique sont une partie essentielle, mais souvent négligée, de toute expérience qPCR. Cela s’applique aussi bien aux essais nouvellement conçus qu’aux essais obtenus en reprenant des amorces issues d’une publication, par exemple. Avec tant d’essais prêts à l’emploi, on peut se demander pourquoi quelqu’un voudrait se donner la peine de concevoir un autre essai. D’autant plus que l’on a l’impression que la conception de son propre test est beaucoup plus complexe et peu commode que de simplement l’acheter à un fournisseur commercial, qui en tout cas aura validé chacun de ses tests. Cette perception est erronée pour deux raisons:
1° il se peut que les amorces commerciales ou les conditions d’analyse n’aient pas été validées ou optimisées de façon expérimentale.
2° on ne peut pas présumer qu’un ensemble d’amorces produira les mêmes résultats dans des conditions expérimentales différentes puisque la performance du dosage peut varier selon les méthodes d’extraction utilisées pour purifier les acides nucléiques.
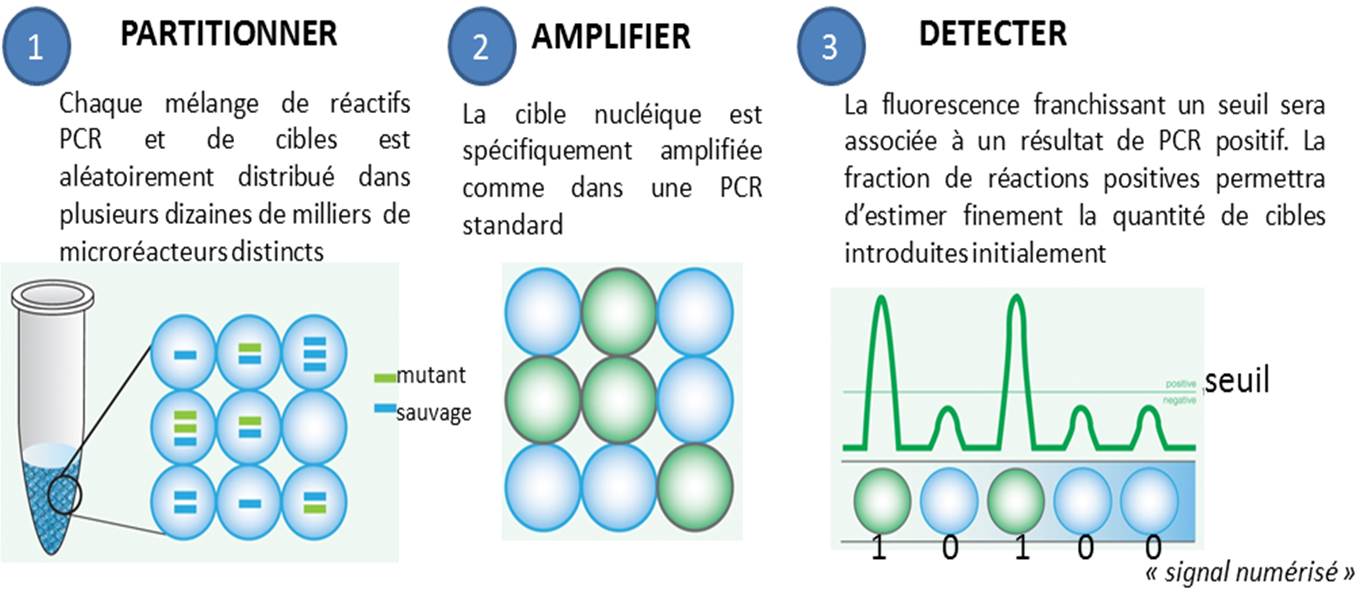
La PCR en point final, celle qui se termine souvent par un dépôt sur gel d’agarose, ainsi que la PCR quantitative, dans laquelle on suit l’évolution de la libération de molécules fluorescentes dont le décollage plus ou moins précoce dépend de la quantité de matrice initialement introduite, se voient déclinées en versions numériques. Par numérique est entendu ici, qu’une PCR peut prendre deux valeurs : 0 ou 1 (idéalement, 0 quand aucune cible n’est introduite au départ d’une PCR et que par voie de conséquence aucun signal issu d’amplification n’est observé, et 1 pour l’exact inverse).
Le principe de la PCR numérique est simple au niveau du concept mais techniquement beaucoup plus difficile à mettre en application. Il s’agit de multiplier le nombre de bio-réacteurs disponibles, ainsi, un échantillon sera partitionné en des milliers de compartiments distincts. Ensuite chaque compartiment sera considéré comme autant de réacteurs indépendants… donc en fin de PCR autant de réactions montrant oui ou non une amplification (autant de 0 et de 1), renvoyant à un système binaire. L’application de la Loi de Poisson permettra ensuite d’estimer très finement la quantité initiale de cibles présentes dans l’échantillon de départ.
Les avantages de la PCR numérique par rapport à la qPCR classique sont assez évidents :
– elle permet une quantification absolue sans établir une courbe standard préalable
– elle est plus sensible voire beaucoup plus sensible. Raindance annonce ainsi détecter un mutant parmi 250 000 molécules sauvages avec une limite inférieure de détection de 1 parmi 1.000.000
– elle est moins sensible aux inhibiteurs. Le fait de diluer la matrice ADN complexe de départ pour la répartir dans un nombre important de réacteurs permet de favoriser le départ de PCR
– elle est nettement plus précise. La technologie sera d’autant plus précise que le nombre de compartiments, le nombre de micro-réacteurs sera important
L’article « Digital PCR hits its stride » (La PCR numérique franchit un nouveau cap) de Monya Baker dans le Nature Methods (juin 2012) permet un aperçu de la technologie qui est passée du concept à des solutions commerciales qui se veulent de plus en plus accessibles.
Actuellement, plusieurs fournisseurs proposent des plateformes aux spécifications très différentes. Les quatre fournisseurs ci-dessous proposent des systèmes de PCR numérique basés sur de la microfluidique pour Fluidigm, des interfaces solides (type OpenArray) de Life Technologies et des micro-gouttes pour Raindance et Biorad.
Ainsi, que le mentionne Jim Huggett (chef d’équipe au LGC), cité dans la publication de Monya Baker (Digital PCR hits its stride), la PCR numérique est réservée à des utilisateurs experts et se trouve encore allouée à des applications spécialisées car encore beaucoup plus chère que la PCR quantitative (traditionnelle) qui reste adaptée à la majorité des applications.
 La PCR est l’un des outils de base dans la boîte du biologiste moléculaire. Devenue accessible, totalement démocratisée dans les laboratoires, elle demande cependant une certaine exigence dans son développement.
La PCR est l’un des outils de base dans la boîte du biologiste moléculaire. Devenue accessible, totalement démocratisée dans les laboratoires, elle demande cependant une certaine exigence dans son développement.
Dans un premier temps il est essentiel de définir à quel type de PCR nous avons à faire :
– PCR de détection (diagnostic ?) : celle qui vous permet de croire que vous avez dans votre extraction l’agent infectieux que vous tâchez de cibler
– PCR de monitoring : celle qui vous suffit pour admettre que vous validez ou non l’expérience que vous désirez suivre, celle qui vous dit que vous avez contaminé votre extraction d’ARN en ADN génomique, par exemple
– PCR de production : celle qui vous permet d’obtenir le plus de produit spécifique afin d’exploiter ces quelques centaines de paires de bases pour en faire ce que vous avez imaginé (reporters à spotter sur puce ADN ou insert pour un clonage)
– PCR quantitative : celle qui se résume à estimer du mieux possible la quantité d’acides nucléiques ciblés présents initialement dans une PCR (cDNA, cible pathogène…).
Il est fondamental de reconnaître à quel genre s’apparente la PCR dont vous avez designé les amorces avec le plus grand soin. Peut être parce que les objectifs visés sont réellement différents voire opposés.
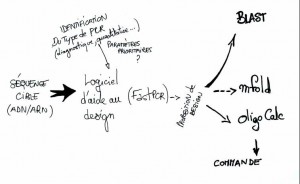
Les grandes étapes aboutissant à un design sont résumées ci-dessus. Il convient d’extraire en format FASTA, la séquence qui vous servira de matrice PCR, le logiciel d’aide permettra d’écarter les amorces de qualités douteuses (auto-appariées, peu spécifiques…), la proposition de design sera soumise dans un Blast (dont les paramètres dépendront du type d’application de votre PCR), dans le cas d’une qPCR SybrGreen il peut être intéressant d’utiliser un mFold sur l’amplicon que vous souhaitez générer, enfin l’ulitsation d’un autre service web tel que OligoCalc permettra de valider les paramètres de vos amorces en confrontant ceux-ci à ceux que vous aurez obtenus en sortie de logiciel d’aide au design.
Commençons par aborder les paramètres d’importances de cette technique de biologie moléculaire : la spécificité, la sensibilité, l’efficacité. Quand la PCR de détection cumulera sensibilité et spécificité, la PCR de monitoring devra être efficace et simple à mettre en place (quelques heures de développement entre ces deux variantes…). La première étape, la plus essentielle, consistera à designer des amorces (nous parlerons des sondes ultérieurement). Avant cela il vous faudra vous doter d’une stratégie de design et peut être d’un (le mieux serait plusieurs) logiciel d’aide au design d’amorces. Parmi tous les logiciels disponibles, rien ne sert de casser le cochon, il existe beaucoup de gratuiciels acceptables pour la grande majorité des applications. Parmi ceux-ci, notons :
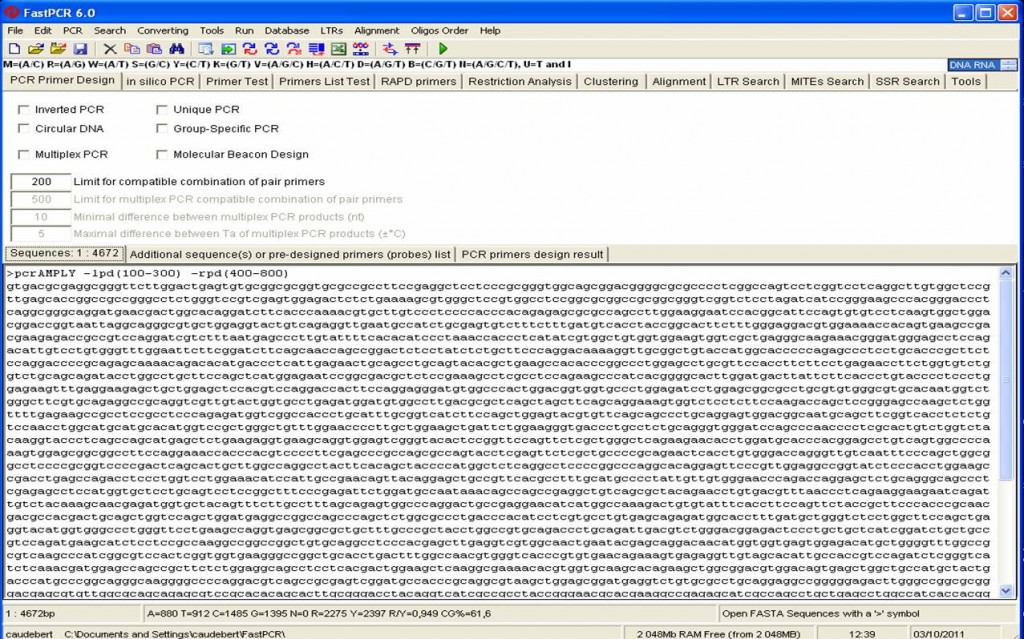
– FastPCR : quelques bugs, mais globalement cette solution à notre préférence pour sa modularité. Le logiciel gère les PCRs multiplexes, propose un grand nombre d’outils intégrés de manipulations de séquences. Il s’agit de l’une des seules solutions gratuites qui est l’équivalent d’une solution payante (mode d’emploi FastPCR disponible). Le logiciel quant à lui est disponible ici (dans sa version antérieure, plus stable, dont nous voyons une capture d’écran ci-dessous).
Parmi les critères généraux, les paramètres de design transposables à tout type de PCR sont les suivants :
– Longueur des oligonucléotides = 16 à 26 nt
– Taux de GC = 40 à 70% (idéalement autour des 55 %)
– Tm des oligonucléotides ~ 60°C (si vous développez une PCR diagnostique la température d’amorçage = Tm, si vous souhaitez produire il faudra diminuer de 2°C la température d’amorçage par rapport au Tm des amorces)
Du GC%, de la concentration en amorces et de la concentrations en cations dépend le Tm (définie comme la température pour laquelle 50 % d’une séquence donnée est sous forme double hélice et 50 % est sous forme simple brin). C’est donc ces paramètres qu’il faut intégrer lors du design. Bien maîtrisé, un design permettra de gagner un temps précieux lors des phases de mise au point et d’optimisation d’une PCR. Ceci étant particulièrement vrai pour ce qui concerne la mise au point d’une PCR quantitative.
D’autres paramètres évidents concernent la séquence ciblée en tant que telle. Sachant qu’en fonction de celle-ci la marge de manœuvre est plus ou moins grande.
– maximiser autant que possible les zones de grandes complexité linguistique (celle avec le moins d’homopolymères, FastPCR tient compte de cette notion pour designer des amorces et se sert de ce paramètre pour calculer son indice de qualité de design)
– en 3′ des amorces, sur les 5 dernières bases, autant que possible, rassembler un maximum de bases A ou T (3 A ou T sur ces 5 dernières bases) afin d’en diminuer le Tm en 3′ et ainsi d’accroître la spécificité des amorces designées
Selon le type de PCR à laquelle nous avons à faire les contraintes de design peuvent être radicalement différentes :
– ainsi, vous souhaiterez designer dans 2 exons différents lorsque vous souhaiterez suivre la contamination en ADNg d’une extraction d’ARN par exemple
– ainsi vous souhaiterez designer sur une région à cheval sur 2 exons (sur la base donc d’une séquence cDNA, ARN) pour les applications de qPCR liées à la mesure du niveau de modulation différentielle de votre ARN cible
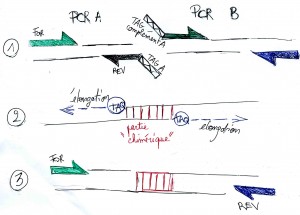
Truc et astuce pour diminuer vos coûts de séquençage d’amplicons en SANGER : la PCR hybride !
1/ Réalisation de PCR en tubes indépendants, l’amorce reverse (PCR A) et l’amorce forward (PCR B) des 2 PCR auront des séquences Tag synthétiques complémentaires
2/ La deuxième étape consistera en une élongation unidirectionnelle. Les 2 amplicons synthétisés lors de la PCR 1 s’apparieront grâce à leur séquence Tag complémentaire. Le produit de la PCR2 sera un hybride PCR A-séquence chimérique – PCR B
3/ Une dernière PCR, prenant la Forward de PCR A et la Reverse de PCR B seront utilisées… le produit néosynthétisé pourra être séquencé (2 ou plus pour le prix d’un, en outre bien optimisée ce type de PCR peut permettre d’amplifier des « signaux faibles »).
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages