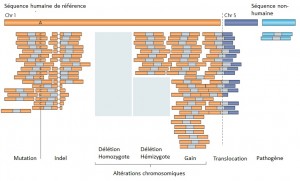
Le séquençage massif parallélisé palliant les limites des anciennes méthodes, a depuis ces dernières années amorcé de nouvelles perspectives, comme l’étude d’altérations génomiques pour la compréhension de nombreuses maladies rares (Plan National Maladies Rares). De plus, la mise sur le marché des derniers séquenceurs de paillasse aux visées hospitalières (positionnement de marché de Ion torrent avec son PGM) contribue également au développement de nombreux diagnostics cliniques.
Face à cette révolution, il convient de faire face au goulot d’étranglement de l’analyse et de l’interprétation, en faisant appel à une stratégie plus appropriée que le séquençage de génome complet et qui consiste à focaliser les efforts de décodage sur les régions codantes enrichies (Exome, gènes cibles, etc…) par une étape préliminaire de d’enrichissement d’exons. Tout en contribuant à une diminution des coûts (on ne séquence que ce qui semble pertinent…), elle permet entre autre d’augmenter la profondeur de séquençage des régions d’intérêts, paramètre indispensable pour l’identification d’altérations génomiques.
Succinctement, les techniques d’enrichissement sont multiples et peuvent être regroupées en trois catégories sur la base de leur principe technique:
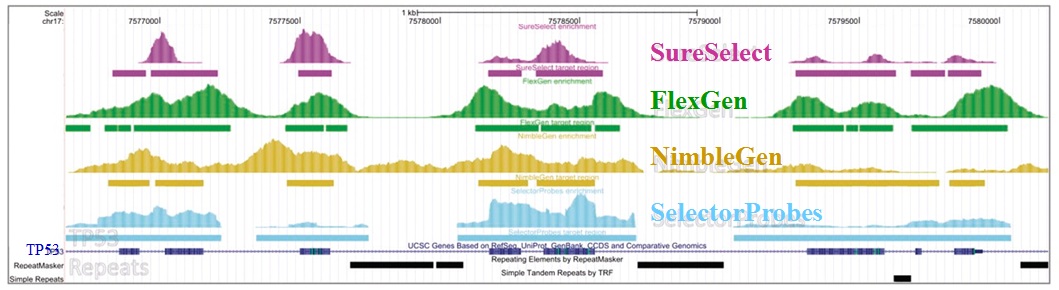
– Enrichissement par capture d’hybrides : La capture par hybridation des régions cibles est initiée à partir de la librairie de séquençage. Elle peut être effectuée soit en solution (billes magnétiques) soit sur support solide (microarrays). Récemment, de nombreuses solutions (commerciales ou « à façon ») ont été développées: SureSelect, Agilent – SeqCap EZ, Roche NimbleGen – Sequence Capture Arrays, Roche Nimblegen – FleXelect, FlexGen – MYselect, MYcroarray – TargetSeq, Life Technologies .
– Enrichissement par circularisation de fragments d’ADN : Cette méthode est supérieure à la précédente en terme de spécificité. En amont de la préparation de la librairie, les fragments d’ADNg (fragmentation enzymatique ou mécanique selon la méthode) sont enrichis via une sonde constituée d’une séquence universelle flanquée aux extrémités de séquences spécifiques de la région cible. HaloPlex, Agilent.
– Enrichissement par PCR : Cette approche intervient avant la préparation de la librairie et consiste grossièrement en une PCR multipléxée ciblant les régions d’intérêts. Une étape préliminaire de design des amorces est requise. SequalPrep, Life technologies. Tout juste disponible, Ion AmpliSeq Designer, Life Technologies propose un PCR Ultra-Plex (jusqu’à 1536 amplicons) avec un étape de digestion des amorces permettant de ne conserver que les régions d’intérêts lors du séquençage.
Une technologie récente et élégante faisant appel à la microfluidique et l’émulsion PCR permet l’amplification multipléxée (jusqu’à 20000) en micro-gouttelettes (une paire d’amorces par microréacteur) en un seul tube. Multiplicom, Fluidigm – RainDance.
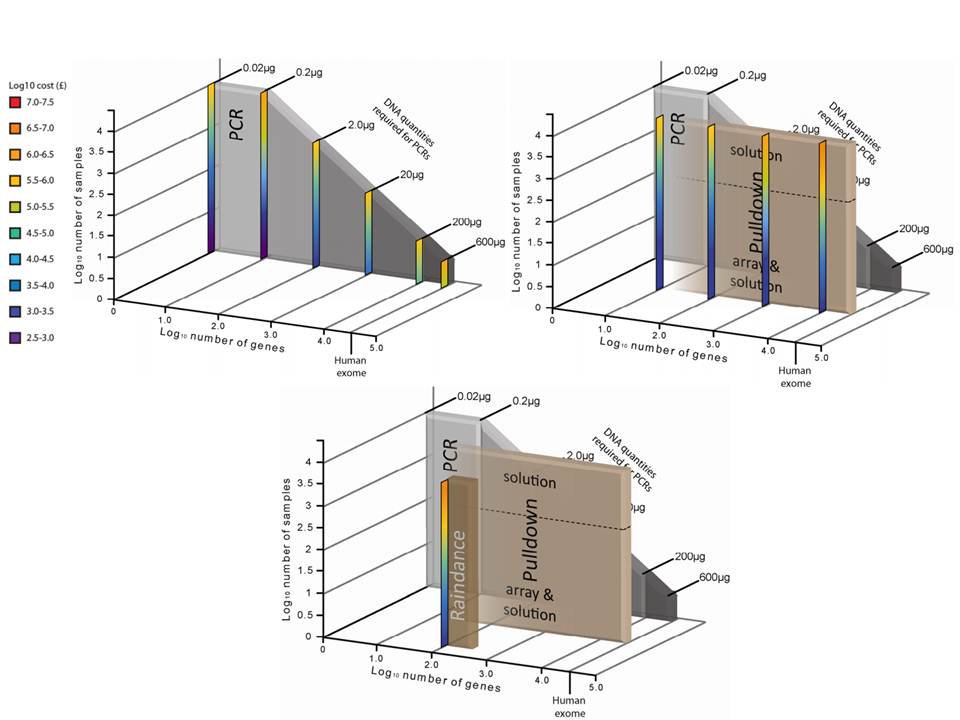
Ceci étant, les considérations fondamentales dans l’analyse de variants résident notamment dans:
– la profondeur de séquençage
– l’homogénéité de couverture des régions d’intérêt
– la reproductibilité de la méthode
– la quantité d’ADN « Input » requis
– le nombre d’échantillons à traiter
– le coût global
L’étude récemment menée par Florian Mertes et al. (nov. 2011) souligne l’importance de ces notions et fait le postulat de performances de captures de régions cibles significativement différentes selon la méthode d’enrichissement employée.
Il est impossible d’identifier une méthode d’enrichissement comme étant la meilleure notamment parce que ces approches sont en continuelle évolution et amélioration. Chacune d’elles répondent à des caractéristiques particulières et à des applications distinctes.
Si l’enrichissement par circularisation offre davantage de spécificité mais moins d’uniformité, l’inverse est aussi valable pour l’enrichissement par capture d’hybrides. Parmi les considérations dans le choix d’une méthode, le nombre d’échantillons et la taille des régions cibles sont essentiels. Si la capture d’hybrides est privilégiée pour l’analyse de mégabases (étude de l’exome) avec un nombre limité de cas, cette méthode sera délaissée au profit d’une approche PCR multipléxée pour l’étude d’un nombre restreint de régions cibles de petite taille appliquée à un grand nombre d’échantillons (Diagnostic).
L’association des méthodes d’enrichissement au séquençage haut débit offre des capacités technologiques ouvrant la voie vers de nombreuses perspectives. Toutefois, si l’émergence des séquenceurs de troisième génération s’accompagne à nouveau d’une diminution des coûts de séquençage, on est en droit de se demander si cette option restera aussi attractive.
 L’exome humain, l’ensemble des exons ou encore l’ensemble de ce qui, encodé sous forme d’ADN, peut être traduit en protéines représente environ 1,2% genome humain, 50 Mb et inclus 85 % des mutations référencées.
L’exome humain, l’ensemble des exons ou encore l’ensemble de ce qui, encodé sous forme d’ADN, peut être traduit en protéines représente environ 1,2% genome humain, 50 Mb et inclus 85 % des mutations référencées.
Emilie Lalonde et al., montre l’efficacité du séquençage d’exomes dans un article paru dans Human Mutation de 2010. La démonstration est faite au niveau de la découverte rapide des mutations associées au syndrome de Fowler, impliqué dans la prolifération anarchique des vaisseaux du cerveau qui empêche son développement. Etudiant les exons de deux patients sans lien de parenté cette équipe de Montréal a démontré l’efficacité de la capture d’exons (SureSelect, Agilent) suivi d’un séquençage haut-débit sur plateforme Illumina GAIIx (en moyenne pour les 2 expériences ont été générés 70 millions reads de 76 pb, environ 5 Gbases de séquences brutes).
Techniquement le séquençage d’exons est relativement simple : il implique une capture par hybridation spécifique (NimbleGen, SureSelect de Agilent sont les plus employés), une purification de ces exons ou parties d’exons capturés et le séquençage haut-débit de ceux-ci… L’article de Majewski et al., disponible ici (J. Med. Genet. 2011) cerne avec brio le formidable potentiel de ces nouvelles études, avec son « qu’est ce que le séquençage d’exons peut faire pour vous ? ». Il est assez aisé de comprendre que le séquençage des régions codantes est plus accessible au niveau budget et souvent suffisant pour répondre à beaucoup de questions de cliniciens travaillant à la recherche des causalités de maladies rares. Le schéma ci-dessous, tiré de Leslie G Biesecker, Nature Genetics 42, 13–14 (2010), synthétise la procédure permettant d’aboutir rapidement à la notion de « gènes candidats » (ou plutôt de mutations causales). Les mutations témoins sont accessibles sur les bases de données mondiales dbSNP ou bien encore par l’intermédiaire du projet 1000 Génomes… dont nous avons parlé dans de précédents articles.

L’article montre à quel point l’investigation visant à la découverte des causalités d’une maladie rare, par exemple, a été totalement bouleversée. Ces technologies couplant capture d’exons et séquençage des régions codantes capturées ont d’ores et déjà montré la preuve de leur efficacité. L’article nous rappelle que le génome humain réalisé en 2001 a coûté plus de 2,7 milliards USD, qu’en 2008 un séquençage humain complet coûtait 1,5 millions de USD et qu’aujourd’hui ce même séquençage approche les 10 000 USD…
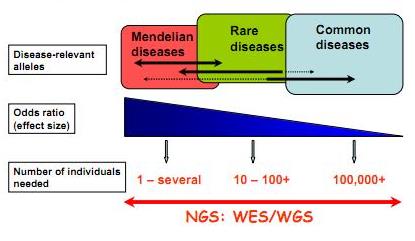
Feuille de route pour l'utilisation du séquençage haut-débit appliqué à l'identification des variations génomiques à l'origine de maladies "génétiques"
Ainsi que le montre Majewski et al. par le schéma ci-dessus plus la maladie que l’on cherche à élucider (si l’on peut s’exprimer ainsi) est la résultante d’une mutation qui a un effet fort, moins la cohorte à séquencer sera importante. En d’autres termes, pour ce qui concerne les maladies multi-factorielles (Alzheimer par exemple), le nombre d’individus dont les exons sont à séquencer sont importants… pour ce type de problématique les GWAS (Genome Wide Association Studies) par l’intermédiaire de scan de régions polymorphes anonymes sur la base de supports tels que les puces Illumina (puces permettant de scanner jusqu’à 4,3 millions de SNPs humains) restent compétitives et d’une puissance comparable. Dans le cas des maladies complexes d’autres stratégies sont développées telles qu’un premier crible sur une grande quantité de cas/témoins sur la base de puce haute densité de SNPs et un focus-séquencing des régions génomiques montrant une association potentielle avec le phénotype étudié…
Le séquençage haut-débit devenant de plus en plus accessible financièrement et par les méthodologies (pipeline d’analyses) de plus en plus disponibles permet d’envisager de nouvelles approches dans la découverte de cibles thérapeutiques potentielles. Ceci étant, l’accessibilité à des quantités de génomes de plus en plus disponibles (sans réel contrôle des organismes d’état) n’est pas sans soulever des problèmes d’éthique profonds et quelque peu négligé dans le débat scientifique actuel.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages