Galaxy : un workflow pour l’analyse bioinformatique
Il y a quelques semaines, nous avions discuté de l’utilisation de logiciel de Workflow pour la bioinformatique. Il est temps de passer à la pratique en vous présentant un de ses dignes représentants : Galaxy.
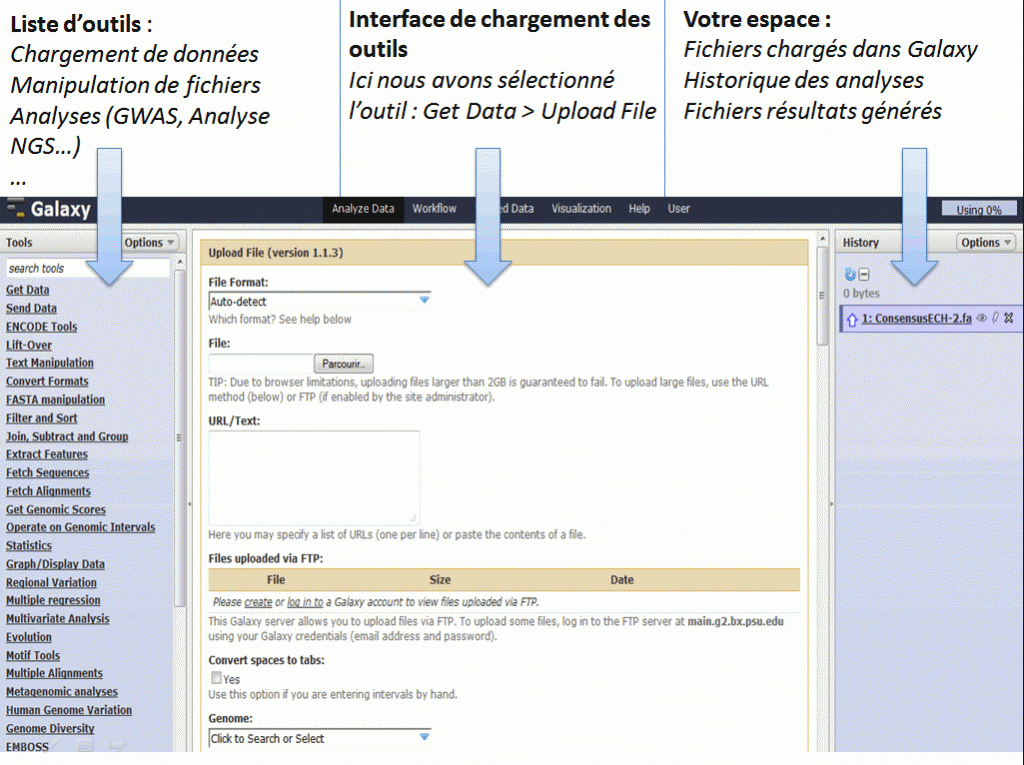
Le workflow Galaxy fournit un ensemble d’outils pour la manipulation et l’analyse de données génomiques. Il est très intuitif dans l’utilisation ce qui en fait une cible de choix pour le biologiste.
Il est possible d’utiliser Galaxy directement depuis le serveur. Avantage conséquent pour les bioinformaticiens il est possible d‘installer sa propre instance de serveur Galaxy, cette option fera l’objet d’un prochain post technique.
Du point de vue de l’interface graphique :
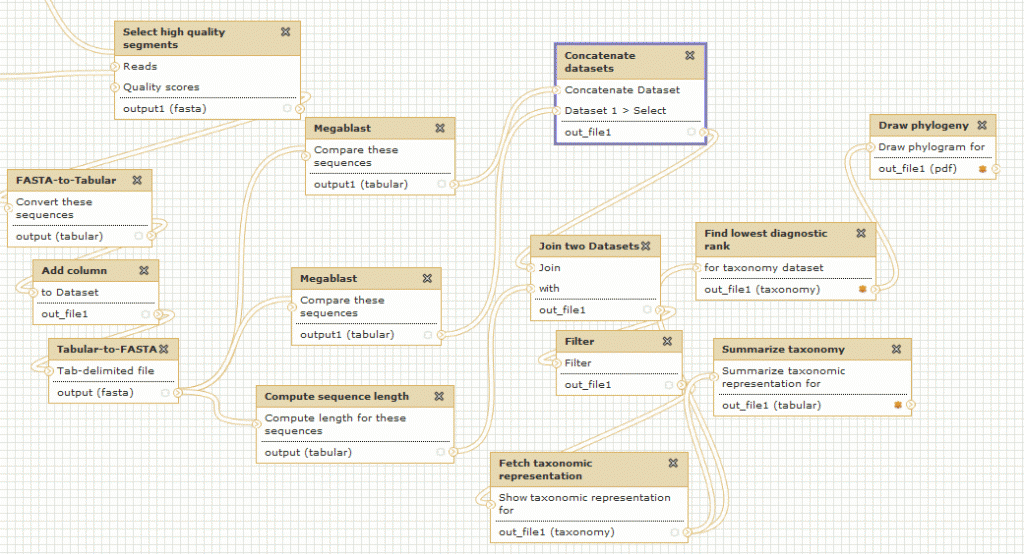
On peut également créer des workflows, les enregistrer dans un espace dédié, les partager, et les exécuter de façon automatique.
Pour exemple ce workflow de métagenomique publié gratuitement par un utilisateur de Galaxy (vous devez être connecté pour visualiser le workflow dans Galaxy)
Les outils dédiés analyse de données NGS sont régulièrement mis à jour et nul doute que d’ici peu, certains seront dédiés IonTorrent.
Les tutoriels sont également très bien faits, on apprend très vite à maitriser l’environnement grâce à des dizaines de vidéos d’aides.
Galaxy offre donc la possibilité d’exécuter des analyses bioinformatiques sans effort de programmation. La version en ligne est intéressante car elle permet de se familiariser aux logiciels et d’exécuter l’analyse depuis un portable, mais la possibilité d’intégrer ces propres outils (nous y reviendrons) est indéniablement un gros avantage de la version locale.
Si nous devions citer un inconvénient, plutôt d’actualité : l’utilisateur est obligé de charger ses données en mémoire dans Galaxy, le temps de chargement peut être très long si l’on manipule des données issues d’expériences NGS. D’autres workflows tels que Ergatis, fonctionnent en local et permettent à l’utilisateur d’utiliser directement les données présentent sur l’ordinateur.
Pour en savoir plus :
La description complète du logiciel Galaxy en Français sur PLUME :
http://www.projet-plume.org/fiche/galaxy
Le Galaxy Wiki :
http://wiki.g2.bx.psu.edu/FrontPage
La publication associée :
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages