on en est pas loin avec cet outil publié aujourd’hui dans Genome Biology
 Lors d’articles précédents nous vous avions présenté le logiciel de Workflow Galaxy, qui permet d’analyser et de visualiser toutes sortes de données biologiques à partir d’une interface simple d’utilisation.
Lors d’articles précédents nous vous avions présenté le logiciel de Workflow Galaxy, qui permet d’analyser et de visualiser toutes sortes de données biologiques à partir d’une interface simple d’utilisation.
Galaxy est en fait une brique d’une collection d’outils dédiés à l’analyse et au stockage de données biologiques : GMOD ( Generic Model Organism Database )
Le lundi 14 mai a été l’occasion pour nous d’assister à une conférence sur l’utilisation de certains des outils GMOD, dont voici les principaux enseignements :
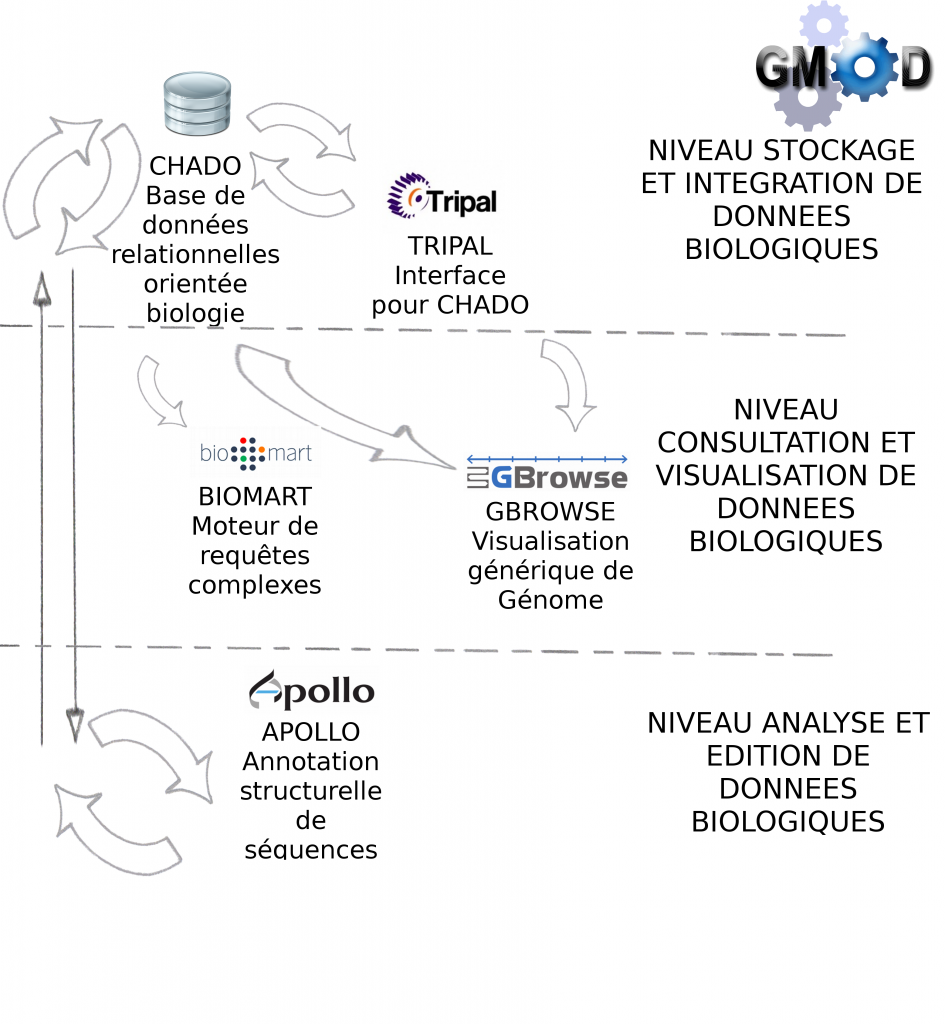
> Le projet GMOD a pour objectif de fournir à l’utilisateur biologiste un ensemble d’outils interconnectés, libre de droit (open-source), générique (pour tous types de données biologiques) et facile d’utilisation (à travers des services Web principalement)
> Les outils GMOD sont développés (et donc installés) par des bioinformaticiens pour une utilisation par des biologistes.
> Certains outils sont indispensables à GMOD, pour la manipulation de données génomiques, c’est le cas des outils Chado et Gbrowse qui sont respectivement les squelettes pour la manipulation et pour la visualisation des données biologiques.
Le schéma ci-dessous décrit les modules et interactions présentés lors de cette journée thématique :
> GBrowse est un outil de visualisation de données biologiques très puissant et certainement l’outil le plus populaire de la suite GMOD
Description détaillée : GMOD-GBROWSE
> Biomart est un outil de recherches avancées (ou requêtes complexes) pour la base de donnée relationnelle Chado
> Apollo est un module pour la correction manuelle d’annotation structurelle
Description détaillée : GMOD-APOLLO
> Tripal est une interface web développée en PHP pour interrogation de Chado
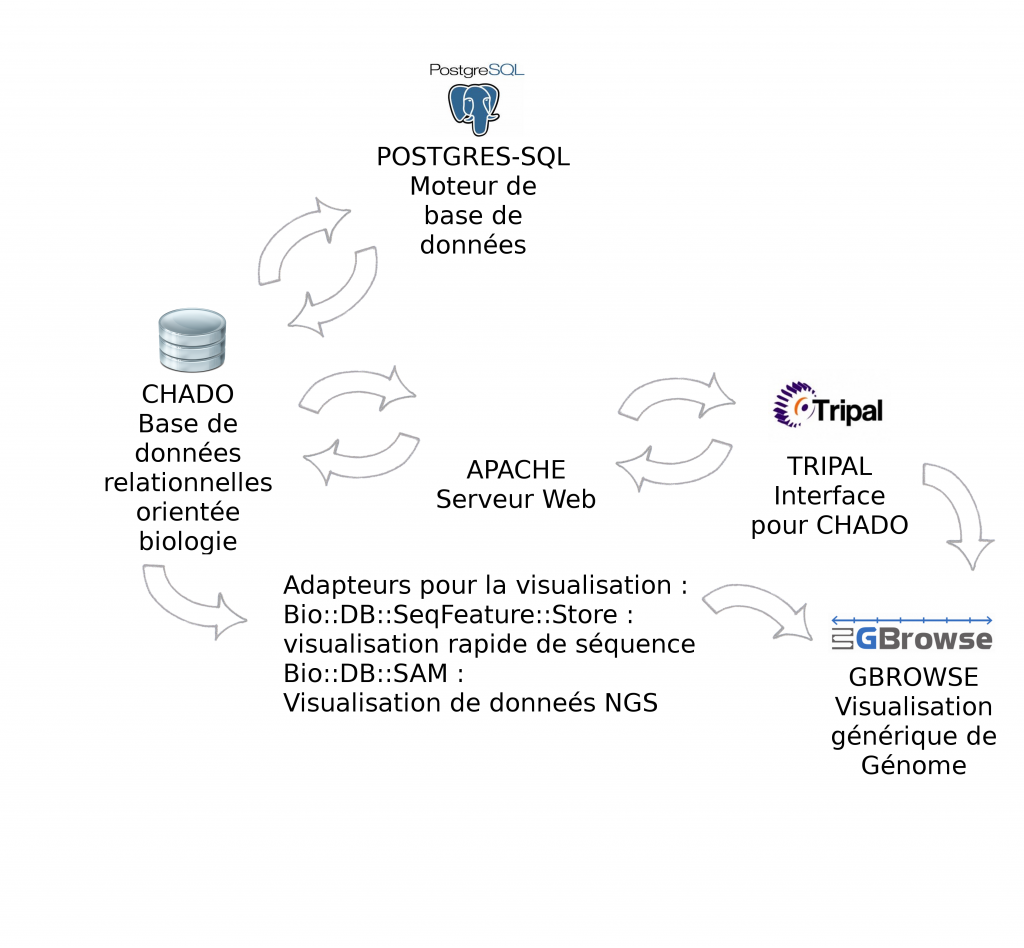
Environnement informatique : CHADO fonctionne par défaut avec Postgres-Sql. Pour l'interfaçage avec Tripal, la solution étant développée avec PHP, il est nécessaire d'installer un serveur Web Apache. Gbrowse peut s'utiliser à partir de l’interface Tripal mais également en "stand-alone". Afin d'accélérer la visualisation des données il est vivement recommandé d'utiliser les adapteurs Bio::DB::*, soit en relais entre Chado et Gbrowse (présentés dans cet article), soit en dupliquant les informations dans les deux bases de données (la visualisation ne se connecte qu'à Bio::DB sans passer par CHADO). Intuitivement, nous privilégions la première solution qui n’entraîne pas de duplication.
Pour conclure, GMOD propose un ensemble de modules pour la standardisation des processus bioinformatique : stockage et manipulation de données biologiques, visualisation et analyses avancées (assemblage, annotation…).
L’utilisation de tels outils (open-source) va dans le bon sens pour le partage scientifique et la standardisation des processus utilisés lors de l’analyse bioinformatique. Cela n’était pas le but de la conférence à laquelle nous avons assistée mais il serait également intéressant de connaître les conditions pour intégrer ces propres outils bioinformatiques en tant que brique GMOD.
GMOD est donc intéressant si :
> vous souhaitez stocker et gérer vos données biologiques
> vous cherchez des solutions d’analyses bioinformatiques déjà développées et robustes
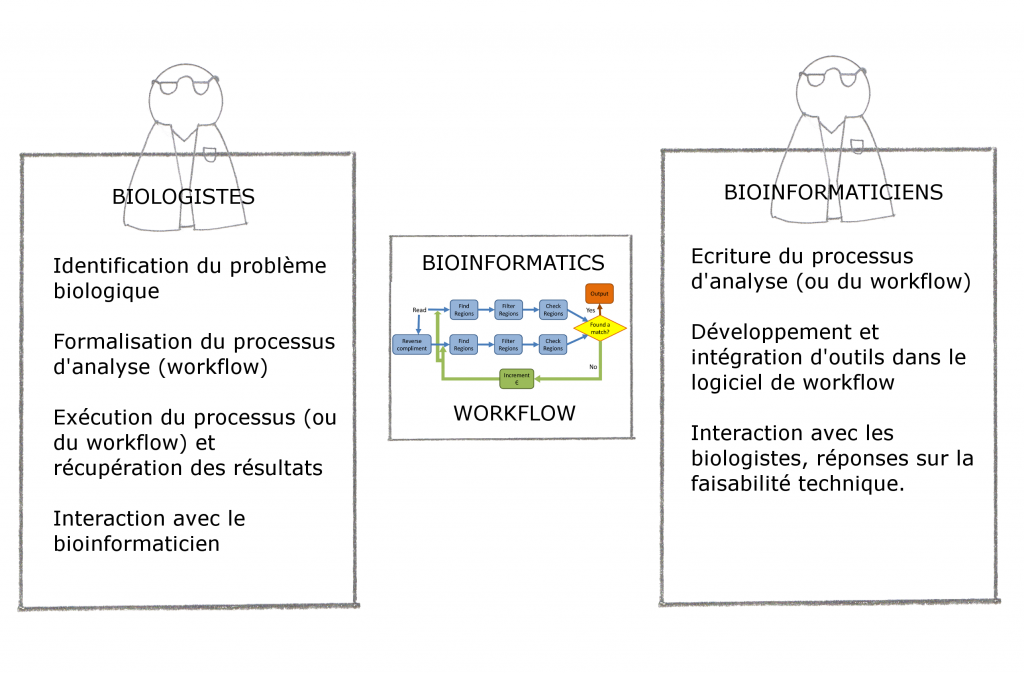
Les biologistes sont les utilisateurs des outils GMOD, en revanche l’installation, l’administration et la formation des utilisateurs ne peuvent échapper à l’intervention, au moins ponctuelle, d’un bioinformaticien.
Si vous projetez d’utiliser ces outils, nous vous conseillons donc dans un premier temps, de regrouper l’ensemble des acteurs, installateurs comme utilisateurs de GMOD, afin de présenter les solutions offertes par l’outil et déterminer les besoins et objectifs pour votre propre utilisation.
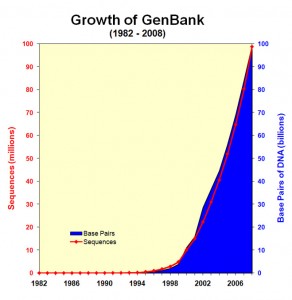
Constat : le séquençage, un générateur fantastique de données
L’explosion de la génération de données génomiques (Figure 1.0) et l’hétérogénéité de ces données entraînent inévitablement un accroissement de l’écart entre les données, les connaissances et l’information que l’on peut en extraire (Figure 1.1).
Le développement d’outils bioinformatiques permet de répondre à l’analyse de données dans l’ensemble des domaines de la biologie, mais le besoin en formalisation (des formats, des processus, des architectures matérielles…) est primordial si l’on veut éviter les problèmes d’incompatibilité et de réutilisation des solutions.
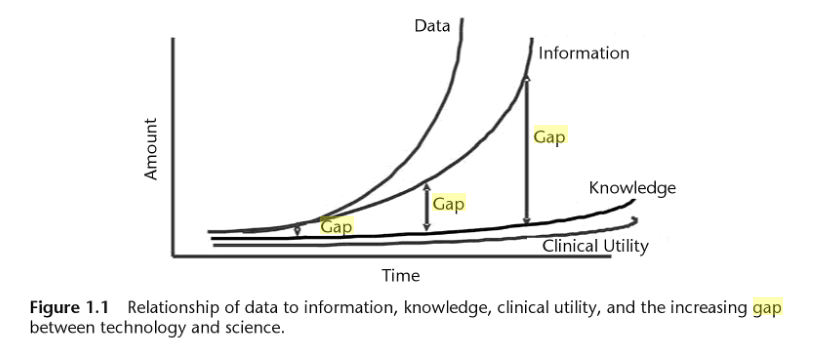
Figure 1.1 : Issu de "Biomedical informatics in translational research" Par Hai Hu,Richard J. Mural,Michael N. Liebman
En conséquence, afin d’obtenir des informations interprétables biologiquement à partir de ces données, la mise en place de processus d’analyse va de pair avec l’utilisation de logiciels à jour, efficaces pour la gestion de données à grande échelle.
En outre, les logiciels doivent aussi simplifier cette gestion de données et fournir des outils d’analyses bioinformatiques accessibles mais aussi reproductibles pour tous types d’utilisateurs – dont les biologistes eux-mêmes. C’est dans ce cadre que s’insèrent l’automatisation de processus (ou « Workflow ») et le développement de « Workflow management system », ou logiciel de Workflow, pour la bioinformatique.
Workflow ou automatisation de processus : définition
Le logiciel de Workflow s’inscrit aussi dans une logique de pérennisation des processus analytiques, qui a pour but de sortir de la logique « projet » (dans le sens ponctuel) en créant des processus d’analyses génériques.
Un logiciel de Workflow est un outil permettant d’exécuter un ensemble de processus de façon automatique. Ces « pipelines » sont très présents en bioinformatique (à défaut d’être tres utilisés) car ils permettent aux chercheurs en biologie d’analyser leurs données (issues de séquencages, génotypages) de façon relativement transparente et (quasiment) sans l’aide d’informaticiens (denrées rares dans la recherche).
Toutefois, il convient de distinguer deux sortes de logiciel de Workflow :
– Les logiciels de Workflow qui permettent aux chercheurs de manipuler leurs données et exécuter leurs analyses sans posséder de connaissances en écriture de scripts ou en bases de données. Les données sont rapatriées au sein du logiciel de Workflow, permettant l’exécution d’un ensemble de tâches, à travers des modules pré-installés. En séquençage, le Workflow permet de convertir des séquences en formats divers, les filtrer ou les assembler… Le logiciel de Workflow ISYS (2001), BioMOBY, Taverna et plus particulièrement Galaxy (qui fera l’objet d’un prochain post ici) entrent dans cette catégorie.
– Les logiciels de Workflow qui assurent un accès direct à des composants (installés sur le serveur) et/ou aux données génomiques sans passer par un rapatriement préalable des données. WildFire, Pegasys ou Ergatis (ce dernier sera décrit dans un prochain post) font partie de cette catégorie. De manière générale ces logiciels de Workflow sont plus difficiles à prendre en main mais sont évidemment plus flexibles.
Pour résumer, quel que soit le logiciel de Workflow utilisé il permet :
– D’automatiser des processus d’analyse (idéalement répétitifs) en les reliant dans un pipeline
– De lancer des analyses sur des architectures matérielles complexes telles des grilles de calculs (voir l’article sur Grisbi) ou des serveurs
– De formaliser le processus d’analyse en vue d’une publication scientifique
Philosophie générale : interaction et exemple de workflow
Pour finir cette introduction, nous vous proposons deux schémas : la place du logiciel Workflow et un exemple concret.
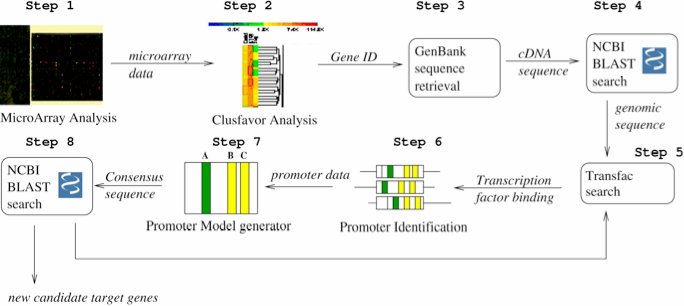
- Figure 1.3 : Exemple de conception d’un Workflow d’identification de promoteurs (extrait de « Accelerating the scientific exploration process with scientific workflows« )
Le PPF Bioinformatique de l’Université Lille 1, l’IFR 142 Médecine Cellulaire et Moléculaire de l’Institut Pasteur de Lille et l’IFR 114 Médecine Prédictive et Recherche Thérapeutique organisent une journée scientifique sur le thème de l’analyse bio-informatique des données produites par les technologies de séquençage à haut débit, dans la lignée de l’édition de 2009. Cette année, les sujets abordés couvriront plus particulièrement les données de type RNA-seq, ChIP-seq,…
Les exposés se tiendront dans l’amphithéatre Butiaux, sur le campus de l’Institut Pasteur de Lille (métro ligne 2, station Grand Palais).
Cette journée sera jumelée au workshop Algorithmique, combinatoire du texte et applications en bio-informatique, qui aura lieu à partir du lendemain au même endroit.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages