Selon une étude : La consommation (modérée) de vin rouge liée à une meilleure santé intestinale…
Une étude du King’s College de Londres tend à montrer que les personnes qui boivent du vin rouge présenteraient une plus grande diversité de microbiote intestinal (un signe de santé intestinale) que les buveurs de vin non rouge.
Dans un article publié le 28 août dans la revue Gastroenterology, une équipe de chercheurs du Department of Twin Research & Genetic Epidemiology au King’s College de Londres a étudié l’effet de la bière, du cidre, du vin rouge, du vin blanc et des spiritueux sur le microbiote intestinal et sur la santé qui en découle chez un groupe de 916 jumelles britanniques. Pourquoi des jumelles? Pour que le fond génétique soit identique, donc la différence de microbiote intestinal entres les jumelles sera en grande partie dû à l’environnement.
Ils ont constaté que le microbiote intestinal des buveurs de vin rouge était plus diversifié que celui des buveurs de vin non rouge. Ceci n’a pas été observé avec la consommation de vin blanc, de bière ou de spiritueux.
La première auteure de l’étude, Caroline Le Roy, du King’s College de Londres, a déclaré : « Bien que nous connaissions depuis longtemps les bienfaits inexpliqués du vin rouge sur la santé cardiaque, cette étude montre qu’une consommation modérée de vin rouge est associée à une plus grande diversité et à un microbiote intestinal plus sain qui explique en partie ses effets bénéfiques sur la santé. »
Le microbiome est la collection de micro-organismes dans un environnement et joue un rôle important dans la santé humaine. Un déséquilibre entre les « bons » microbes et les « mauvais » microbes dans l’intestin peut entraîner des effets néfastes sur la santé, comme un affaiblissement du système immunitaire, un gain de poids ou un taux de cholestérol élevé.
Le microbiote intestinal d’une personne ayant un nombre plus élevé d’espèces bactériennes différentes peut-être considéré comme un marqueur de la santé intestinale.
L’équipe a observé que le microbiote intestinal des consommateurs de vin rouge contenait un plus grand nombre d’espèces bactériennes différentes que celui des non-consommateurs. Ce résultat a également été observé dans trois cohortes différentes au Royaume-Uni, aux États-Unis et aux Pays-Bas. Les auteurs ont tenu compte de facteurs tels que l’âge, le poids, le régime alimentaire régulier et le statut socio-économique des participants et ont continué à voir l’association.
Les auteurs croient que la raison principale de cette association est due aux nombreux polyphénols présents dans le vin rouge. Les polyphénols sont des produits chimiques de défense naturellement présents dans de nombreux fruits et légumes. Ils ont de nombreuses propriétés bénéfiques (y compris des antioxydants) et agissent principalement comme un carburant pour les microbes présents dans notre système.
L’auteur principal, le professeur Tim Spector du King’s College de Londres, a déclaré : « Il s’agit de l’une des plus importantes études jamais réalisées sur les effets du vin rouge sur l’intestin de près de trois mille personnes dans trois pays différents et elle montre que les niveaux élevés de polyphénols dans la peau du raisin pourraient être responsables d’une grande partie des bienfaits controversés pour la santé lorsqu’ils sont utilisés avec modération. »
« Bien que nous ayons observé une association entre la consommation de vin rouge et la diversité du microbiote intestinal, boire du vin rouge rarement, comme une fois toutes les deux semaines, semble suffisant pour observer un effet. Si vous devez choisir une boisson alcoolisée aujourd’hui, c’est le vin rouge qu’il faut choisir, car il semble exercer un effet bénéfique sur vous et sur vos microbes intestinaux, ce qui peut aussi aider à réduire le poids et le risque de maladies cardiaques. Cependant, il est toujours conseillé de consommer de l’alcool avec modération, vous n’avez pas à boire du vin rouge, et vous n’avez pas à commencer à en boire si vous ne buvez pas », a ajouté le Dr Le Roy.
Effectivement, comme dans toute étude, corrélation n’est pas raison! Ainsi et même si la catégorie socio-économique est prise en compte dans l’étude, il pourrait exister d’autres facteurs, non mesurés dans cette étude, qui expliqueraient en partie la bonne santé microbienne des individus. Rappelons que ça n’est pas l’alcool qui est associé avec une meilleure santé intestinale mais d’autres composants du vin rouge (l’hypothèse étant que ce sont les polyphénols), que l’on trouvera aisément dans d’autres aliments (fruits, légumes, noix, cacao…) .
Emplacement de la publication originale :
https://www.sciencedirect.com/science/article/abs/pii/S0016508519412444
L’abus d’alcool est dangereux pour la santé, consommez avec modération
 Pour avoir connu moult galères dans la quête d’un jeu de données (en l’occurrence issu de Ion Torrent) afin de tester des versions de pipelines analytiques ou pour optimiser les paramètres de ceux-ci, nous avons décidé de publier un jeu de données, les résultats en sortie de pipeline et les conclusions statistiques puis biologiques qui en découlent. Avant, dans et après la moulinette bio-informatique.
Pour avoir connu moult galères dans la quête d’un jeu de données (en l’occurrence issu de Ion Torrent) afin de tester des versions de pipelines analytiques ou pour optimiser les paramètres de ceux-ci, nous avons décidé de publier un jeu de données, les résultats en sortie de pipeline et les conclusions statistiques puis biologiques qui en découlent. Avant, dans et après la moulinette bio-informatique.
En espérant, que ceci puisse éviter des galères à d’autres… :
Targeted metagenomic sequencing data of human gut microbiota associated with Blastocystis colonization
Concernant l’exploitation « biologique » de ce jeu de données, l’article publié dans Scientific Data est rattaché à celui-ci :
Colonization with the enteric protozoa Blastocystis is associated with increased diversity of human gut bacterial microbiota
La plate-forme de renseignement de marché de CB Insights analyse des millions de données sur le capital-risque, les start-ups, les brevets, les partenariats pour détecter les opportunités de demain qui dès lors qu’elles sont détectées deviennent celles d’hier. Triste devenir des données hautement périssables !
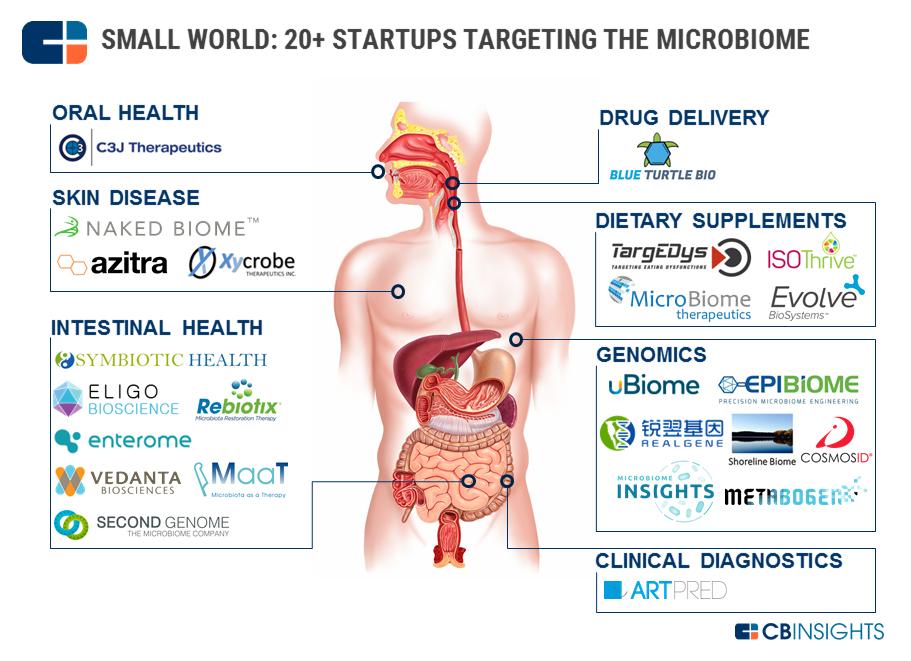
Dans son radar du jour… les start-ups s’attaquant au microbiote à des fins diagnostiques, thérapeutiques, pro- ou pré-biotiques… Beaucoup de ces sociétés telles que Blue Turtle Bio ou Microbiome therapeutics par exemple ont l’ambition de modifier « l’organe » le plus facilement (cela reste à démontrer) malléable de notre corps : le microbiome… Ces entreprises utilisent le séquençage et la métagénomique à haut-débit pour tenter de valider leurs allégations. Il y a fort à parier que nous sommes en train d’assister à une bulle qui continuera de grossir encore puisqu’en matière de métagénomique un grand nombre de projets liés à l’étude des sols vont arriver à maturité d’ici peu… (terroirs pour les vins, microbiotes sols pollués etc.).
Un article du Sydney Morning Herald du 20 novembre 2016 réalise un assez bon plan de communication pour la société Microba.
L’objet : les tribulations gastro-dentaires d’un testeur du service que souhaite bientôt proposer la société investissant le marché de la métagénomique personnelle… Dans la roue des sociétés proposant des services de génomique personnelle voire de génomique récréative, voici venir celles proposant d’appliquer le séquençage haut-débit couplé à de l’analyse automatisée de séquences pour révéler une part de ce qui constitue votre microflore corporelle. Presque triviales, ces analyses métagénomiques sont aujourd’hui à la portée de qui peut les payer. Vous qui ne savez qu’offrir à Noël… peut être une piste de cadeau ! La question que l’on peut légitimement se poser est : à quelles fins analyser votre flore intestinale…? revenons sur l’article du Sydney Morning Herald pour tenter d’y trouver un intérêt ou deux.

L’article commence sur un mode enthousiaste, la technologie permet de révéler une part de notre microbiote… à quoi bon s’en priver ? Le gonzo-journaliste rédacteur de l’article se propose comme cobaye et reçoit des « cotons tiges » de prélèvements qui lui permettent de procéder à l’envoi d’échantillons censés être des prélèvements de sa microflore corporelle -buccale et intestinale. Pas simple l’échantillonnage de la flore intestinale avec un coton tige ! Ainsi que le dit Dr Alena Rinke, porte-parole de Microba : « la recherche dans ce domaine vient d’exploser au cours des cinq ou dix dernières années. Nous constatons qu’il existe un nombre incroyable de liens entre les microbes de notre corps et des états pathologiques« . Ces « liens » peuvent être très divers : diabète, cancer du côlon, maladie inflammatoire de l’intestin, mélanome, polyarthrite rhumatoïde. En guise de commentaire, disons que tout comme corrélation n’est pas raison, liaison n’est pas causalité non plus. L’argument santé finit de motiver le jeune investigateur de sa flore intérieure. Donc le journaliste expédie ses cotons tiges à la société australienne qui réalise une métagénomique ciblée (séquençage haut-débit d’une portion du gène de l’ARN 16S).
Six semaines plus tard, ses résultats lui parviennent. Bilan : beaucoup de Ruminococcus, Blautia, Prevotella copri. Le petit bestiaire personnel qu’il héberge le fascine. Le test de Microba tente de mettre une couche d’interprétation de ces données de séquences mais le rendu semble un peu insatisfaisant pour le jeune intrépide. Il demande au professeur Andrew Holmes (plutôt sceptique sur l’intérêt du service) d’examiner rapidement ses résultats. La première métrique clé qui lui saute aux yeux est le nombre de Proteobacteria présente, dit – il. » En général, une personne en bonne santé a une faible proportion de bactéries dites « pro-inflammatoires ». Il faudrait pour Proteobacteria être inférieur à 5 %, et idéalement moins de 2 %. »
Le journaliste se voit rassuré avec une proportion de Proteobacteria, inférieure à 1 %.
Son microbiote est légèrement plus diversifié que la moyenne – c’est un bon point. Cela signifie qu’il est en bonne santé avec/grâce à un régime alimentaire équilibré et lui-même diversifié. Dans l’ensemble, le test de Microba conclut que sa flore intestinale suggère qu’il est en «excellente» santé avec un IMC de 20,7. Basé sur l’information de son microbiote, le test prédit qu’il a 28 ans (plutôt pas mal puisqu’il en accuse 26, avec un IMC de 21,9).
L’enthousiasme retombe un peu quand le cobaye-journaliste promoteur du service de Microba apprend qu’il a des proportions de Dialister en quantités supérieures à la moyenne. Dialister est un bon prédicteur de la gingivite. Il va donc se programmer une visite chez le dentiste ce qui fait toujours plaisir !
Comme l’a fait remarquer le professeur Holmes, les résultats reçu par le journaliste comporte beaucoup de données intéressantes – mais pas beaucoup d’idées utiles. A l’instar de ce que l’on a pu constater pour d’autres fournisseurs de « génomique personnelle », chaque échantillon reçu par Microba permet d’améliorer sa méthode de prédiction d’éléments interprétés du microbiote. Le service sera lancé au public au début d’année 2017.
Moralité : à moins d’avoir un Holmes sous la main, ces sociétés devront fournir à leurs clients des résultats automatiquement pré-interprétés pour que ces derniers puissent avoir un outil influençant leur mode de vie ou leur gestion de planning-dentiste. En effet, l’inventaire à la Prévert de ce que l’on a dans le ventre n’a que peu de sens et nécessiterait d’être remis en perspective. Cette liste, ces métriques devraient être rattachées à la singularité du client du service pour réellement pouvoir lui être utiles. Un modèle prédictif basé sur une méthode d’apprentissage automatique ne peut suffire ! On comprend que plus Microba va séquencer, plus précis sera son modèle mais son développement futur passera par une meilleure intégration d’un résultat interprétable par le commun des mortels.
Lille, le 19 décembre 2012
 Le PPF Bioinformatique de l’Université Lille 1 organise une journée scientifique sur le thème de la métagénomique et des méthodes d’analyse bio-informatique et biostatistique associées : assemblage des génomes, classification et quantification des espèces, assignation taxonomique, reconstruction phylogénétique… Cette réunion s’inscrit dans la lignée des éditions 2009 et 2011 sur l’analyse des données NGS.
Le PPF Bioinformatique de l’Université Lille 1 organise une journée scientifique sur le thème de la métagénomique et des méthodes d’analyse bio-informatique et biostatistique associées : assemblage des génomes, classification et quantification des espèces, assignation taxonomique, reconstruction phylogénétique… Cette réunion s’inscrit dans la lignée des éditions 2009 et 2011 sur l’analyse des données NGS.
Les exposés se tiendront dans l’amphithéatre Butiaux, sur le campus de l’Institut Pasteur de Lille (métro ligne 2, station Grand Palais).
Programme
- 10h00 : café d’accueil
- 10h20 : présentation de la journée (David Hot)
- 10h30-11h30 : Nicolas Pons, INRA Jouy-en-Josas : The potential of the new science of metagenomics in curing human complex diseases
- 11h30-12h20 : Guy Perrière, LBBE, Lyon
- 12h20-13h45 : déjeuner
- 13h45-14h35 : Frédéric Mahé, TU Kaiserslautern : TARA OCEANS, exploring worldwide marine biodiversity
- 14h35-15h25 : Mihai Pop, University of Maryland : Towards Holistic Analyses of Metagenomic Data
- 15h25-15h40 : pause
- 15h40-16h30: Jean-Jacques Daudin, AgroParisTech : Bioinformatics and Statistical challenges from the analysis of NGS-Metagenomics experiments
- 16h30-17h20 : Sara Vieira-Silva, Vlaams Instituut voor Biotechnologie
Inscriptions
La participation est gratuite, mais nous vous demandons de vous inscrire pour permettre l’organisation des pauses et du déjeuner.
Merci d’envoyer un mail à mohcen.benmounah@lifl.fr avec les informations suivantes pour le 7 décembre.
--------------------------------------------------
19 decembre 2012 - journee Metagenomique
Nom:
Prenom:
Laboratoire :
Dejeuner : oui/non
--------------------------------------------------
Vous retrouverez l’intégralité de ces informations sur le site du ppf : http://www.lifl.fr/~touzet/PPF/metagenomique12.html
 La paléogénétique, la science qui permet de remonter le temps, a trouvé sous la forme de séquenceurs haut-débit, sa DeLorean… en route vers l’Ardèche, dans l’antre de la grotte Chauvet. La grotte Chauvet, inventée en 1994, comporte 420 représentations d’animaux d’une incroyable diversité technique. C’est en partie les déductions menées à partir de l’étude de cette grotte, que l’on a remis en cause l’idée d’un art (pariétal) évoluant lentement et de manière linéaire. L’art peut être vu comme une suite d’apogées et de déclins, un concept s’opposant à l’idée d’un art co-évoluant avec le progrès technique et la diffusion du savoir-faire et des connaissances.
La paléogénétique, la science qui permet de remonter le temps, a trouvé sous la forme de séquenceurs haut-débit, sa DeLorean… en route vers l’Ardèche, dans l’antre de la grotte Chauvet. La grotte Chauvet, inventée en 1994, comporte 420 représentations d’animaux d’une incroyable diversité technique. C’est en partie les déductions menées à partir de l’étude de cette grotte, que l’on a remis en cause l’idée d’un art (pariétal) évoluant lentement et de manière linéaire. L’art peut être vu comme une suite d’apogées et de déclins, un concept s’opposant à l’idée d’un art co-évoluant avec le progrès technique et la diffusion du savoir-faire et des connaissances.
Les œuvres de la grotte Chauvet démontrent q’au début du Paléolithique supérieur, des artistes étaient capables d’une abstraction intellectuelle (une certaine conceptualisation) pour préparer la paroi calcaire et penser le dessin, tout en maitrisant des techniques complexes (estompes,perspective…). L’observation des traces humaines, à la grotte Chauvet comme dans beaucoup de lieux montrant une activité préhistorique, est accompagnée d’analyses de mesures physico-chimiques. Ainsi, avec d’autres méthodes, la datation radiométrique au carbone 14 a été employée pour permettre une estimation de l’âge absolu d’un échantillon organique… la grotte ardéchoise aurait hébergé les artistes du paléolithique autour de 31 000 ans avant notre ère. Assez nouvellement les techniques de génomique trouvent un champ d’application au niveau de la l’histoire des populations et viennent compléter les autres types d’observations pour nous reconstituer ce que le temps a détruit ou transformé.
Le carbone 14 génomique pourrait (en quelque sorte) être constitué par l’ADN mitochondrial. L’ADNmt est ici pris comme témoin (plus ou moins stable) des espèces disparues et sert de base aux études phylogénétiques, loin de Jurassic Park mais néanmoins enthousiasmant… mais est ce vraiment aussi simple ?
En effet, de par sa stabilité présumée au fil de l’évolution, l’ADNmt est devenu une cible de choix pour l’étude de la diversité des populations : a priori, les gènes déterminant la fonction respiratoire de la cellule sont moins la cible d’adaptations fréquentes à des changements environnementaux que, par exemple, ceux de l’immunité, situés, eux, dans l’ADN nucléaire. Je vous conseille vivement, à ce sujet, la lecture de l’article suivant, remettant en cause ce paradigme (cf. CNRS – le journal – mars 2007).
Mais revenons à l’utilisation du séquençage haut-débit pour remonter le temps. Le temps a fait son œuvre quant à la première étape de la préparation de la librairie, la fragmentation. « Dans les régions tempérées, ce sont les échantillons provenant de grottes qui sont souvent les mieux conservés (cf. figure ci-dessous). Dans les grottes, la température reste très stable, autour d’une valeur comprise entre 10 et 15°C. De plus, les grottes se creusent dans des environnements karstiques, riches en calcium et de pH neutre ou basique, qui sont des conditions propices à la conservation de la matière organique et de l’ADN en particulier. » – Extrait de la thèse de Céline Bon (CEA – page 50 – 25 juin 2012).
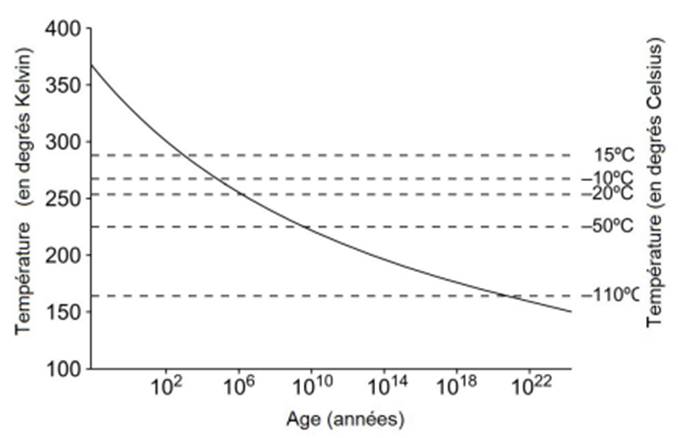
Temps de survie d’un ADN de taille supérieure à 100 pb
Le séquençage haut-débit devient réellement d’un grand intérêt dans l’étude du métagénome des coprolithes et des ossements retrouvés dans ces grottes. La capture d’écran ci-dessous vous mènera en cliquant dessus, sur une partie du site du CEA où une vidéo simple et efficace, reprend les propos de Jean-Marc Elalouf. Ce dernier aborde l’exceptionnelle conservation des ossements qui a permis aux biologistes d’en analyser l’ADN et ainsi de faire de nouvelles avancées sur l’étude des espèces éteintes et la phylogénie des ours des cavernes.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages