L’évolution des technologies de biologie moléculaire combinée à des systèmes de détection toujours plus sensibles, des techniques d’amplification accessibles dans des systèmes intégrés ont rendu possible l’analyse du transcriptome d’une cellule isolée. Dans de nombreux domaines tels que la cancérologie, la biologie des cellules souches, l’ingénierie des tissus, le signal moyen peut cacher la pertinence du signal noyé dans la masse des cellules d’intérêt.
Défier la loi de la moyenne, en tenant compte de l’hétérogénéité cellulaire pour tenter de capter ce signal qui échappe aux systèmes n’analysant que le mélange de cellules, est le credo du système proposé par Fluidigm : le C1™ Single-Cell Auto Prep System.
Isoler des cellules pour extraire leur ARN peut être une tâche technique ardue consommatrice de temps et très sensible aux contaminations.
La société américaine, Fluidigm propose donc un système plutôt simple permettant de capturer quelques cellules pour en extraire leurs acides nucléiques. Le consommable est composé d’une plaque microfluidique comportant 96 pièges à cellules enchaînés. La multiplicité de ces pièges permet d’accroître les chances de capturer une cellule dans l’état escompté. Une fois la phase de capture accomplie, les divers pièges sont individualisés. Un système de by-pass permet de « pousser » chacune des cellules piégées dans une succession de « microréacteurs » isolés enchaînant phases de lyse, de transcription inverse, d’amplification des ARNs (WTA) et de collecte des acides nucléiques amplifiés.
Le schéma ci-dessous reprend les 2 phases principales permettant d’aboutir à l’analyse du transcriptome de cellules isolées :
(i) l’amplification d’ARN de cellules isolées
(ii) l’analyse de ces ARNs
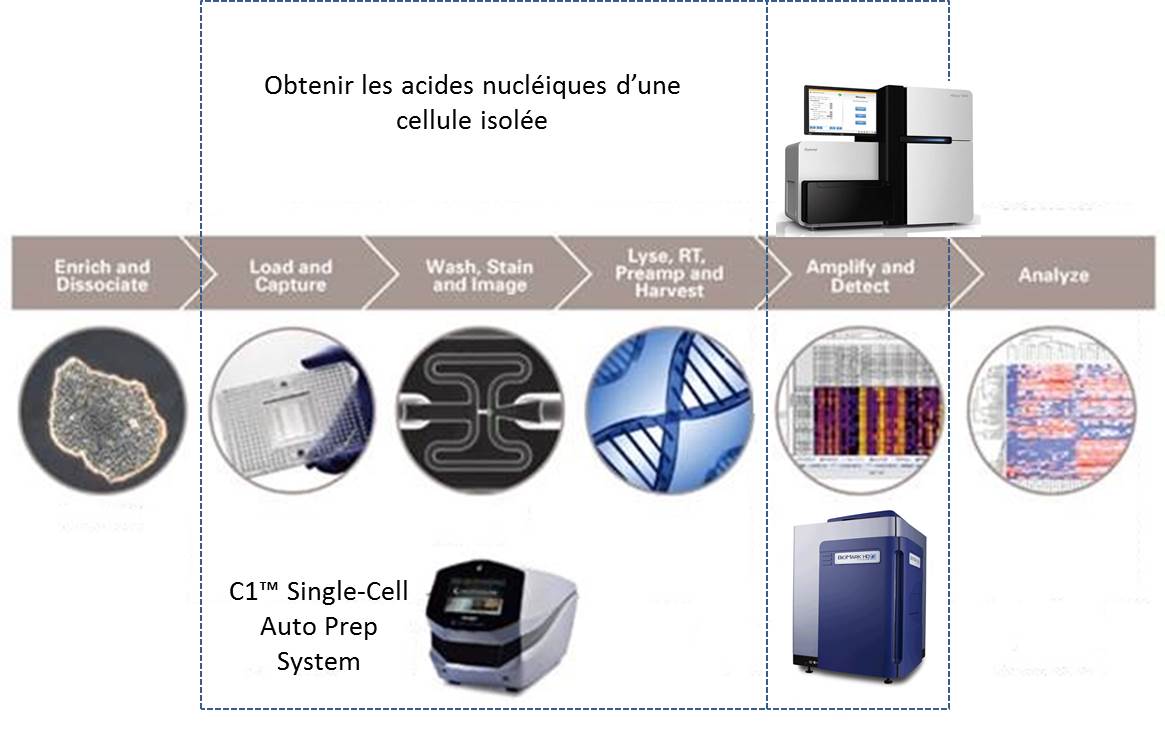
Ainsi que le montre la vidéo promotionnelle ci-dessous le système est compact et se veut simple d’utilisation. Il va de soi, malgré tout, que pour optimiser vos chances de capturer un signal d’intérêt un marquage préalable cellules vivantes/mortes peut s’imposer. Ceci impose aussi d’observer la plaque microfluidique au microscope à fluorescence (en se hâtant quelque peu). Bien évidemment l’adjonction de marqueurs discriminants (anticorps couplés à un agent fluorescent) est la bienvenue.
Cette dernière vidéo aborde le « workflow » aboutissant à l’analyse du transcriptome monocellulaire par Biomark, il va de soi qu’il s’agit là d’une « suggestion de présentation » puisqu’il est possible d’analyser le transcriptome amplifié par le système C1™ Single-Cell Auto Prep System avec un grand nombre de systèmes de qPCR ou de séquençage haut-débit.
http://youtu.be/TF4NJRE4Xg4
L’analyse des biomarqueurs des cellules tumorales circulantes devient un élément majeur de la médecine personnalisée (lire L’enjeu des cellules tumorales circulantes). L’article d’Ashley A. Powell et al. (Single Cell Profiling of Circulating Tumor Cells: Transcriptional Heterogeneity and Diversity from Breast Cancer Cell Lines (Plos One, 2102)) montre l’application de ce système au niveau de l’analyse de « biopsie liquide« .
L’outil proposé par Fluidigm trouve d’ores et déjà des applications cliniques très concrètes : l’analyse de biomarqueurs des cellules tumorales circulantes à visée pronostique.
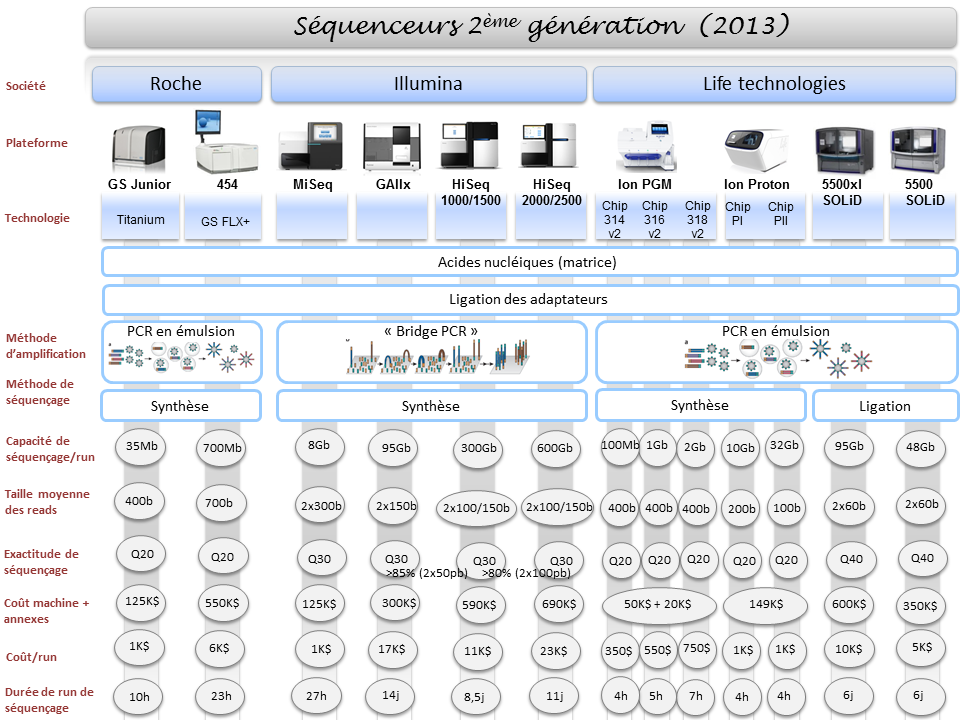
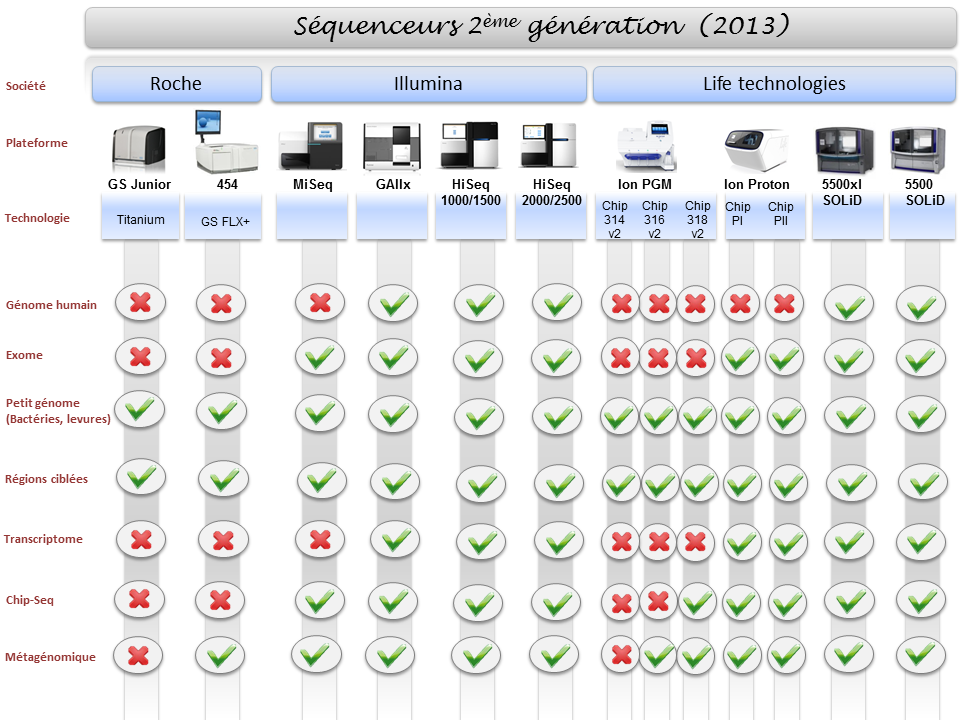
La diversité du parc technologique des séquenceurs de deuxième génération n’est plus une surprise pour personne. Ceci étant, il devenait indispensable de mettre à jour l’ensemble des informations postées sur ce site, il y a exactement deux ans (2011), faisant un état de l’art des différentes caractéristiques technologiques des séquenceurs, ainsi que les possibles applications biologiques associées.
Pour ne mentionner que les trois plus gros fournisseurs du marché, les sociétés Roche, Illumina et Life Technologies n’ont cessé de faire évoluer leur gamme, tant sur le plan des équipements que sur le plan des capacités de séquençage.
Par voie de conséquence, ce survol est l’occasion de refaire le point sur les technologies appropriées selon l’application biologique recherchée. A noter que le Ion proton, dernier en date sur le marché des séquenceurs de deuxième génération disposera au cours de l’année 2014 d’une puce « PIII » permettant de générer environ 64Gb. Cette capacité de séquençage permettra à Ion torrent de se positionner sur le séquençage de génome humain à partir d’un séquenceur de paillasse et accèdera ainsi à la gamme complète des applications citées ci-dessous.
Bertrand Jordan a eu son doctorat en physique nucléaire à 1965 et a très bien fait de se convertir à la biologie moléculaire. Entre autres choses, il a réalisé en 1982 l’isolement du premier gène d’histocompatibilité humain, et en 2000, il a fondé la Génopole de Marseille. Bertrand Jordan est membre de l’Organisation européenne de biologie moléculaire (EMBO) ainsi que de l’organisation internationale HUGO (Human Genome Organisation).
Mais surtout, et c’est ce qui nous intéresse ici, l’auteur de plus de 150 publications scientifiques fait œuvre de vulgarisation et offre depuis plus de dix ans, des chroniques génomiques publiées au sein de la revue Médecine / Sciences. Ces chroniques sont accessibles gratuitement (vous pouvez y accéder directement sur le site de www.medecinesciences.org ou en cliquant sur l’image ci dessus en haut à gauche).
On peut vous conseiller de lire le point de vue de Bertrand Jordan sur les séquenceurs haut-débit dans sa chronique de mars 2010 intitulée : le boom des séquenceurs nouvelle génération, sans oublier le point de vue partagé (parfois longtemps après lui) par un nombre croissant de chercheurs sur les GWAS, dans sa chronique de mai 2009 : le déclin de l’empire des GWAS. C’est là tout l’intérêt de ces chroniques qui, bénéficiant d’un propos clair, simple, construit, argumenté et rare, permettent de prendre un peu de recul sur les applications d’une science qui n’ont pas toujours tenu leurs promesses. Ces chroniques peuvent être l’occasion de faire tomber le masque, de dégonfler l’air de ces vessies qui se prennent parfois pour des lanternes.
Bertrand Jordan, chroniqueur mais aussi auteur de quelques livres aux titres évocateurs, parfois mémorablement provocateurs :
Voyage autour du Génome : le tour du monde en 80 labos. Editions Inserm/John Libbey, Paris, 1993
Voyage au pays des gènes. Editions. Les Belles Lettres/ Inserm, Paris, 1995
Génétique et génome, la fin de l’innocence. Editions Flammarion, Paris, 1996
Les imposteurs de la génétique. Editions du Seuil, Paris, 2000 (Prix Roberval Grand Public 2000)
Le chant d’amour des concombres de mer. Editions du Seuil, Paris, 2002
Les marchands de clones. Editions du Seuil, Paris, 2003
Chroniques d’une séquence annoncée. Editions EDK, Paris, 2003
Le clonage, fantasmes et réalité. Essentiel Milan, Editions Milan, 2004
Thérapie génique : espoir ou illusion ? Editions Odile Jacob, Paris, 2007
L’humanité au pluriel : la génétique et les questions de race. Le Seuil, collection Sciences ouvertes, Paris, 2008
Autisme, le gène introuvable : Le Seuil, Paris, 2012
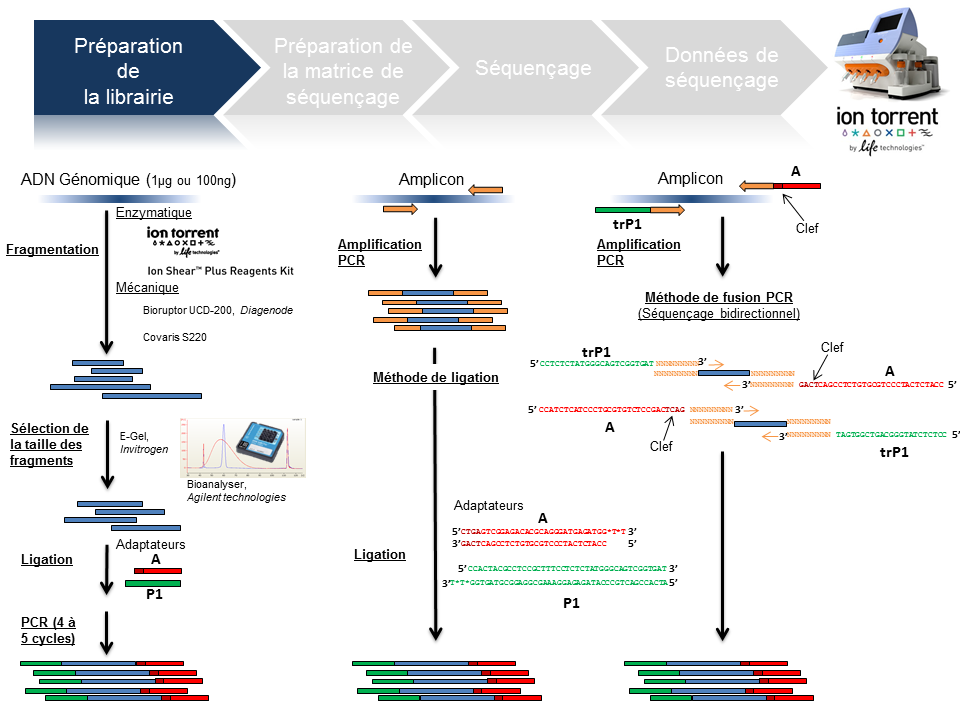
Ce poste a vocation de passer en revue les différentes étapes de la technologie de séquençage à haut-débit du PGM Ion Torrent, depuis la préparation de la librairie jusqu’à l’obtention des données brutes. Ce survol technologique permet de rassembler un maximum d’explications techniques et de termes clefs associés à cette technologie. L’intérêt est de répondre essentiellement à une attente de néophytes ou futurs utilisateurs du PGM friands de retours d’expériences.
L’arbre de décision Ion Torrent s’étant considérablement développé, seuls les axes « Whole genome » et « Amplicon » serviront de support à l’ensemble de la présentation.
Préparation de la librairie
La finalité d’une préparation de librairie pour le PGM Ion Torrent est de lier aux fragments d’ADN à séquencer le couple d’adaptateurs A et P1. La taille médiane des fragments est variable et définie selon la chimie employée: 100, 200 , 300 ou 400 bases (Cf tableau ci-dessous).
Le traitement d’un échantillon d’ADN génomique débute par une étape de fragmentation mécanique ou enzymatique. Cette dernière présente l’avantage d’être considérablement plus rapide.
En amplicon-seq, la méthode pour flanquer les adaptateurs est double, par ligation ou par fusion PCR.
Par ailleurs, il est envisageable de traiter plusieurs échantillons en parallèle en utilisant des adaptateurs avec code barre ( En standard chez Life technologies: Au nombre de 96 pour les échantillons ADN et 16 pour les échantillons ARN).
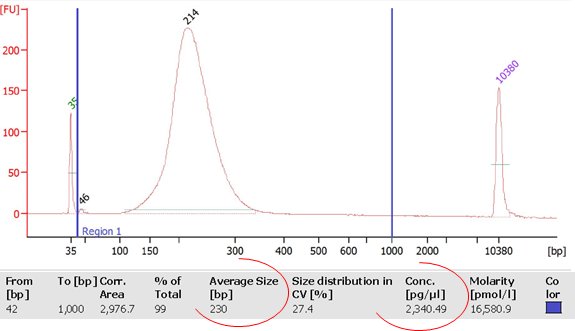
La librairie est monitorée sur puce DNA High sensitivity (Bioanalyzer, Agilent) avec comme objectifs de calculer sa concentration et d’identifier la taille moyenne des fragments qui la composent. Ces valeurs permettront ainsi de déterminer la concentration molaire de la libairie et d’y appliquer le facteur de dilution nécessaire pour favoriser le ratio idéal 1/1 (Fragment ADN de la librairie/Ion Sphere Particle) pour l’étape suivante de PCR en émulsion.
Préparation de la matrice de séquençage
Cette étape automatisée permet l’amplification clonale (OneTouch2) suivi d’un enrichissement en « Ion Sphere Particles » (ISPs) à la surface desquelles un fragment de librairie est amplifié (OneTouch ES).
L’amplification clonale est réalisée au cours de la PCR en émulsion (emPCR) et contribuera à atteindre un seuil de détection du signal nécessaire et suffisant au moment du séquençage. Malgré une optimisation du ratio 1/1 (ISP / Fragment ADN), plusieurs configurations de microréacteurs sont envisageables. Seule la configuration de monoclonalité est souhaitée car elle seule, est source de données de séquençage. Les autres configurations généreront des données qui seront filtrées lors de la primo-analyse par la « Torrent suite ».
L’amorce ePCR-A couplée à la biotine permettra l’enrichissement ultérieure par un système de capture sur billes liées à la streptavidine.
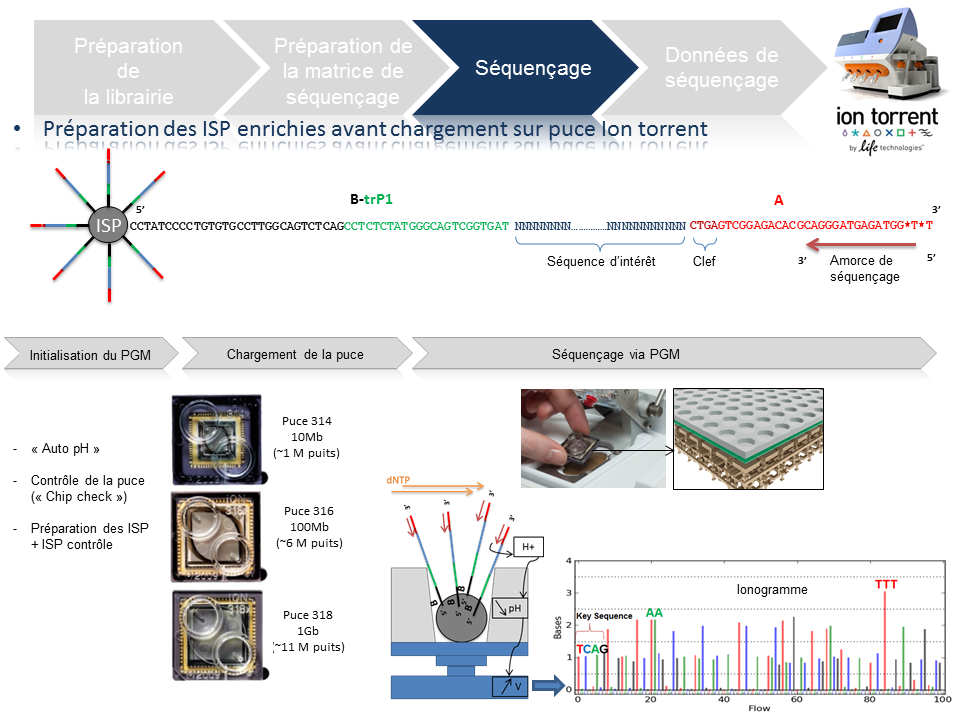
Séquençage
En amont de l’étape de séquençage, une initialisation du PGM est requise et permet notamment une homogénéisation des valeurs de pH ~ 7,8 au sein des différents réactifs de l’appareil (« Auto pH« ).
La matrice de séquençage couplée aux amorces de séquençage et à la polymérase est chargée sur la puce Ion Torrent selon un protocole bien spécifique. Les puces se déclinent selon 3 capacités de séquençage ( Chip 314 >10Mb, Chip 316 >100Mb, Chip 318 >1Gb). A noter qu’une version « v2 » pour chacune des puces précitées existe et est indispensable pour toute application de séquençage nécessitant la chimie 400. Le séquençage multiparallélisé revient donc au décryptage simultané des fragments ADN couplés aux ISP. A chaque polymérisation de nucléotides non modifiés, la libération d’ ions H+ entraine une variation de pH, elle même détectée au niveau de la couche mince (technologie des semi-conducteurs) située au fond de chaque puits. L’ensemble des données brutes générées est transcrits sous forme de ionogrammes.
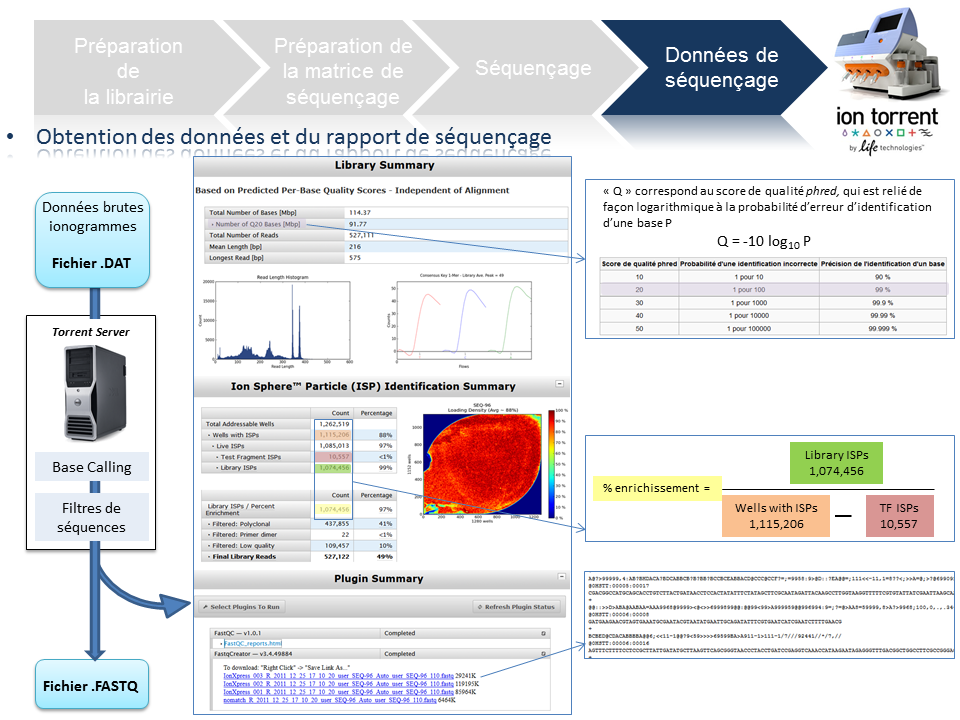
Données de séquençage
A l’issue du « run » de séquençage, le fichier .DAT regroupe l’ensemble des données brutes (ionogrammes). Ces fichiers sont transférés du PGM vers le Torrent Server. L’algorithme de « base calling » permet la conversion des données sous forme de lettres en séquences (A,T,C,G) formant le read (séquence au format fasta) associé à un score de qualité (Phred Score codé en ASCII), les deux types de données étant associés dans un fichier .FASTQ (qui tend à devenir le format de référence).
Un prétraitement est également appliqué sur la base des reads générés et qui équivaut au nombre d’ISPs vivantes ou « Live ISPs » (On parle d’ISPs vivantes pour les ISPs associées à la clef):
– trimming : élimination des adaptateurs et/ou portions de reads de mauvaise qualité
– filtres : élimination des « reads » de petites tailles, de mauvaise qualité, des polyclonaux
L’ensemble de ces informations est repris au travers du « report » généré à l’issue du séquençage et du prétraitement. Y sont également renseignés, le nombre de reads générés ainsi que leur taille moyenne.
 QIAGEN est un fournisseur très connu pour faire dans le haut de gamme du réactif de biologie moléculaire. Très connu pour ses colonnes de silice et sa polymérase plutôt robuste (HotStart Taq Plus). QIAGEN s’est lancé, à coups de rachats dans la qPCR et la robotique de pipettage, afin de mettre des instruments à son catalogue, instruments qui utiliseront ses consommables (on n’est jamais mieux servi que par soi-même). Ainsi la société allemande s’est payée Corbett Life Science, en 2008, 135 millions de dollars en cash. La société australienne a permis à QIAGEN d’enfin mettre à son catalogue une qPCR (à la technologie tout à fait singulière) et une robotique repeinte en bleu et gris.
QIAGEN est un fournisseur très connu pour faire dans le haut de gamme du réactif de biologie moléculaire. Très connu pour ses colonnes de silice et sa polymérase plutôt robuste (HotStart Taq Plus). QIAGEN s’est lancé, à coups de rachats dans la qPCR et la robotique de pipettage, afin de mettre des instruments à son catalogue, instruments qui utiliseront ses consommables (on n’est jamais mieux servi que par soi-même). Ainsi la société allemande s’est payée Corbett Life Science, en 2008, 135 millions de dollars en cash. La société australienne a permis à QIAGEN d’enfin mettre à son catalogue une qPCR (à la technologie tout à fait singulière) et une robotique repeinte en bleu et gris.
QIAGEN arrive sur le marché, mais encore une fois un peu tard, des séquenceurs haut débit de deuxième génération. La société de Francfort a donc racheté Intelligent Bio-Systems (IBS) dont la technologie est sous licence de Jingyue Ju (Université de Columbia). Cette licence propose une méthode permettant de diluer les molécules marquées (les terminateurs réversibles, ces mêmes molécules qui sont utilisées par la technologie d’Illumina dans Hiseq et Miseq). Ainsi les coûts des consommables s’en trouvent diminués.
Le Mini20 comme se nomme la plateforme développée par IBS, a tout d’une grande. Les coûts annoncés, il y a un an de cela, avoisinaient les 300 $ / Gbase (à la même date sur Miseq, les 600 $ / Gbase étaient dépassés). La technologie rachetée par QIAGEN permet d’utiliser « 20 flow-cells » , adressables individuellement pour un débit de 20 M reads / flow cell, débit total de 80 Gbases. Le séquenceur était annoncé à un prix de 120 k$ venant clairement marcher sur les plates bandes d’un Miseq. A n’en pas douter, l’arrivée d’un poids lourd comme QIAGEN sur le marché du séquençage haut-débit de 2ème génération devrait dynamiser et quelque peu perturber le ménage à deux de Life Technologies et Illumina. Certes, QIAGEN, avec ses solutions de préparations d’échantillons (chimie et instruments de pipettage), serait un nouvel acteur proposant des solutions complètes (et notamment en bio-informatique grâce à un partenariat avec SAP).
L’ambition de QIAGEN semble être de fournir une solution complète de l’échantillon à l’analyse des séquences que leur Mini-20 aura généré. Il faut juste espérer que les perspectives de croissance sur le marché des séquenceurs de 2ème génération ne soient pas anéanties par l’arrivée d’un séquenceur de 3ème génération à coût raisonnable. QIAGEN arrive tard dans la course mais avec une offre « tout inclus » qui serait susceptible de faire mouche pour qui n’a pas accès à un plateau technique de biologie moléculaire ou encore à des compétences en bio-informatique.
 Après de longues péripéties où il s’agissait de ralentir la progression d’une molécule d’ADN à travers un nanopore, après quelques investissements (de la part d’Illumina, essentiellement) il semblerait que la société, Oxford Nanopore s’apprête à vendre les deux produits dont elle fait la promotion depuis plusieurs mois. Ces deux produits n’existaient alors que dans les couloirs de l’AGBT (en 2012, parce que cette année il semble qu’Oxford Nanopore fasse profil bas à l’AGBT2013… à moins que…) et sur le site internet de la société encore un peu britannique.
Après de longues péripéties où il s’agissait de ralentir la progression d’une molécule d’ADN à travers un nanopore, après quelques investissements (de la part d’Illumina, essentiellement) il semblerait que la société, Oxford Nanopore s’apprête à vendre les deux produits dont elle fait la promotion depuis plusieurs mois. Ces deux produits n’existaient alors que dans les couloirs de l’AGBT (en 2012, parce que cette année il semble qu’Oxford Nanopore fasse profil bas à l’AGBT2013… à moins que…) et sur le site internet de la société encore un peu britannique.
Quelques médias d’outre-Manche évoquent cette fameuse technologie de séquençage en l’affublant du qualificatif de « future grande invention britannique » : lire à ce sujet la page web de « The Raconter » : Britain greatest inventions ». Cette invention est associée à la seule médecine personnalisée comme pour envisager le futur marché du séquençage haut-débit.
Les voyants semblent donc au vert pour Oxford Nanopore. Pour préparer le terrain, sort en ce début d’année, un article de Nature Methods : « disruptive nanopores« . Un titre qui fait écho à celui de Forbes (février 2012) repris dans notre image ci-dessus.
Nicole Rusk, rédactrice en chef à Nature Methods, vient avancer les principales caractéristiques du futur produit :
– des reads entre 10 et 100 kb
– des taux d’erreurs entre 1 et 4 %
– la possibilité de connaître les bases méthylées
– la possibilité de séquencer directement l’ARN
– la méthode est non destructive

La technologie de séquençage de 3ème génération par nanopores promet de révolutionner plusieurs applications à commencer par le séquençage de novo en laissant peu d’espoir à la technologie de Pacific Biosciences. Elle devrait remplacer PacBio dans les stratégies de séquençage hybride (qui consiste « à coupler » un séquenceur de 2ème génération permettant d’être très profond et d’une technologie de 3ème génération permettant de générer des reads très longs ce qui permet au final un assemblage de meilleure qualité).
Après avoir engendré de la curiosité, de l’impatience, puis déçu avec une arrivée sans cesse repoussée, il semble qu’Oxford Nanopore doive prouver de l’efficacité de sa technologie. Ainsi, la société britannique a annoncé le 8 janvier 2013, une série d’accords avec plusieurs institutions telles que l’ Université de l’Illinois, l’Université Brown, l’Université de Stanford , l’Université de Boston, de Cambridge et de Southampton. Oxford Nanopore prend son temps ou rencontre des difficultés avec son exonucléase. Malgré ses dizaines de brevets, pour conserver sa crédibilité la société a dû communiquer pour convaincre de l’efficience de sa technologie en minimisant les difficultés de développements, en alimentant les tuyaux de communication avec des séquenceurs sorti de palettes graphiques loin d’être finalisés.
Ce retard de lancement s’apparente t’il à un gage de sérieux ou est-il la preuve que le séquençage par nanopores rencontre de grosses, très grosses difficultés de développement ? Réponse en 2013.
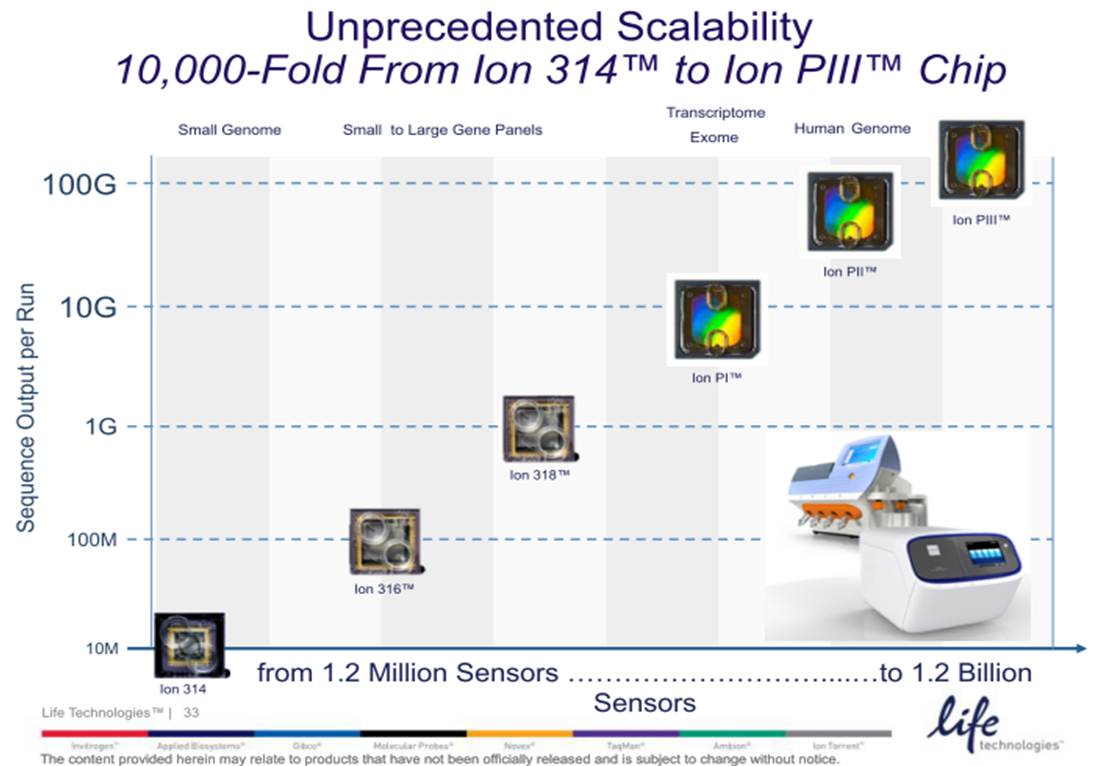 Le mois de décembre 2012 a connu le lancement du kit 400 bases (
Le mois de décembre 2012 a connu le lancement du kit 400 bases (
Ion PGM™ Template OT2 400 Kit) qui est principalement attendu pour les applications de séquençage de novo et de séquençage d’amplicons ciblant le 16S, 18S ou encore ITS2 (respectivement pour l’étude de la diversité bactérienne, eucaryote ou fongique) pour les études métagénomiques. Comme vous pouvez le constater cette application est uniquement supportée par le One Touch 2 devenu nécessaire pour accéder aux reads de 400 bases (c’est en partie pour cette raison que notre plateforme PEGASE-Biosciences a décidé de se doter de cette automate).
L’autre nouveauté est portée par les consommables de séquençage associés au Ion Proton (le grand frère du PGM). En effet, si la Puce Ion PIII est encore virtuelle, la PI et PII, permettent de délivrer : 10 Gb et 70 millions de reads filtrés environ pour la première, contre plus de 280 de millions de reads et plus de 70 Gb pour la seconde (de quoi séquencer un génome humain à 20 X).
Du côté de Life Technologies et de sa technologie Ion Torrent, sans aucun doute, la principale nouveauté de l’année 2013 touche l’amplification clonale (phase où, à partir de la librairie préparée, est réalisée la matrice de séquençage fixée de manière covalente sur des billes qui se logeront dans les puits présents sur la puce). Ainsi, la fameuse (et pour certains la fâcheuse) phase de PCR en émulsion trouvera une alternative avec la chimie isothermique dite « avalanche » (dont vous pouvez voir l’aperçu d’un run ci-dessous). A en croire Life Technologies, cette chimie ouvre la voie à des tailles de séquences générées encore plus longues.
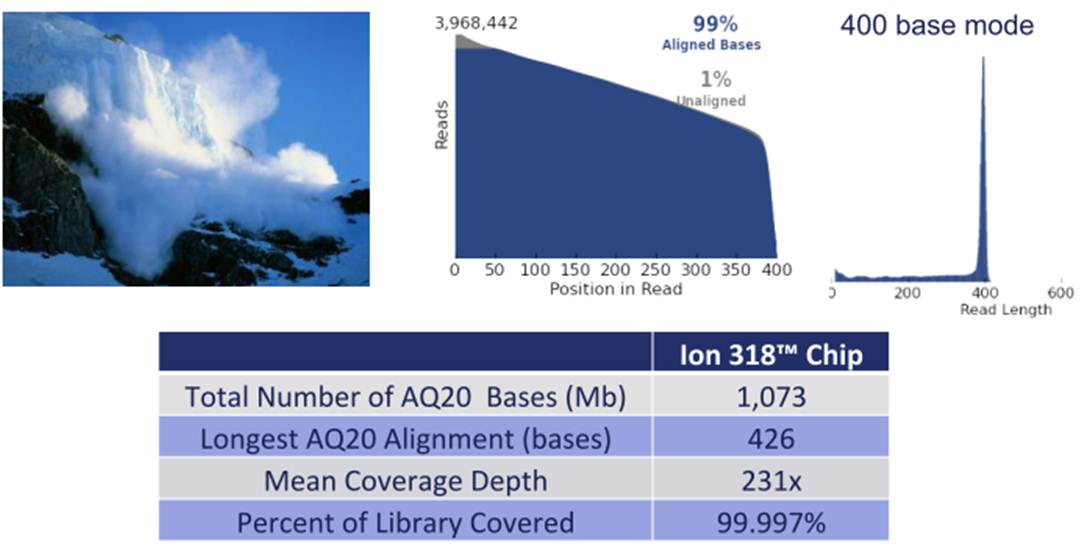
Bien entendu, Life Tech annonce l’arrivée d’un nouvel automate permettant d’intégrer la nouvelle chimie mais aussi de charger les puces (et pour quelques dizaines de k€, d’épargner au manipulateur les deux phases délicates du séquençage).
Pour conclure brièvement, l’une des nouveautés et non des moindres : l’acceptation du PGM comme diagnostic moléculaire CE-IVD (un parcours pas vraiment de tout repos) a n’en pas douter une conformité qui ne devrait pas laisser insensible les chercheurs, praticiens hospitaliers.
 Le séquençage haut-débit utilisant des analogues de nucléotides « portant des groupes bloquants en 3′ qui sont utilisés pour produire une terminaison de chaîne réversible appliqués pour le séquençage par synthèse nucléotidique » (je sais cette phrase est « un peu » lourde…) est un brevet de Caliper Technologies -autrement dit de Perkin Elmer : la firme qui n’a pas su saisir sa chance lors de la révolution du séquençage haut-débit. Le séquençage par synthèse (avec réversion du blocage des nucléotides fluorescents polymérisés -un peu moins lourde cette phrase là…) est le cœur de la technologie faisant une partie du chiffre d’affaire d’Illumina (l’intégralité de sa gamme de séquenceurs fonctionne sur ce principe).
Le séquençage haut-débit utilisant des analogues de nucléotides « portant des groupes bloquants en 3′ qui sont utilisés pour produire une terminaison de chaîne réversible appliqués pour le séquençage par synthèse nucléotidique » (je sais cette phrase est « un peu » lourde…) est un brevet de Caliper Technologies -autrement dit de Perkin Elmer : la firme qui n’a pas su saisir sa chance lors de la révolution du séquençage haut-débit. Le séquençage par synthèse (avec réversion du blocage des nucléotides fluorescents polymérisés -un peu moins lourde cette phrase là…) est le cœur de la technologie faisant une partie du chiffre d’affaire d’Illumina (l’intégralité de sa gamme de séquenceurs fonctionne sur ce principe).
Le principe assez simple du séquençage SBS (Sequencing By Synthesis) est en train de se décliner au sein de machines haut-débit plus accessibles en terme de coût… il s’agit là de l’arrivée sur le marché d’une nouvelle concurrence : des séquenceurs génériques tels que ceux que proposent le distributeur Azco Biotec, Inc. Ces séquenceurs viennent directement concurrencer ceux d’Illumina accompagnés de leurs réactifs « génériques » tels que ceux distribués par la société Proteigene…
Ces séquenceurs « génériques » ne révolutionnent rien, leur part de marché est actuellement marginale… les laboratoires académiques préfèrent souvent l’original à la copie, malgré tout la concrétisation de la « promesse » –qui l’a formulée au juste ?– d’un génome humain pour 100 $ d’ici 2015 passe par la large diffusion de machines dont la technologie a fait ses preuves… Le retard pris par les séquenceurs de 3ème génération laisse le champ libre à la 2ème. Le marché risque de se saturer en séquenceurs de paillasse (séquenceur de 2ème génération tels que le MiSeq d’Illumina et les PGM et Ion Proton de Life Technologies).
Face aux arguments commerciaux développés en faveur de tels ou tels séquenceurs de paillasse à haut débit, l’étude menée par Loman et al., Nature Biotechnology propose une comparaison des performances du 454 GS Junior (Roche), du MiSeq (Illumina) et du PGM Ion torrent (Life Technologies), à partir du séquençage de novo d’un isolat d’E.coli O104 :H4 responsable de l’intoxication alimentaire en Allemagne (Mai 2011). Pour cela, le génome de référence a été généré à l’aide du GS FLX (Roche) et permettra d’évaluer l’efficacité des séquenceurs et algorithmes d’assemblages associés.
Sur la base des caractéristiques techniques établies en 2011, les trois plateformes ont permis de générer un draft exploitable du génome bactérien sans pour autant parvenir à le reconstituer à 100%. Par conséquent, il est impossible de désigner un séquenceur comme étant celui de choix, chaque plateforme étant un compromis entre avantages et inconvénients.
Deux « runs » de 454 GS Junior (associés au bénéfice de reads de 600 bases) ont permis d’atteindre 96,28% de couverture par rapport au génome de référence (Contre 96,05% pour le MiSeq et 95,4% pour le PGM Ion torrent).
Malgré une capacité de séquençage du PGM Ion torrent supérieure à celle du 454 GS Junior (100Mb contre 70Mb) et une précision de séquençage voisine (Q20, soit 1 erreur toutes les 100 bases), la technologie reposant sur les semiconducteurs fait face à ses limites au niveau de la gestion des homopolymères et accessoirement d’une longueur de reads de 100 bases.
Vingt fois supérieur au 454 GS Junior en terme de quantité de bases décodées, un « run » de MiSeq ne parvient qu’à talonner les performances d’un double « run » de la technologie Roche. Bien qu’étant la seule à gérer les homopolymères et la seule doter d’une précision à Q30 (soit 1 erreur toutes les 1000 bases), la technologie Illumina faillit sur ses tailles de « reads » qui ne permettent pas une gestion optimum des séquences répétées lors de l’assemblage.
L’approche analytique est également évoquée et selon le système d’assemblage utilisé (Velvet, CLC, MIRA), de nombreuses différences sont observées.
Enfin, ce document est l’occasion de rassembler des notions assez pragmatiques comme le tarif des plateformes (et annexes associées), la durée d’un « run » de séquençage ou encore la quantité de données générées.
Ceci étant, on est en droit d’émettre quelques réticences quant à la pertinence de cette étude qui base son comparatif sur une application (séquençage de novo) qui n’est pas la vocation première de cette catégorie de séquenceurs. Par ailleurs, dans le laps de temps où cette étude a été réalisée et la parution des résultats, la technologique du PGM Ion torrent a évolué. Testé sur les bases d’une puce 316 (100Mb) et d’une technologie de 100 bases en longueur de « reads », le PGM bénéficie désormais de la puce 318 (1 Gb) associée à une technologie de reads de 200 bases. Sans tarder, Life technologies réagit (“Loman et al reflects the past, not the present”) et propose une mise à jour au travers de ce document.
En pleine guerre froide des compagnies de biotechnologies, développeuses et vendeuses de matériel de séquençage… une fuite de documents est organisée. Le tout pour fournir aux personnes prescriptrices ou décideuses d’un futur achat de séquenceur, les arguments pour orienter ou justifier leur choix… parfois même leur mauvaise foi.

En cliquant sur la volute nucléique (depuis le 30/08/2012 – ce lien est censuré par Illumina- cf. commentaires de ce même article) ci-dessus vous aurez accès à un vrai-faux document confidentiel fourni par Illumina. Ce document assez bien réalisé a devancé celui de Life Technologies présenté dans notre article précédent. Il met l’accent sur le bénéfice, au niveau de la qualité des données générées, que procure un MiSeq (Illumina) par rapport à un PGM (Ion Torrent, Life Technologies)
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages