Face aux arguments commerciaux développés en faveur de tels ou tels séquenceurs de paillasse à haut débit, l’étude menée par Loman et al., Nature Biotechnology propose une comparaison des performances du 454 GS Junior (Roche), du MiSeq (Illumina) et du PGM Ion torrent (Life Technologies), à partir du séquençage de novo d’un isolat d’E.coli O104 :H4 responsable de l’intoxication alimentaire en Allemagne (Mai 2011). Pour cela, le génome de référence a été généré à l’aide du GS FLX (Roche) et permettra d’évaluer l’efficacité des séquenceurs et algorithmes d’assemblages associés.
Sur la base des caractéristiques techniques établies en 2011, les trois plateformes ont permis de générer un draft exploitable du génome bactérien sans pour autant parvenir à le reconstituer à 100%. Par conséquent, il est impossible de désigner un séquenceur comme étant celui de choix, chaque plateforme étant un compromis entre avantages et inconvénients.
Deux « runs » de 454 GS Junior (associés au bénéfice de reads de 600 bases) ont permis d’atteindre 96,28% de couverture par rapport au génome de référence (Contre 96,05% pour le MiSeq et 95,4% pour le PGM Ion torrent).
Malgré une capacité de séquençage du PGM Ion torrent supérieure à celle du 454 GS Junior (100Mb contre 70Mb) et une précision de séquençage voisine (Q20, soit 1 erreur toutes les 100 bases), la technologie reposant sur les semiconducteurs fait face à ses limites au niveau de la gestion des homopolymères et accessoirement d’une longueur de reads de 100 bases.
Vingt fois supérieur au 454 GS Junior en terme de quantité de bases décodées, un « run » de MiSeq ne parvient qu’à talonner les performances d’un double « run » de la technologie Roche. Bien qu’étant la seule à gérer les homopolymères et la seule doter d’une précision à Q30 (soit 1 erreur toutes les 1000 bases), la technologie Illumina faillit sur ses tailles de « reads » qui ne permettent pas une gestion optimum des séquences répétées lors de l’assemblage.
L’approche analytique est également évoquée et selon le système d’assemblage utilisé (Velvet, CLC, MIRA), de nombreuses différences sont observées.
Enfin, ce document est l’occasion de rassembler des notions assez pragmatiques comme le tarif des plateformes (et annexes associées), la durée d’un « run » de séquençage ou encore la quantité de données générées.
Ceci étant, on est en droit d’émettre quelques réticences quant à la pertinence de cette étude qui base son comparatif sur une application (séquençage de novo) qui n’est pas la vocation première de cette catégorie de séquenceurs. Par ailleurs, dans le laps de temps où cette étude a été réalisée et la parution des résultats, la technologique du PGM Ion torrent a évolué. Testé sur les bases d’une puce 316 (100Mb) et d’une technologie de 100 bases en longueur de « reads », le PGM bénéficie désormais de la puce 318 (1 Gb) associée à une technologie de reads de 200 bases. Sans tarder, Life technologies réagit (“Loman et al reflects the past, not the present”) et propose une mise à jour au travers de ce document.
Voici un lien sur le site Molecular Biology Freeware for Windows… un lien destiné aux biologistes un peu allergiques à Linux et qui ne souhaitent pas débourser des milliers d’euros dans des logiciels qui ne peuvent s’avérer utiles que sporadiquement (pour certains).
Le site Molecular Biology Freeware for Windows est particulièrement laid mais il permet de référencer ces logiciels utiles pour bon nombre d’applications : phylogénie, alignement de séquences, etc.
L’alliance un peu paradoxale du logiciel gratuit, fiable et utile sous Windows !
Ce site-référence est pour quelques uns d’entre vous… : il classe les logiciels outils de biologie moléculaire en fonction des catégories suivantes :
A. DNA, RNA and genomic analysis
B. Plasmid graphic packages
C. Primer design
D. Protein analysis
E. Viewing three dimensional structures
F. Alignments
G. Phylogeny
H. Miscellaneous
I. Graphic packages
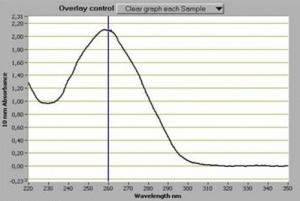
 Souvent perçu comme une évidence, le dosage d’acides nucléiques est un point fondamental. Il faut pourtant le considérer comme le premier « monitoring » important d’une expérience de biologie moléculaire. Que vous soyez équipés d’un NanoDrop™, d’un plus antique spectrophotomètre, d’un fluorimètre… les outils sont parfois complémentaires et ont des spécificités propres qu’il vous faudra évidemment connaître (au moins approximativement).
Souvent perçu comme une évidence, le dosage d’acides nucléiques est un point fondamental. Il faut pourtant le considérer comme le premier « monitoring » important d’une expérience de biologie moléculaire. Que vous soyez équipés d’un NanoDrop™, d’un plus antique spectrophotomètre, d’un fluorimètre… les outils sont parfois complémentaires et ont des spécificités propres qu’il vous faudra évidemment connaître (au moins approximativement).
En matière de dosage des acides nucléiques, il apparaît fondamental (si vous utilisez un spectrophotomètre) de connaître la fameuse loi de Beer où A=Ɛ.l.C
A étant l’absorbance (sans unité) et Ɛ est le coefficient d’absorption moléculaire (unité : L.mol -1 .cm-1)
l : la longueur du trajet optique (valeur fixe, donnée par l’épaisseur d’une cuve ou d’un ménisque sur un Nanodrop) (unité : cm)
C, souvent ce que l’on souhaite déterminer: la concentration d’acides nucléiques (unité : mol.L-1)
Cette formule permet d’estimer assez aisément une concentration en acides nucléiques, elle est vérifiée lorsque la solution est de concentration inférieure à : c < 0,1 mol.L-1
| A260 nm | ||
| acides nucléiques | Ɛ (M-1.cm-1) | pour 1 unité absorbance (µg.mL-1) |
| ADN db | 6200 | 50 |
| ADN ss | 8350 | 37 |
| ARN | 7700 | 40 |
| standard : température ambiante, l=1cm, A260/A280 =1,80 | ||
Assez rapidement, la concentration, le plus souvent en ng.µL-1, est déduite. Le point fort de la méthode spectrophotométrique (comparée à la méthode fluorométrique, plus sensible, quant à elle) est fournie par les informations que l’on tire des ratios suivants:
Ratio A260/A280 : un ratio de 1,80 (+/- 0,1) permet de qualifier une extraction d’ADN comme pure, ce ratio se situe autour de 2,0 pour une extraction d’ARN (+/- 0,1). Si ce ratio est inférieur cela signifie que l’extraction est polluée par des protéines et/ou des composés phénoliques
Ratio A260/A230 : la valeur attendue de ce ratio se situe entre 2,0 et 2,20. Si ce ratio est plus faible cela indique nécessairement qu’un composé absorbe à 230 nm. Ces composés peuvent le plus souvent être de l’EDTA, des sucres, et encore une fois le phénol et particulièrement le TRIzol reagent.
Ces valeurs de ratios peuvent parfois être négligées, cependant elles donnent une indication quant à la présence d’éventuels inhibiteurs qui viendraient perturber le devenir de vos acides nucléiques (PCR et autres réactions enzymatiques). Il est important de réaliser une valeur de blanc sur la solution qui vous aura servi à éluer vos acides nucléiques (car comme il a été précédemment mentionné l’EDTA absorbe à 230 nm… et bien souvent le TE 1X sert à reprendre et conserver des acides nucléiques)
vous trouverez ci-contre le mode d’emploi du Nanodrop 1000.
Une autre méthode alternative, permettant une meilleure sensibilité : méthode picogreen. Cette méthode, bien que très sensible, donne des résultats non valides (car très largement sous-estimés) dès lors que votre solution d’acides nucléiques est polluée par des traces d’agents détergents (type SDS). En effet, ces « adjuvants » quenchent la fluorescence émise par l’agent qui s’intercale au sein des acides nucléiques que l’on souhaite doser.
Note : bien souvent il vous est utile de calculer la quantité de molécules (quantité de copies).
Sachant que la masse molaire moyenne d’une base azotée => Masse Molaire (MM) : 309 g.mol-1
&
avec le nombre d’Avogadro 6,02214129.1023 molécules.mol-1
La formule devient : nombre de copies pour 1 µL = [C (concentration mesurée en ng/µL) x V (volume, ici 1 µL) x 10-9 ]/ [(MM environ 309 g.mol-1) x la taille de votre génome en pb x 2] le tout que l’on multiplie par le nombre d’Avogadro… le tour est joué !
plus simplement : 978 Mb pèsent 1 pg (en avant le produit en croix).
 Plusieurs de nos articles évoquent la technologie d’Oxford Nanopore, entre impatience et curiosité.
Plusieurs de nos articles évoquent la technologie d’Oxford Nanopore, entre impatience et curiosité.
Le principe de cette technologie : séquencer sans pré-amplification (il s’agit d’un séquenceur de 3ème génération) une molécule d’ADN en en hydrolysant les nucléotides élémentaires, qui passeront dans des pores. Ces pores seront couplés à des capteurs saisissant la signature singulière de chaque base constitutive de la molécule à séquencer. Jusqu’à présent le problème majeur sur lequel ont buté les ingénieurs d’Oxford Nanopore : accélérer la célérité d’acquisition des capteurs ou ralentir le passage des nucléotides au travers des pores -nucléotides qui, au départ, s’engouffraient à plus de 1000 nucléotides par seconde ce qui provoquait des problèmes d’acquisitions des signaux et donc des erreurs, sachant que l’objectif étant de ne pas dépasser les 400 bases / seconde, seuil au-delà duquel le capture devient fou…
Le 3 mai 2012, Oxford Nanopore annonce une collecte de fonds de 31,4 millions de livres (environ 50,8 millions de dollars). Encore plus de fond pour un séquenceur qui deviendrait pour un peu, un gouffre financier… « Ce tour de table a permis une levée de fonds dont la quasi-totalité provient d’investisseurs existants, ces fonds permettront un développement de l’entreprise, comprenant le développement de notre infrastructure, un renforcement de nos forces de vente, une optimisation de la fabrication et la poursuite de la R & D » a déclaré le Dr Gordon Sanghera, chef de la direction d’Oxford Nanopore. Oxford Nanopore se préparerait donc (enfin) à vendre ses produits qui sont attendus pour la fin d’année 2012. Phénomène plutôt rassurant pour la société anglaise, elle bénéficie de la confiance renouvelée de ces investisseurs, plus informés que nous sur ce que renferment (vraiment) ses tiroirs.
Eviter l’écueil d’un PacBio, trop cher, commettant trop d’erreurs, arrivé pour occuper le marché balbutiant du séquençage troisième génération, semble être la marque d’Oxford Nanopore -une société qui prend son temps. Après la possibilité de séquencer des molécules d’ADN à l’état natif, de taille très longue, en un temps record, il semble qu’Oxford Nanopore, avec ce nouvel appel de fonds, ficelle sa stratégie commerciale pour un lancement en grande pompe de leurs 2 références : le GridION & le MinION, la clé USB qui séquence dont vous pouvez voir la photographie ci-contre
Voici un documentaire diffusé sur Arte qui aborde en un peu plus de 50 minutes la biologie synthétique. Laetita Ohnona, la réalisatrice du film, pondère les interventions des Craig Venter et autres ingénieurs du MIT, technoscientistes par plusieurs interventions d’un lanceur d’alerte, Jim Thomas, filmé devant une campagne rassurante qui contraste avec l’univers d’un laboratoire. Ce documentaire très pédagogique (à mes yeux, il s’agit de sa principale qualité), consacre les ingénieurs face aux chercheurs -de la recherche appliquée vers la recherche fondamentale, en quelque sorte. Ainsi des parallèles sont réalisés entre des Steve Jobs & Bill Gates, deux personnages ayant révolutionné les applications liées à l’informatique et des Randy Rettberg & Tom Knight (co-fondateurs du prix Igem), des ingénieurs-chercheurs ayant œuvré en informatique pour terminer leur carrière dans les biotechnologies.
L’expérience de Philippe Marlière est développée en fin de documentaire pour apporter un début de solution au contrôle des organismes synthétiques comme outils de production.
Ce documentaire avivera la curiosité des scientifiques en herbe attiré par l’image d’Épinal de biogeek construisant un biofermenteur dans le garage de papa, effraiera les sceptiques, et très certainement produira un peu ces deux effets sur les initiés. On pourra simplement regretter l’absence d’un regard bioéthique dans ce documentaire qui permettrait de prendre un certain recul, évitant les allers-retours entre fascination technologique et peur de fin du monde, un monde ravagé par des bactéries tueuses luminescentes…
Ci-dessous : le synopsis d’Arte concernant ce documentaire
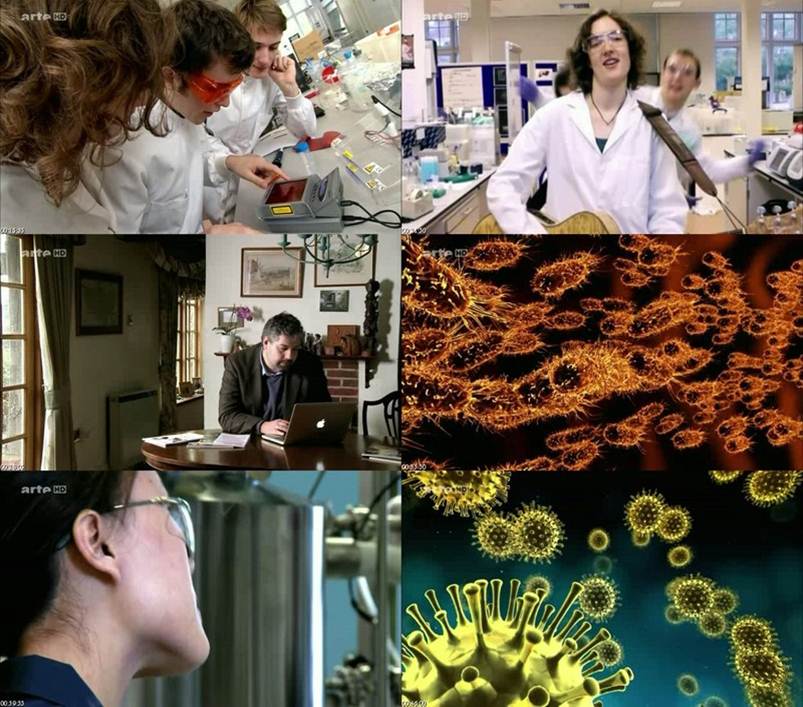
Les spécialistes sont unanimes : la biologie connaît une révolution aussi décisive que la découverte de l’ADN ou le séquençage complet du génome humain. La biologie de synthèse doit permettre à moyen terme à l’homme de se faire démiurge, pour écrire des séquences partielles ou entières d’ADN – et donc créer ainsi artificiellement la vie. Une perspective tellement nouvelle qu’elle suscite des vocations en dehors des laboratoires eux-mêmes. Dans de simples garages ou des hangars, les « biohackers » sont aujourd’hui capables d’assembler des séquences d’ADN synthétiques (via des DNA cassettes ou biobricks disponibles sur Internet). Peut-être donneront-ils vie à de nouvelles cellules, voire à des organismes entiers. Certains experts posent la question sans détour : l’homme va-t-il, pour la première fois, prendre la main sur l’évolution naturelle des espèces ? Et si oui, quelles seront les conséquences pour les écosystèmes les plus fragiles, et l’équilibre des espèces vivantes ? De nombreux acteurs, dont les plus gros laboratoires, investissent en tout cas dans la biologie de synthèse, jusqu’à Bill Gates, qui finance un programme colossal contre la malaria reposant sur elle. C’est cette révolution en marche que ce film souhaite montrer pour la première fois, en partant à la rencontre de ceux qui la portent.
(France, 2012, 52mn)
ARTE
Date de première diffusion : Jeudi 26 avr. 2012, 22h25
Date(s) de rediffusion : Samedi 5 mai 2012, 10h45
Vidéo disponible sur : http://videos.arte.tv/fr/videos/fabriquer_le_vivant-6609804.html
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages