Selon une étude : La consommation (modérée) de vin rouge liée à une meilleure santé intestinale…
Une étude du King’s College de Londres tend à montrer que les personnes qui boivent du vin rouge présenteraient une plus grande diversité de microbiote intestinal (un signe de santé intestinale) que les buveurs de vin non rouge.
Dans un article publié le 28 août dans la revue Gastroenterology, une équipe de chercheurs du Department of Twin Research & Genetic Epidemiology au King’s College de Londres a étudié l’effet de la bière, du cidre, du vin rouge, du vin blanc et des spiritueux sur le microbiote intestinal et sur la santé qui en découle chez un groupe de 916 jumelles britanniques. Pourquoi des jumelles? Pour que le fond génétique soit identique, donc la différence de microbiote intestinal entres les jumelles sera en grande partie dû à l’environnement.
Ils ont constaté que le microbiote intestinal des buveurs de vin rouge était plus diversifié que celui des buveurs de vin non rouge. Ceci n’a pas été observé avec la consommation de vin blanc, de bière ou de spiritueux.
La première auteure de l’étude, Caroline Le Roy, du King’s College de Londres, a déclaré : « Bien que nous connaissions depuis longtemps les bienfaits inexpliqués du vin rouge sur la santé cardiaque, cette étude montre qu’une consommation modérée de vin rouge est associée à une plus grande diversité et à un microbiote intestinal plus sain qui explique en partie ses effets bénéfiques sur la santé. »
Le microbiome est la collection de micro-organismes dans un environnement et joue un rôle important dans la santé humaine. Un déséquilibre entre les « bons » microbes et les « mauvais » microbes dans l’intestin peut entraîner des effets néfastes sur la santé, comme un affaiblissement du système immunitaire, un gain de poids ou un taux de cholestérol élevé.
Le microbiote intestinal d’une personne ayant un nombre plus élevé d’espèces bactériennes différentes peut-être considéré comme un marqueur de la santé intestinale.
L’équipe a observé que le microbiote intestinal des consommateurs de vin rouge contenait un plus grand nombre d’espèces bactériennes différentes que celui des non-consommateurs. Ce résultat a également été observé dans trois cohortes différentes au Royaume-Uni, aux États-Unis et aux Pays-Bas. Les auteurs ont tenu compte de facteurs tels que l’âge, le poids, le régime alimentaire régulier et le statut socio-économique des participants et ont continué à voir l’association.
Les auteurs croient que la raison principale de cette association est due aux nombreux polyphénols présents dans le vin rouge. Les polyphénols sont des produits chimiques de défense naturellement présents dans de nombreux fruits et légumes. Ils ont de nombreuses propriétés bénéfiques (y compris des antioxydants) et agissent principalement comme un carburant pour les microbes présents dans notre système.
L’auteur principal, le professeur Tim Spector du King’s College de Londres, a déclaré : « Il s’agit de l’une des plus importantes études jamais réalisées sur les effets du vin rouge sur l’intestin de près de trois mille personnes dans trois pays différents et elle montre que les niveaux élevés de polyphénols dans la peau du raisin pourraient être responsables d’une grande partie des bienfaits controversés pour la santé lorsqu’ils sont utilisés avec modération. »
« Bien que nous ayons observé une association entre la consommation de vin rouge et la diversité du microbiote intestinal, boire du vin rouge rarement, comme une fois toutes les deux semaines, semble suffisant pour observer un effet. Si vous devez choisir une boisson alcoolisée aujourd’hui, c’est le vin rouge qu’il faut choisir, car il semble exercer un effet bénéfique sur vous et sur vos microbes intestinaux, ce qui peut aussi aider à réduire le poids et le risque de maladies cardiaques. Cependant, il est toujours conseillé de consommer de l’alcool avec modération, vous n’avez pas à boire du vin rouge, et vous n’avez pas à commencer à en boire si vous ne buvez pas », a ajouté le Dr Le Roy.
Effectivement, comme dans toute étude, corrélation n’est pas raison! Ainsi et même si la catégorie socio-économique est prise en compte dans l’étude, il pourrait exister d’autres facteurs, non mesurés dans cette étude, qui expliqueraient en partie la bonne santé microbienne des individus. Rappelons que ça n’est pas l’alcool qui est associé avec une meilleure santé intestinale mais d’autres composants du vin rouge (l’hypothèse étant que ce sont les polyphénols), que l’on trouvera aisément dans d’autres aliments (fruits, légumes, noix, cacao…) .
Emplacement de la publication originale :
https://www.sciencedirect.com/science/article/abs/pii/S0016508519412444
L’abus d’alcool est dangereux pour la santé, consommez avec modération
Comment réagiriez vous si l’on vous « offrait » votre génotypage complet, ouvrant la possibilité de prédire d’éventuelles maladies, réactions à certains médicaments etc. ? Certes, dans le cas de l’Estonie (contrairement à ce que propose des sociétés privées telles 23andMe) il s’agit d’une démarche s’inscrivant en santé publique pour le meilleur du bien public et non pour le pire du bien privé… Néanmoins, l’ampleur de la cohorte humaine visée n’est pas sans poser des questions.
L’Estonie a lancé un programme visant à recruter et à génotyper 100.000 nouveaux participants à la biobanque (pour une population nationale totale de 1,316 million de citoyens estoniens !) dans le cadre de son programme national de médecine personnalisée. Cette biobanque comportait déjà 50.000 génotypes de citoyens estoniens. Le gouvernement veut développer un système de soins de santé en offrant à un maximum de ses résidents un génotypage scannant le génome qui sera traduit en rapports personnalisés. Ce rapport serait intégré à la pratique médicale quotidienne par l’entremise du portail national de cybersanté.
Cette biobanque a été initiée à trois fins (Leitsalu et al., International Journal of Epidemiology, 2015) :
- promouvoir le développement de la recherche génétique ;
- recueillir des informations sur l’état de santé de la population estonienne, ainsi que des informations génétiques
- utiliser les résultats de la recherche génétique pour améliorer la santé publique.

Au sein de l’article de Leitsalu et al., les auteurs abordent les forces et faiblesses de leur entreprise :
La principale force de la Biobanque estonienne consiste au fait qu’il s’agit d’une biobanque basée sur la population avec une base de données longitudinales et prospectives. Cela signifie qu’un large éventail de groupes d’âge et de phénotypes sont représentés. Alors que les populations urbaines ont généralement tendance à être surreprésentées, ce n’est pas le cas pour la cohorte de la Biobanque estonienne. La biobanque dispose d’ADN, de plasma et de globules blancs pour chaque donneur. Cela signifie qu’il est possible d’analyser les effets directs des variants de séquence sur le métabolisme. De plus, il est possible de transformer les cellules en lignées cellulaires ou en cellules souches pluripotentes induites (iPS) et de réaliser directement des expériences de biologie moléculaire ou de génétique. Une autre force est fournie par la HGRA (the Estonian Human Genes Research Act) ainsi que par le formulaire de consentement général qui permet de participer à un large éventail de projets de recherche sans avoir à communiquer de nouveau et à demander un nouveau consentement. La HGRA et le formulaire de consentement permettent également aux donneurs de demander la divulgation de leurs données génétiques, de leurs caractéristiques héréditaires et des risques génétiques obtenus à partir de la recherche génétique menée. Cela permettrait à terme de mener des projets sur les tests du génome personnel, la perception des risques et la gestion des risques en milieu industriel.
De plus, la HGRA permet à la Biobanque d’obtenir des renseignements supplémentaires en reliant les dossiers aux registres électroniques nationaux et aux principaux hôpitaux. Tous les registres sont reliés de façon centralisée par une infrastructure technique à l’échelle nationale qui permet l’échange sécurisé de données entre les bases de données. La HGRA a également imposé des restrictions sur les activités de l’EGCUT (Estonian Genome Center of the University of Tartu) et les données collectées dans la Biobanque estonienne. La participation devait être entièrement volontaire – seules les personnes intéressées pour rejoindre la Biobanque estonienne, après en avoir entendu parler soit lors d’événements promotionnels spéciaux, soit par les médias, soit par des amis, soit au cabinet du médecin de famille ou à l’hôpital, sont recrutées. L’EGCUT n’a pas été autorisé à envoyer les lettres d’invitation à leur adresse domiciliaire. Par conséquent, la biobanque ne représente pas un échantillon aléatoire classique et pourrait être sujette à un biais de recrutement. Une proportion considérable de la population recrutée pourrait toutefois compenser ce biais. Par conséquent, bien qu’elle ne soit pas aléatoire sur le plan classique, la cohorte peut quand même être considérée comme représentative de la population. Bien que le recrutement était ouvert à tous, il y a une disproportion d’Estoniens ethniques et de Russes ethniques dans la biobanque, les Estoniens étant surreprésentés (81% dans la biobanque contre 70% dans la population générale) et les Russes sous-représentés (16% dans la biobanque contre 25% dans la population générale). Une autre faiblesse est la profondeur limitée de certains sous-questionnaires. Par exemple, un questionnaire relativement bref sur la fréquence des aliments a été administré sans information détaillée sur l’apport en énergie ou en nutriments ; les mesures des traits glycémiques à jeun, comme le niveau d’insuline, ne sont disponibles que pour un nombre limité d’échantillons. La profondeur limitée des données recueillies peut parfois limiter le nombre de projets dans lesquels les données peuvent être utilisées. Toutefois, des questionnaires plus complets auraient exigé des durées d’entrevue encore plus longues et auraient coûté beaucoup plus cher, ce qui aurait pu entraîner une réduction de la taille de la cohorte.
Le pays dispose de nombreuses solutions numériques sécurisées incorporées dans les fonctions gouvernementales qui relient les diverses bases de données du pays par des voies cryptées de bout en bout. Un site Web a été créé dans le cadre du projet, afin que les Estoniens puissent se porter volontaires et donner leur consentement à être génotypés. La génération des données est assurée par l’institut de génomique de l’Université de Tartu (aujourd’hui les 50.000 premiers génotypages ont été réalisés et analysés, les 100.000 pousseront à une population estonienne à 10 % génotypée)
Les efforts internationaux ont permis d’identifier des milliers d’associations entre les variants génétiques et les maladies, ou traits génétiques, et de créer des cartes des variations uniques au sein des populations.
« Aujourd’hui, nous avons suffisamment de connaissances sur le risque génétique des maladies complexes et la variabilité interindividuelle des effets des médicaments pour commencer à utiliser systématiquement ces informations dans les soins de santé au quotidien « , a déclaré Jevgeni Ossinovski, ministre de la Santé et du Travail. « En coopération avec l’Institut national pour le développement de la santé et l’Université de Tartu, nous allons permettre à 100.000 autres personnes de rejoindre la biobanque estonienne, afin de stimuler le développement de la médecine personnalisée en Estonie et de contribuer ainsi à l’avancement des soins de santé préventifs. «
Le gouvernement estonien a alloué 5 millions d’euros au programme au cours de l’année 2018. Le projet sera coordonné par l’Institut national pour le développement de la santé, dont la tâche est d’élaborer et de mettre en œuvre des procédures et des principes pour la mise en œuvre efficace de la recherche scientifique dans la pratique médicale.
Andres Metspalu, directeur du Centre estonien du génome à l’Université de Tartu, se félicite de l’initiative du ministère des Affaires sociales d’augmenter le nombre de participants à la biobanque. « Nous sommes heureux qu’avec le soutien de ce projet, les résultats des travaux à long terme du centre de génomique seront transférés en médecine pratique et donneront un nouvel élan à nos recherches futures. L’Université contribuera également à la création d’un système de rétroaction pour les participants de la biobanque, et à la formation des professionnels de la santé pour qu’ils puissent donner aux patients une rétroaction fondée sur l’information génétique« .
Le projet sera mis en œuvre sur la base de la loi estonienne sur la recherche sur les gènes humains et du même formulaire de consentement général qui a été utilisé pour les 50.000 premiers participants. Le prélèvement officiel d’échantillons a débuté le 2 avril 2018. A voir si l’expérience relativement pionnière ,à cette échelle, menée en Estonie, fera école ou tâche d’huile dans d’autres pays de l’Union Européenne !

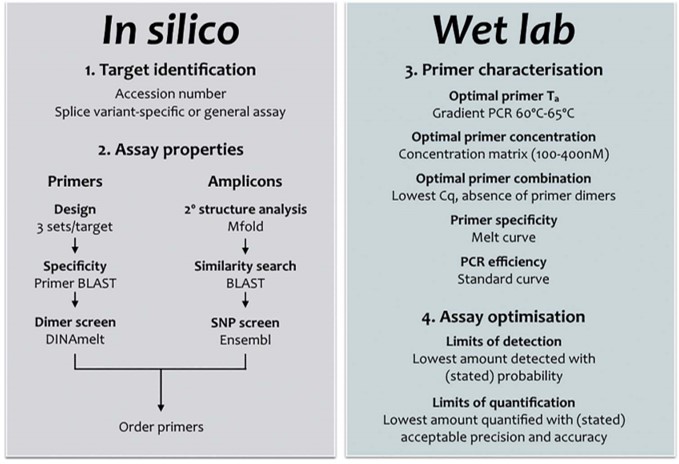
La qPCR est une méthode permettant de doser la quantité d’acides nucléiques ciblés introduits dans une réaction de PCR. Pour des raisons de rapidité, de sensibilité et de coût, souvent l’option de travailler avec un agent intercalant (sans sonde) est choisie. La bonne vieille qPCR SybGreen nécessitant le seul design d’une paire d’amorces…
Simple ? Pas nécessairement, car cette approche, certainement plus que la version qPCR Taqman nécessite un travail in silico et de validation/optimisation expérimentales comme passages obligés. C’est ce que montre la publication de Stephen Bustin et Jim Huggett dans Biomolecular Detection and Quantification. Cette publication incontournable pour les férus de qPCR SybrGreen est un beau travail pour lequel la publication vous est mise à disposition en cliquant ci-dessous. On attend ardemment une déclinaison Taqman, HRM, MolecularBeacon de ce type de revues permettant de formaliser des procédures visant à optimiser l’approche d’optimisation.
La quantification par qPCR SybrGreen suppose une relation linéaire entre le logarithme de la quantité initiale introduite en PCR et la valeur Cq obtenue lors de l’amplification. Ceci permet de calculer l’efficacité d’amplification d’un test et de borner ses limites de détection et de quantification. Les caractéristiques d’un test qPCR (bien) optimisé sont les suivantes:
• Une excellentissime spécificité révélée par un pic unique lors de l’établissement de la courbe de fusion
• Une efficacité d’amplification élevée (95-105%)
• Une courbe étalon linéaire (R2 > 0,980)
• Une bonne répétabilité
• Peu ou prou de dimères d’amorces
Pour paraphraser la conclusion de l’article, afin de finir par convaincre de lire cet « essentiel » de la qPCR :
La conception, le design d’une PCR est souvent au cœur de tout projet de recherche visant à quantifier les acides nucléiques. Il doit être réalisé avec soin, mais peut être simplifié en suivant un flux de travail simple, comme décrit ci-dessus (cf. diagramme workflow design qPCR).
Cela signifie généralement une spécificité absolue, l’absence de structures en épingle à cheveux ou de potentielles dimérisations croisées. Une bonne conception des essais doit tenir compte de la structure de l’amplicon (paramètre souvent négligé) et veiller à ce que les cibles de l’amorce soient exempts de structure secondaire. Il existe de nombreuses opinions et lignes directrices; une recherche sur Internet pour les termes « qPCR Assay Design » renvoie 695.000 pages. Cependant, bon nombre de ceux-ci sont basés sur des mythes ou peuvent être appropriés pour la PCR mais nécessitent des modifications subtiles (ou moins subtiles) pour être utilisés pour développer une qPCR. Chaque « nouveau » dosage doit être correctement validé, la validation in silico servant de filtre initial pour éliminer des designs ne permettant pas d’aboutir à une bonne qPCR. L’optimisation et la validation empirique sont une partie essentielle, mais souvent négligée, de toute expérience qPCR. Cela s’applique aussi bien aux essais nouvellement conçus qu’aux essais obtenus en reprenant des amorces issues d’une publication, par exemple. Avec tant d’essais prêts à l’emploi, on peut se demander pourquoi quelqu’un voudrait se donner la peine de concevoir un autre essai. D’autant plus que l’on a l’impression que la conception de son propre test est beaucoup plus complexe et peu commode que de simplement l’acheter à un fournisseur commercial, qui en tout cas aura validé chacun de ses tests. Cette perception est erronée pour deux raisons:
1° il se peut que les amorces commerciales ou les conditions d’analyse n’aient pas été validées ou optimisées de façon expérimentale.
2° on ne peut pas présumer qu’un ensemble d’amorces produira les mêmes résultats dans des conditions expérimentales différentes puisque la performance du dosage peut varier selon les méthodes d’extraction utilisées pour purifier les acides nucléiques.
 Voici une initiative originale conciliant deux tendances du moment : étude d’un microbiote & le concept de smart city. Saugrenu ? Science tendance ? Projet formaté pour la vulgarisation, la valorisation médiatique ? Quoi qu’il en soit, ce projet nous est plutôt bien vendu, à grand renfort d’infographies, de photographies, de vidéos un peu inquiétantes de personnes en train de réaliser des prélèvements de surface dans le métro. Derrière la communication, l’idée du projet est séduisante : utiliser des données biologiques, en l’occurrence des profils de l’ADNr 16S ou des séquençages génomes entiers définissant un microbiote pour améliorer nos écosystèmes urbains. Il est vrai qu’à une époque où l’on conçoit du mobilier urbain volontairement inconfortable pour ne pas que le passant puisse faire autre chose que passer, il ne semble pas intuitif d’aller chasser le microbiote pour concevoir une ville moins idiote. Même si la ville est le terrain de tous les paradoxes, il est captivant d’observer que cet environnement quotidien, banal, renferme une part de mystère… mystère qui serait à l’origine des odeurs du métro ? quelles espèces bactériennes propres à New York, Paris ou Rome sont à découvrir… quelle ville a le microbiote le plus diversifié ? quelle est celle qui aura le privilège d’héberger le plus de bactéries résistantes aux antibiotiques, la plus propre ou la plus sale ?
Voici une initiative originale conciliant deux tendances du moment : étude d’un microbiote & le concept de smart city. Saugrenu ? Science tendance ? Projet formaté pour la vulgarisation, la valorisation médiatique ? Quoi qu’il en soit, ce projet nous est plutôt bien vendu, à grand renfort d’infographies, de photographies, de vidéos un peu inquiétantes de personnes en train de réaliser des prélèvements de surface dans le métro. Derrière la communication, l’idée du projet est séduisante : utiliser des données biologiques, en l’occurrence des profils de l’ADNr 16S ou des séquençages génomes entiers définissant un microbiote pour améliorer nos écosystèmes urbains. Il est vrai qu’à une époque où l’on conçoit du mobilier urbain volontairement inconfortable pour ne pas que le passant puisse faire autre chose que passer, il ne semble pas intuitif d’aller chasser le microbiote pour concevoir une ville moins idiote. Même si la ville est le terrain de tous les paradoxes, il est captivant d’observer que cet environnement quotidien, banal, renferme une part de mystère… mystère qui serait à l’origine des odeurs du métro ? quelles espèces bactériennes propres à New York, Paris ou Rome sont à découvrir… quelle ville a le microbiote le plus diversifié ? quelle est celle qui aura le privilège d’héberger le plus de bactéries résistantes aux antibiotiques, la plus propre ou la plus sale ?

En juillet 2017, Stockholm accueillera la 3ème conférence annuelle « Metagenomics and Metadesign of Subways and Urban Biomes (MetaSUB)« , qui rassemblera des chercheurs dédiés à la cartographie du métagénome urbain de plus de 67 villes à travers le monde (dont Paris et Marseille). Ce projet ambitieux a débuté il y a deux ans, alors que le professeur Christopher Mason et son équipe ont réalisé la première étude sur la microflore de surface, dressant le microbiome de la ville de New York. Avec ces données moléculaires essentiellement basées sur le séquençage haut-débit 16S, le projet a étudié la façon dont ce «microbiome urbain» change avec des variables telles que la météo, la propreté, les matériaux de construction et même les niveaux socio-économiques du quartier. L’équipe a cherché à établir des profils de microbiotes dans le métro, à identifier les bio-menaces potentielles et à fournir des données complémentaires qui peut être utilisé par la ville pour créer une «ville intelligente», c’est-à-dire qui agrège des données hétérogènes pour améliorer son urbanisme.
Près de la moitié (48%) de l’ADN séquencé ne correspondait pas aux organismes connus, soulignant qu’un microbiota incognita entoure les usagers du métro. Le projet du métro de New York n’était que le début, ce qui a conduit à la création d’un consortium international de laboratoires pour établir une « cartographie » mondiale de microbiomes dans les systèmes de transport en commun notamment. En cliquant sur la capture d’écran ci-dessous, on pourra se faire une idée de la communication « Metasubienne » qui n’a rien à envier à celle de Tara Oceans ou de MetaHit (qui sont toutes trois, d’excellentes communications autour d’un projet et d’un consortium).
Voici en quelques mots les promesses annoncées dans l’article Geospatial Resolution of Human and Bacterial Diversity with City-Scale Metagenomics (Cell Syst, 2016) :
« La région métropolitaine de New York City (NYC) est un endroit idéal pour entreprendre une étude métagénomique à grande échelle car c’est la plus grande et la plus dense des États-Unis; 8,2 millions de personnes vivent sur une masse continentale de seulement 755 km2. En outre, le métro de New York est le plus grand système de transport en commun dans le monde (par le nombre de stations), qui s’étend sur plus de 406 km et utilisé par 1,7 milliard de personnes par an. Ce vaste écosystème urbain est une ressource précieuse qui nécessite un suivi pour le maintenir et le sécuriser contre les actes de bioterrorisme, les perturbations environnementales ou les épidémies. Ainsi, nous avons cherché à caractériser le métagénome de NYC en examinant le matériel génétique des microorganismes et d’autres ADN présents dans, autour et au-dessous de New York, en mettant l’accent sur les métros et les zones publiques très empruntés. Nous envisageons cela comme une première étape vers l’identification des menaces biologiques potentielles, la protection de la santé des New-Yorkais et la mise à disposition de données moléculaires qui pourront être utilisée par la ville pour créer une «ville intelligente», c’est-à-dire celle qui utilise des données de grande dimension pour améliorer l’urbanisme, la gestion de l’environnement bâti, des transports en commun et de la santé humaine. »
Bien évidemment cette infographie vue d’ici vous parait assez peu lisible. Après avoir cliqué dessus, vous pourrez vous apercevoir qu’elle permet de s’y retrouver dans la jungle de ces molécules aux noms tous plus baroques… 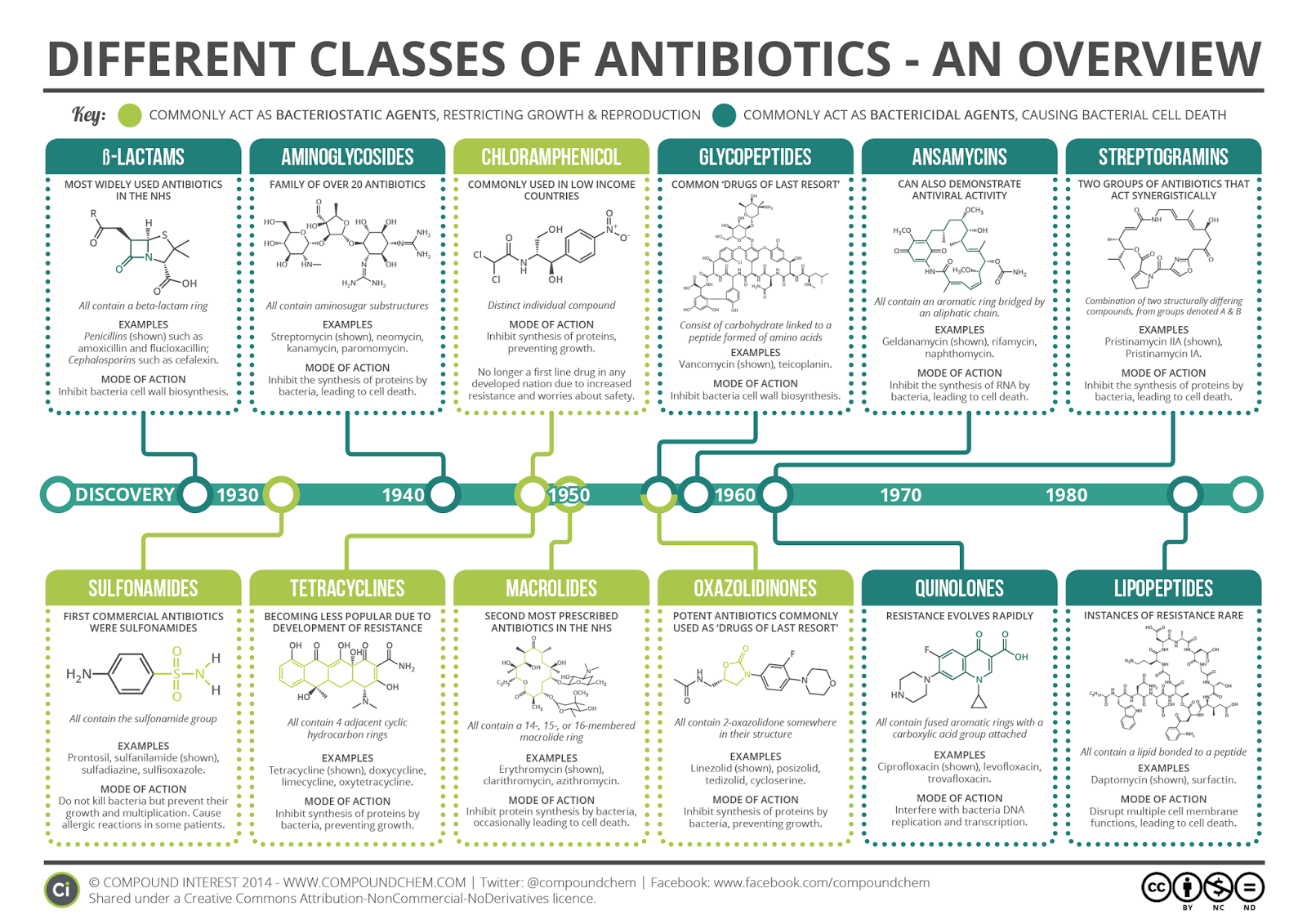
Un article du Sydney Morning Herald du 20 novembre 2016 réalise un assez bon plan de communication pour la société Microba.
L’objet : les tribulations gastro-dentaires d’un testeur du service que souhaite bientôt proposer la société investissant le marché de la métagénomique personnelle… Dans la roue des sociétés proposant des services de génomique personnelle voire de génomique récréative, voici venir celles proposant d’appliquer le séquençage haut-débit couplé à de l’analyse automatisée de séquences pour révéler une part de ce qui constitue votre microflore corporelle. Presque triviales, ces analyses métagénomiques sont aujourd’hui à la portée de qui peut les payer. Vous qui ne savez qu’offrir à Noël… peut être une piste de cadeau ! La question que l’on peut légitimement se poser est : à quelles fins analyser votre flore intestinale…? revenons sur l’article du Sydney Morning Herald pour tenter d’y trouver un intérêt ou deux.

L’article commence sur un mode enthousiaste, la technologie permet de révéler une part de notre microbiote… à quoi bon s’en priver ? Le gonzo-journaliste rédacteur de l’article se propose comme cobaye et reçoit des « cotons tiges » de prélèvements qui lui permettent de procéder à l’envoi d’échantillons censés être des prélèvements de sa microflore corporelle -buccale et intestinale. Pas simple l’échantillonnage de la flore intestinale avec un coton tige ! Ainsi que le dit Dr Alena Rinke, porte-parole de Microba : « la recherche dans ce domaine vient d’exploser au cours des cinq ou dix dernières années. Nous constatons qu’il existe un nombre incroyable de liens entre les microbes de notre corps et des états pathologiques« . Ces « liens » peuvent être très divers : diabète, cancer du côlon, maladie inflammatoire de l’intestin, mélanome, polyarthrite rhumatoïde. En guise de commentaire, disons que tout comme corrélation n’est pas raison, liaison n’est pas causalité non plus. L’argument santé finit de motiver le jeune investigateur de sa flore intérieure. Donc le journaliste expédie ses cotons tiges à la société australienne qui réalise une métagénomique ciblée (séquençage haut-débit d’une portion du gène de l’ARN 16S).
Six semaines plus tard, ses résultats lui parviennent. Bilan : beaucoup de Ruminococcus, Blautia, Prevotella copri. Le petit bestiaire personnel qu’il héberge le fascine. Le test de Microba tente de mettre une couche d’interprétation de ces données de séquences mais le rendu semble un peu insatisfaisant pour le jeune intrépide. Il demande au professeur Andrew Holmes (plutôt sceptique sur l’intérêt du service) d’examiner rapidement ses résultats. La première métrique clé qui lui saute aux yeux est le nombre de Proteobacteria présente, dit – il. » En général, une personne en bonne santé a une faible proportion de bactéries dites « pro-inflammatoires ». Il faudrait pour Proteobacteria être inférieur à 5 %, et idéalement moins de 2 %. »
Le journaliste se voit rassuré avec une proportion de Proteobacteria, inférieure à 1 %.
Son microbiote est légèrement plus diversifié que la moyenne – c’est un bon point. Cela signifie qu’il est en bonne santé avec/grâce à un régime alimentaire équilibré et lui-même diversifié. Dans l’ensemble, le test de Microba conclut que sa flore intestinale suggère qu’il est en «excellente» santé avec un IMC de 20,7. Basé sur l’information de son microbiote, le test prédit qu’il a 28 ans (plutôt pas mal puisqu’il en accuse 26, avec un IMC de 21,9).
L’enthousiasme retombe un peu quand le cobaye-journaliste promoteur du service de Microba apprend qu’il a des proportions de Dialister en quantités supérieures à la moyenne. Dialister est un bon prédicteur de la gingivite. Il va donc se programmer une visite chez le dentiste ce qui fait toujours plaisir !
Comme l’a fait remarquer le professeur Holmes, les résultats reçu par le journaliste comporte beaucoup de données intéressantes – mais pas beaucoup d’idées utiles. A l’instar de ce que l’on a pu constater pour d’autres fournisseurs de « génomique personnelle », chaque échantillon reçu par Microba permet d’améliorer sa méthode de prédiction d’éléments interprétés du microbiote. Le service sera lancé au public au début d’année 2017.
Moralité : à moins d’avoir un Holmes sous la main, ces sociétés devront fournir à leurs clients des résultats automatiquement pré-interprétés pour que ces derniers puissent avoir un outil influençant leur mode de vie ou leur gestion de planning-dentiste. En effet, l’inventaire à la Prévert de ce que l’on a dans le ventre n’a que peu de sens et nécessiterait d’être remis en perspective. Cette liste, ces métriques devraient être rattachées à la singularité du client du service pour réellement pouvoir lui être utiles. Un modèle prédictif basé sur une méthode d’apprentissage automatique ne peut suffire ! On comprend que plus Microba va séquencer, plus précis sera son modèle mais son développement futur passera par une meilleure intégration d’un résultat interprétable par le commun des mortels.
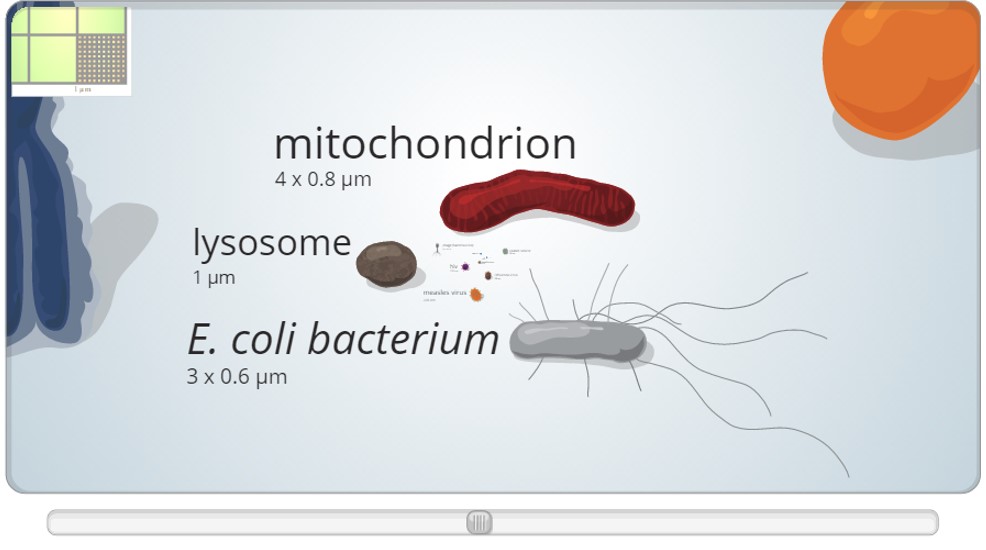
En cliquant sur l’image ci-dessus, vous pourrez vous apercevoir qu’un ribosome fait la même taille qu’un rhinovirus… une infographie très simple mais efficace pour relativiser les « tailles du vivant »
Le premier MOOC, en français, ambitieux, pour tous les botaniques en herbe… du néophyte au plus confirmé. Ce MOOC s’est ouvert le lundi 05 septembre 2016, dans cette première session (il y en aura d’autres), pour une durée de 7 semaines. Des semaines durant lesquelles vous pourrez collectionner des badges synonymes de votre épanouissement culturelle au sujet des plantes. Vous pouvez accéder directement au MOOC de Tela Botanica en cliquant sur la capture d’écran ci-dessous.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages