Dans la forme, cette vidéo (en anglais) se veut humoristique mais le fond est assez sérieux puisqu’il montre ce qui ne devrait pas arriver quand un scientifique effectue une demande (courtoise) de partage de données : Un enchainement d’échanges souvent à la limite de l’absurde reflet d’une situation malheureusement assez commune dans le domaine de la science.
Auteurs : Karen Hanson, Alisa Surkis and Karen Yacobucci

Pour rien vous cacher, il s’agit ici d’un article avec un petit goût d’auto-promotion. En effet, les 3 membres de Biorigami participent aussi activement à une plateforme d’expertise nouvellement créée, cette entité a pour ambition la réalisation de quelques prestations, principalement en génomique haut-débit, mais aussi la participation à des projets nécessitant conseils, expertises et analyses… pour répondre au mieux aux questions biologiques posées. Enchaînons en citant les pages actualités du site internet de Gènes Diffusion : « cette alliance entre les deux structures (ndlr : Gènes Diffusion et l’Institut Pasteur de Lille) a pris le nom de PEGASE (Plateforme d’Expertises Génomiques Appliquées aux Sciences Expérimentales). Elle permet de mutualiser les moyens de la branche R&D génomique de Gènes Diffusion et du laboratoire TAG (Transcriptomic analysis and Applied Genomics) de l’Institut Pasteur de Lille.
La création de cette unité devrait permettre le développement de nouvelles activités, collaborations scientifiques et prestations, dès les prochains mois
Issue de la collaboration entre Gènes Diffusion et l’Institut Pasteur de Lille, PEGASE (Plateforme d’Expertises Génomiques Appliquées aux Sciences Expérimentales) vient de lancer son site web. Cette présence sur le web permettra à la nouvelle structure de faire l’interface avec les différents acteurs de la génomique.
Par ailleurs, le site permettra également de faire partager à la communauté scientifique les différentes expertises en biologie moléculaire et bio-informatique issues des travaux de recherche de l’unité. »
Vous pourrez obtenir plus de précision concernant cette nouvelle plateforme en suivant le lien suivant : www.pegase-biosciences.com.
Pour les plus joueurs d’entre vous, un concours est lancé pour fêter cette naissance, le projet exécutable sur puce 318 de Ion Torrent, sera réalisé par nos soins, gratuitement… pour plus de précisions, nous vous invitons à suivre le lien suivant : concours PEGASE.

Lors d’une réunion le 21 mai dernier à Lille, Lionel Choplin, responsable du programme chaires industrielles venait présenter les ambitions de l’ANR pour associer le secteur privé –le co-financier et bénéficiaire des fruits de la valorisation de la recherche- et le secteur public –le pourvoyeur de cerveaux (de préférence provenant d’une grande université étrangère) et de structure d’accueil. Vous trouverez la présentation en suivant ce lien.. Ce programme est très largement inspiré du PCI canadien (Professeurs-Chercheurs Industriels) du CRSNG (Conseil de Recherches en Sciences Naturelles et en Génie du Canada) qui « offre » trois types de subventions :
- professeur-chercheur industriel principal pour les chercheurs principaux distingués (subvention renouvelable d’une durée de cinq ans);
- professeur-chercheur industriel agrégé pour les chercheurs en début de carrière qui démontrent un potentiel exceptionnel (subvention renouvelable une fois d’une durée de cinq ans);
- professeur-chercheur industriel cadre pour les professionnels exceptionnels de la recherche et développement (subvention non renouvelable d’une durée de cinq ans).
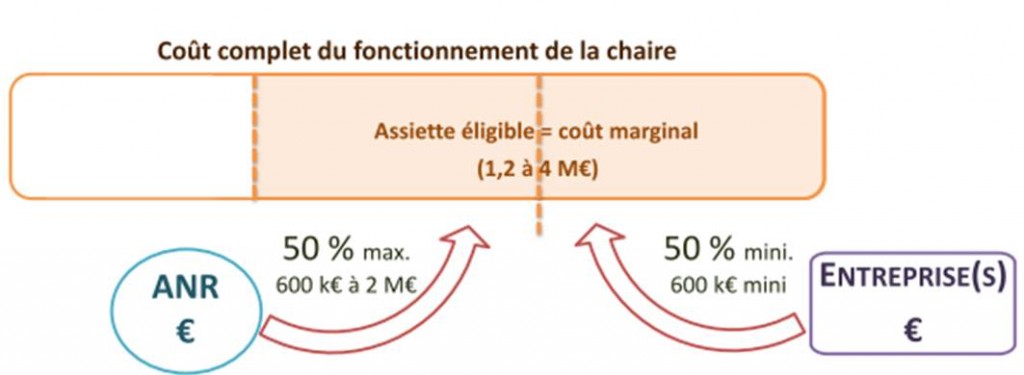
Dans sa version française, la subvention est clairement destinée aux deux premiers cas mentionnés ci-dessus. Ainsi la relation industriels-organismes de recherche devient plus monodirectionnelle. L’industriel lève des fonds (le ticket d’entrée étant de 600 k€ minimum sur 4 ans), l’ANR abonde d’autant (la stratégie du quitte ou double), la structure d’accueil hébergeant le titulaire de la chaire assure son bon fonctionnement (sachant que l’investissement en gros équipement est limité à 20 % de l’assiette). Le titulaire de la chaire ainsi que la structure d’accueil bénéficient de ce financement, l’industriel, quant à lui, aura un retour en terme d’image, de retombées sous forme de Propriété Intellectuelle et surtout en terme de connaissances sur un sujet qui est censé impacter sa croissance.
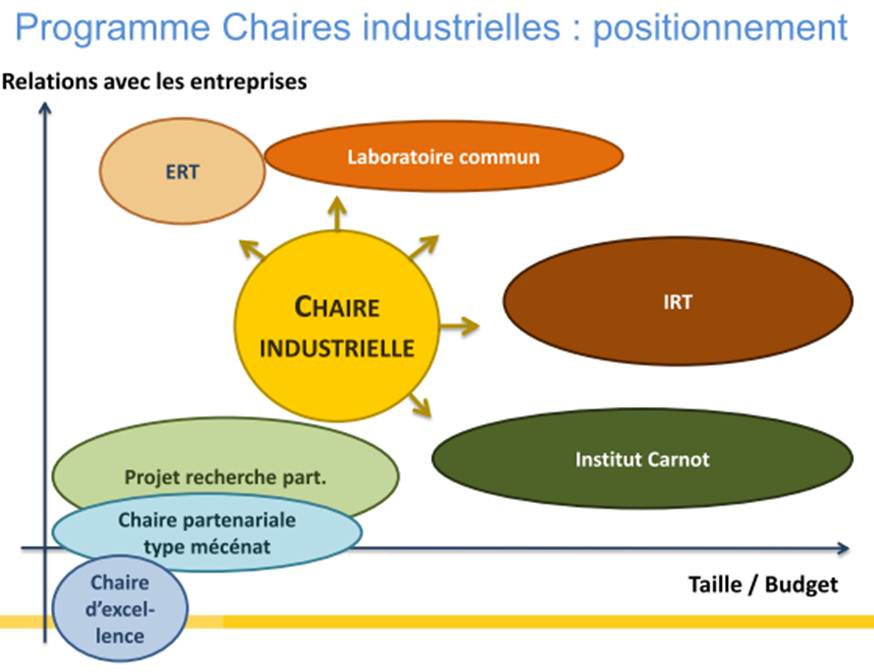
Le schéma ci-dessous reprend le positionnement relatif de la chaire industrielle par rapport aux autres programmes de l’ANR.
Par ce type de programmes, l’ANR vise plusieurs objectifs :
– Créer une dynamique à partir d’une offre de compétences et d’expertise en adéquation avec les besoins des acteurs du monde socio-économique
– Organiser les recherches dans une logique d’exploitation des résultats de recherche
– Insuffler au sein de l’établissement d’accueil une dynamique de recrutement de qualité dans un cadre de développement concerté fort
– Permettre un échange en temps réel de technologies et de savoir-faire entre les acteurs publics et privés de la recherche
– Assurer des retombées en termes d’image, résultant de l’accueil d’éminents enseignants-chercheurs au plan international
– Participation des acteurs publics au développement de produits innovants
– Mise en place de modèles économiques construits sur l’économie de la connaissance
Ce programme est comme beaucoup, critiquable, certainement imparfait mais il a le mérite d’afficher la volonté étatique de concilier secteur privé et secteur public, les industriels et les structures d’accueil… dans une relation plus équilibrée que de coutume afin que ces deux acteurs tirent bénéfice de cette relation. Rappelons ici que même aux États-Unis, souvent cités en exemple, les partenariats universités-entreprises restent modestes : 6 % des dépenses réalisées en 1999, au plus fort de la bulle Internet, depuis, cette part est redescendue à 4 %.
Il convient tout d’abord de distinguer deux types de bioinformaticiens :
-Ceux qui sont intégrés dans une unité de recherche : ils sont souvent spécialisés sur une thématique ou même un projet précis : généralement isolés, non permanent, impliqués et informés des problématiques biologiques
-Ceux qui font partie d’une unité de bioinformatique : en émulation avec d’autres bioinformaticiens, moins précaires, plus loin des problématiques biologiques
Ce document se veut une libre opinion sur la bonne intégration d’un, ou d’une équipe, de bioinformaticiens au sein d’un groupe de recherche et les clés pour la bonne réussite de celle-ci. Il est en grande partie inspiré de l’éditorial « On the organization of bioinformatics core services in biology-based research institutes » agrémenté de commentaires plus personnels.
Pour le bien-être interne et externe d’une plateforme de bioinformatique, il convient de :
-Bien séparer les tâches dîtes de prestation et les activités de recherches.
-Scinder les sujets traités par tâches bien définies : par exemple, support en développement et maintenance de base de données, analyse statistique de données haut-débit, support pour le déploiement d’applications sur environnement HPC, analyse de donnée NGS…
-Mettre en place un comité d’utilisateurs incluant des utilisateurs biologistes/bioanalystes chargés d’établir les priorités
-Mettre en place des outils pour suivre l’activité bionformatique de la façon la plus transparente possible
-Mettre en place au maximum des projets transversaux pour favoriser l’interface entre biologiste et bioinformaticiens, les interactions pluridisciplinaires sont les plus enrichissantes et souvent les plus productives. Comme toutes disciplines, la bioinformatique peut devenir endogame, particulièrement dans ou le cadre où l’on parle de bioinformatique comme une science à part entière, et non comme un outil au service de problématiques biologiques : une équipe de bioinformatique peut très bien travailler sur des sujets « porteurs » (donc plus facile à financer) sans interagir, ou seulement sporadiquement, avec des biologistes pour valider le bien-fondé du travail. C’est une des dérives du système d’évaluation actuel, pour plus d’information, lire la libre opinion publiée dans ce blog : Chemins de traverses : la recherche d’excellence ou quand les mots perdent leur sens.
-Mettre en place des formations pour les biologistes : collaborer avec des personnes connaissant un minimum les problématiques bioinformatiques est plus efficace
-Dans le même esprit, nommer un bioinformaticien responsable pour le support utilisateurs pour assister les biologistes dans leurs tâches quotidiennes de bioanalyse est très utile pour entretenir de bonne relation avec les laboratoires de recherche
-Pour les bioinformaticiens intégrés dans une unité, il est primordial de trouver des structures permettant de rencontrer et de partager avec d’autres bioinformaticiens, à défaut, passer quelques heures par semaine à effectuer de la veille technologique peut être un palliatif.
Pour conclure, une équipe de bioinformaticiens doit communiquer un maximum avec leurs partenaires biologistes afin de favoriser une saine interaction, un bioinformaticien, seul, entourer de biologistes se doit de rester à la page, idéalement en partageant et en communiquant sur ces projets à d’autres bioinformaticiens.
L’accroissement des capacités technologiques en biologie moléculaire à pour conséquence la possibilité de séquencer/génotyper mais aussi stocker toujours plus de données constituant une manne d’informations sans limite pour qui sait en extraire du sens. C’est le travail du bioinformaticien de proposer à la fois un support technique et analytique au biologiste en même temps que la capacité en terme de ressources informatiques pour répondre aux besoins.
Ce premier post sur la place du bioinformaticien se focalise sur l’interaction avec le biologiste au sein d’un projet.
On peut dissocier un projet en trois phases :
– La préparation : identifier la question biologique, étudier la faisabilité et les coûts, mettre en place la méthodologie…
– La réalisation : génération des données biologiques et analyse bioinformatique
– La finalisation : interprétation des résultats, mise en production ou introduction d’une nouvelle question biologique
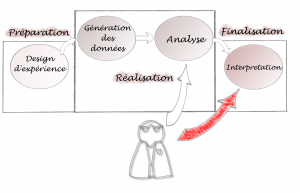
Le schéma ci-dessous montre un cheminement possible de l’intervention bioinformatique :
L’intervention du bioinformaticien est ici minimisée : il n’intervient qu’à partir de la phase analytique et dans l’interprétation des résultats (pour aider le biologiste à critiquer les résultats générés). Ceci pose plusieurs problèmes : les outils bioinformatiques doivent s’adapter, à tout prix, à la méthodologie mise en place précédemment. Si on constate que les méthodes d’analyses doivent être modifiées une fois la génération de données effectuées, la bioinformatique peut devenir ce qu’on appelle communément un goulot d’étranglement dans le projet…
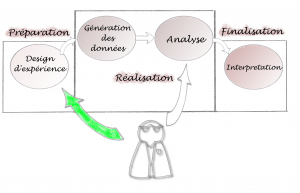
Au contraire, Le schéma ci-dessous nous parait plus judicieux :
Ici, l’intervention bioinformatique est optimisée : le bioinformaticien est totalement impliqué dans la préparation du projet. Et, au contraire, en fin de projet, les interventions sont minimisées car la méthodologie mise en place est adaptée pour une bonne compréhension par le biologiste. L’idéal et de fournir les outils nécessaires pour que le biologistes puisse lui-même exécuter ses analyses.
Pour résumer, l’ interaction doit se faire en amont et en aval de l’analyse et l’on se doit de minimiser les interventions pendant la phase d’analyse. En aval pour conseiller sur les méthodologies à utiliser, en amont pour contrôler les erreurs éventuelles rencontrées pendant la phase d’analyse (exécuter par le biologiste).
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages