Le bien beau document que voilà ! Se repérer dans toutes les options de préparation des échantillons à séquencer, options qui sont fonctions de l’origine et de l’application que l’on souhaite mettre en oeuvre par l’intermédiaire du séquençage, peut être un réel casse-tête que ce document souhaite simplifier ici.
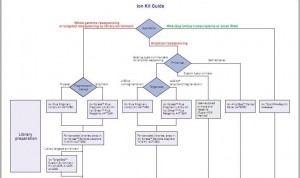 Cet arbre de décision s’étoffera très rapidement : d’autres branches pousseront avec les protocoles « mate pair » & « paired end », les nouvelles indexations… et surtout l’arrivée du kit permettant de séquencer 400 b… Peut être que les branches portant les options 100 b seront, quant à elles, pour l’occasion élaguées.
Cet arbre de décision s’étoffera très rapidement : d’autres branches pousseront avec les protocoles « mate pair » & « paired end », les nouvelles indexations… et surtout l’arrivée du kit permettant de séquencer 400 b… Peut être que les branches portant les options 100 b seront, quant à elles, pour l’occasion élaguées.
Ce document, et beaucoup d’autres, est disponible sur le site de la communauté Ion Torrent.
 Les scientifiques seraient ils des parangons de vertu ? Comment et pourquoi les imposteurs travestissent la vérité en la parfumant de sciences et de techno-science ? C’est à ces questions que répond clairement Michel de Pracontal dans un ouvrage (ici ré-édité) publié, pour la première fois, il y a plus de 20 ans. Dans sa version de 2001, en quelques 332 pages le journaliste scientifique oeuvrant pour le Nouvel Observateur et Mediapart (son blog est totalement recommandable) réalise un manuel de référence pour tout zététicien qui se respecte. Certes, ce manuel abordant Rika Zaraï, l’astrologie, l’homéopathie, l’affaire Baltimore, les oeuvres de moult scientifiques peu vertueux inventeurs de données frelatées tel un Cyril Burt, d’ imposteurs en herbe et de gourous scientistes avides, est parsemé d’humour grinçant, sardonique mais il est avant tout, même avec quelques maladresses, une ode à l’esprit critique utile à tous. Le livre est structuré en manuel avec un exercice en fin de chapitre permettant d’aiguiser son esprit critique et de se détacher de l’autorité scientifique qui va de soi… Les cas abordés sont pléthoriques, couvrent tout le spectre de l’escroquerie intellectuelle et scientifique… le lecteur peut être troublé ou amusé de voir mis sur le même plan l’astrologue et le physicien peu scrupuleux. Or l’origine de la fraude et la manière dont elle est crédibilisée ne sont pas les mêmes… le profane peut s’y perdre un peu.
Les scientifiques seraient ils des parangons de vertu ? Comment et pourquoi les imposteurs travestissent la vérité en la parfumant de sciences et de techno-science ? C’est à ces questions que répond clairement Michel de Pracontal dans un ouvrage (ici ré-édité) publié, pour la première fois, il y a plus de 20 ans. Dans sa version de 2001, en quelques 332 pages le journaliste scientifique oeuvrant pour le Nouvel Observateur et Mediapart (son blog est totalement recommandable) réalise un manuel de référence pour tout zététicien qui se respecte. Certes, ce manuel abordant Rika Zaraï, l’astrologie, l’homéopathie, l’affaire Baltimore, les oeuvres de moult scientifiques peu vertueux inventeurs de données frelatées tel un Cyril Burt, d’ imposteurs en herbe et de gourous scientistes avides, est parsemé d’humour grinçant, sardonique mais il est avant tout, même avec quelques maladresses, une ode à l’esprit critique utile à tous. Le livre est structuré en manuel avec un exercice en fin de chapitre permettant d’aiguiser son esprit critique et de se détacher de l’autorité scientifique qui va de soi… Les cas abordés sont pléthoriques, couvrent tout le spectre de l’escroquerie intellectuelle et scientifique… le lecteur peut être troublé ou amusé de voir mis sur le même plan l’astrologue et le physicien peu scrupuleux. Or l’origine de la fraude et la manière dont elle est crédibilisée ne sont pas les mêmes… le profane peut s’y perdre un peu.
Voici le quatrième de couverture : « Plus de 50 % des Français croient à la transmission de pensée et à la guérison par magnétiseur, près de la moitié fait confiance à l’astrologie, 35 % aux rêves prémonitoires. De l’horoscope sur Internet au phénomène « X-Files », de la mémoire de l’eau aux « paramédecines », de la réincarnation aux « expériences NDE », la patrie de Descartes se passionne plus que jamais pour l’irrationnel. Le progrès des sciences et des techniques s’accompagne d’un essor des pseudo-sciences et des fraudes scientifiques. Des savants renommés accréditent la téléportation et le voyage dans le temps. Des autodidactes inventifs proposent des théories « alternatives » à celles de Darwin et d’Einstein.
Loin d’être marginale, l’imposture scientifique est devenue une nouvelle norme intellectuelle. Baigné d’ondes positives, planant au-dessus des basses arguties de la raison, l’homme nouveau du xxte siècle goûtera-t-il le vrai bonheur ? Pour s’y préparer, voici l’indispensable manuel du pipeau et de la baliverne. Cet ouvrage vivant et didactique remet (totalement) à jour sa première édition, publiée en 1986. L’honnête citoyen comme l’apprenti charlatan y trouveront tous les conseils utiles, illustrés d’exemples concrets et d’anecdotes captivantes.
Le lecteur s’instruira en s’amusant, et apprendra comment départager le vrai du faux dans une culture surmédiatisée, régie par la dictature du marché et de l’audimat. Quand l’impact du message finit par l’emporter sur son contenu, le réel s’affaiblit. L’univers orwellien de 1984, où le charbon est blanc et où deux et deux font cinq, a cessé d’être une pure fiction. Contre un tel danger, la dernière arme est peut-être l’humour
Michel de Pracontal, 46 ans, est titulaire dune maîtrise de mathématiques et d’un doctorat en sciences de l’information sur la vulgarisation scientifique. Journaliste scientifique depuis vingt-deux ans (depuis 1990 au Nouvel Observateur), il est l’auteur de Les Mystères de la mémoire de l’eau (La Découverte, 1990) et de La Guerre du tabac (Fayard, 1998). » (table des matières).
Ce manuel est à placer entre le « petit cours d’autodéfense intellectuelle » de Normand Baillargeon et « gourous, sorciers et savants » de Henri Broch, deux ouvrages qui feront l’objet de chroniques futures.
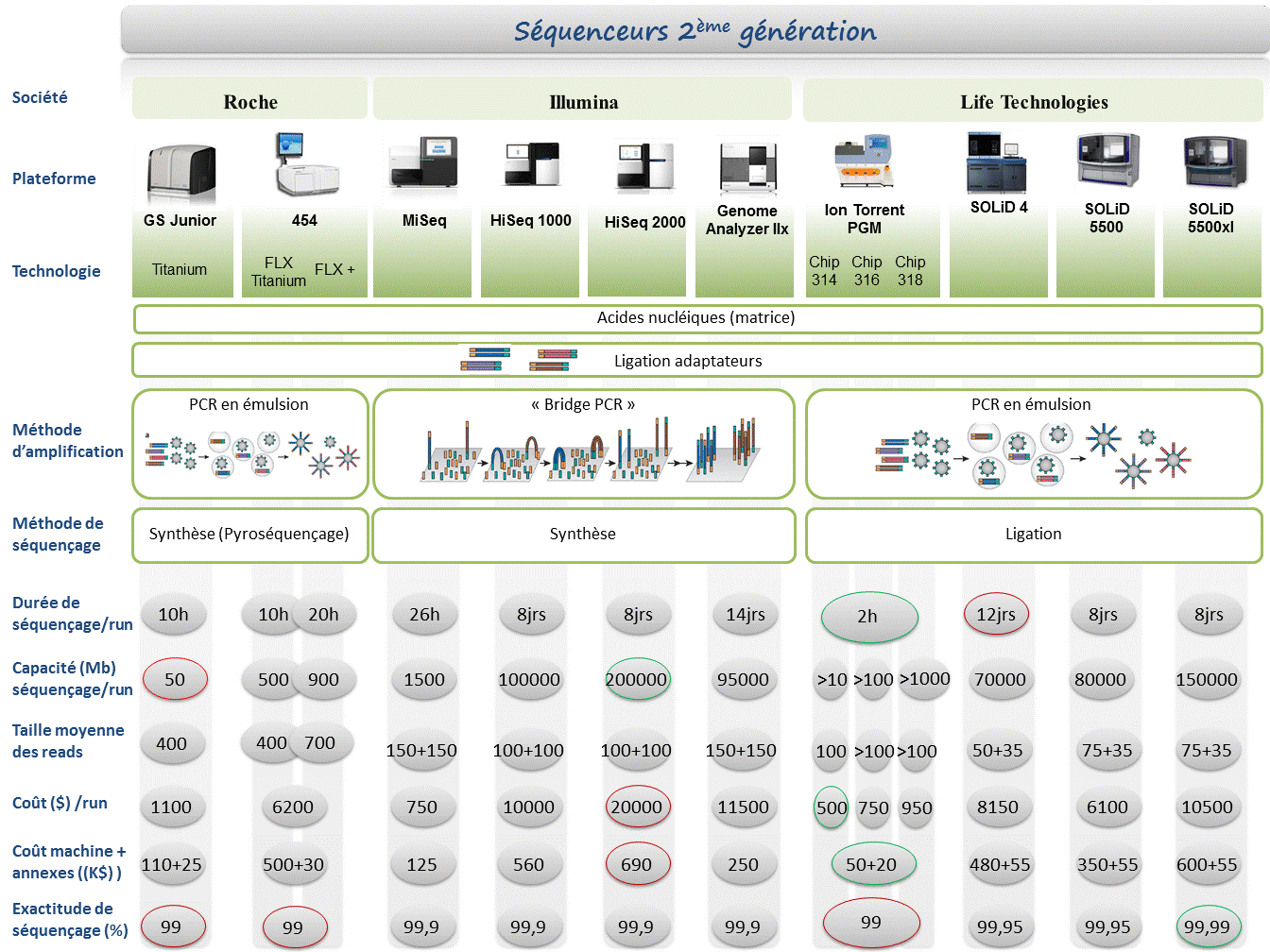
Le terme « Nouvelle génération de séquençage à haut-débit » ( ou « Next generation sequencing » ) regroupe l’ensemble des technologies ou plateformes de séquençage développées depuis 2005 par quelques sociétés de biotechnologies.
L’objectif de cet article est de proposer de manière synthétique, un tour d’horizon des différents principes et caractéristiques de ces nouveaux outils et ainsi fournir quelques orientations et solutions techniques en réponses à des questions biologiques.
La position actuelle dans laquelle nous nous trouvons, entre la commercialisation de certains séquenceurs et ceux en cours de développement, est caractéristique d’une période charnière dissociant les technologies à haut débit dites de 2ème génération qui requièrent une étape d’amplification des molécules d’ADN en amont du décodage, de celles dites de 3ème génération permettant le décryptage direct d’une seule molécule d’ADN. Cette dernière catégorie fera à elle seule l’objet d’un prochain article.
Le marché des séquenceurs de 2éme génération est couvert par 3 grand groupes que sont Roche, Illumina et Life Technologies ayant respectivement proposés de manière successive, leur première plateforme à savoir le 454, le Genome Analyser et enfin le SOLiD. Depuis, le marché s’est étoffé proposant un panel de technologies au principe et caractéristiques propres telles qu’elles sont mentionnées ci dessous. A noter que parmi ce panel, le PGM, Ion torrent est le seul a connaitre une évolution constante en terme de capacités de séquençage (10Mb – reads de 100b – Juin 2011 / 100Mb – reads de 100b – Sept 2011 / 100Mb – reads 200b – Nov 2011 / 1Gb – Jan 2012 )
Chaque plateforme possède ses avantages et inconvénients et nombreuses sont celles configurées pour répondre à de nombreuses approches « omics », dans certaines limites. Il s’agira de faire un choix technologique selon les champs d’applications souhaités.
De manière générale, le type d’organisme étudié prédéterminera la technologie à employer. La notion de profondeur est récurrente à chaque application et dans l’objectif d’un reséquençage, le choix de la plateforme peut être identifié, de manière simplifiée, sur la base d’un calcul rapide ( P = N / L où P: Profondeur, N: Nombre des nucléotides totaux des reads, L: Taille du génome étudié).
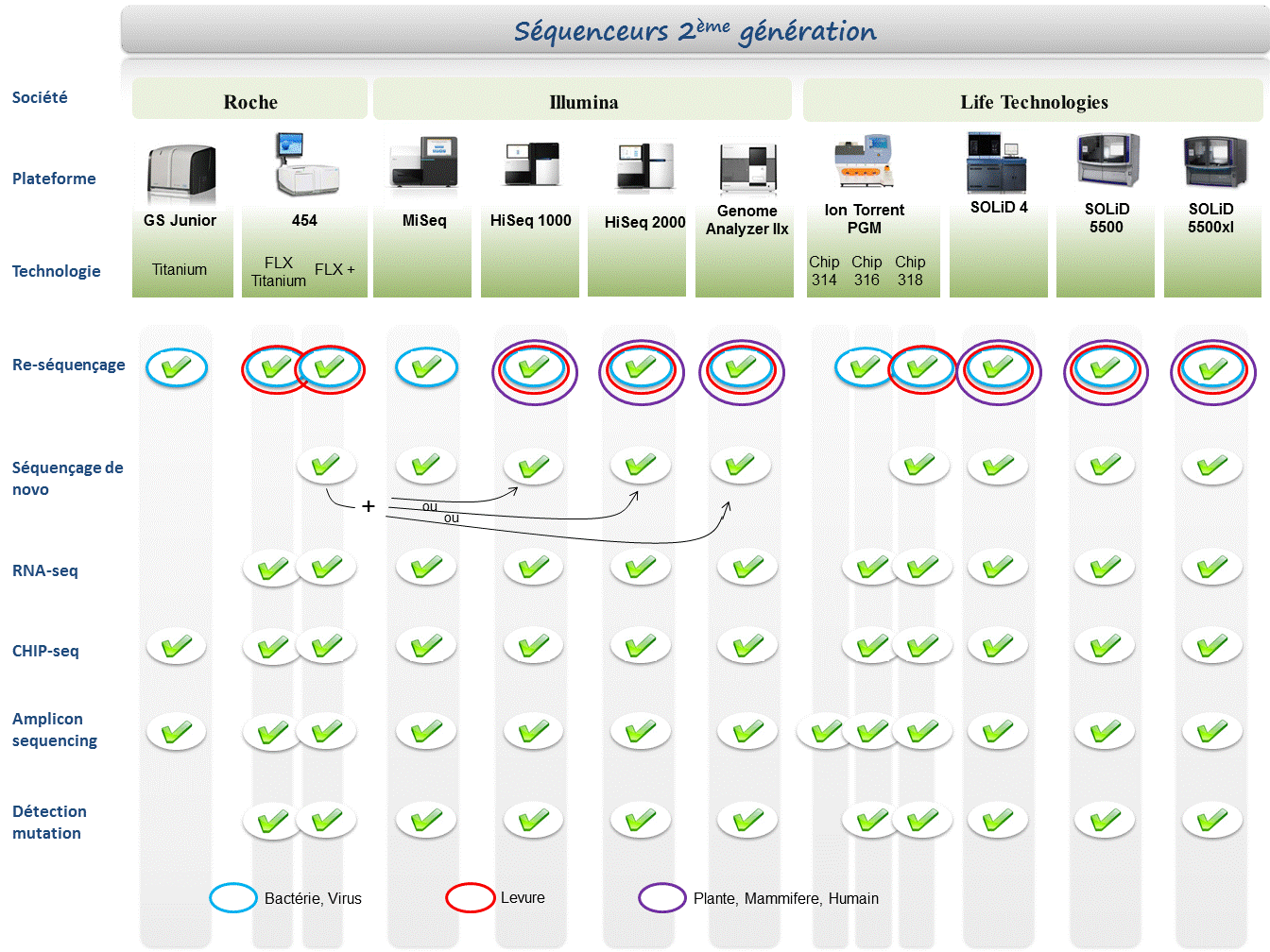
Concernant les séquenceurs de 2ème génération, le séquençage de novo est une application mentionnée chez de nombreux fournisseurs (cf le tableau ci-dessous). Toutefois, l’association de deux technologies générant à la fois des reads longs (type 454, Roche) et une profondeur conséquente (type GAIIx, Illumina) palliant aux problèmes liés aux homopolymères et erreurs de séquençage, est préconisée (Au cours de l’article à venir sur les séquenceurs de 3ème génération, nous aborderons les plateformes davantage configurées pour cette application).
Ce paramètre de profondeur sera également à prendre en considération pour les champs d’applications incluant la notion d’analyse quantitative (RNAseq, ChIPseq, …). Si la profondeur permet d’atténuer les erreurs de séquençage, il reste néanmoins préférable de s’orienter vers des technologies à Q30 minimum (1 erreur sur 1000) pour la détection de SNPs.
Selon les technologies évoquées ci-dessus, les caractéristiques et champs d’applications ont évolués. Aussi, je vous propose de retrouver l’ensemble de ces informations actualisées en cliquant sur ce lien.
L’ensemble des informations sont détaillées dans l’article mentionné ci-dessous:
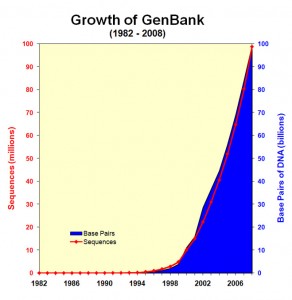
Constat : le séquençage, un générateur fantastique de données
L’explosion de la génération de données génomiques (Figure 1.0) et l’hétérogénéité de ces données entraînent inévitablement un accroissement de l’écart entre les données, les connaissances et l’information que l’on peut en extraire (Figure 1.1).
Le développement d’outils bioinformatiques permet de répondre à l’analyse de données dans l’ensemble des domaines de la biologie, mais le besoin en formalisation (des formats, des processus, des architectures matérielles…) est primordial si l’on veut éviter les problèmes d’incompatibilité et de réutilisation des solutions.
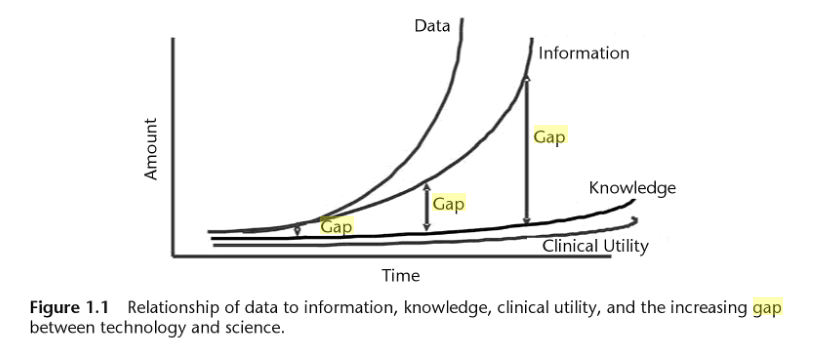
Figure 1.1 : Issu de "Biomedical informatics in translational research" Par Hai Hu,Richard J. Mural,Michael N. Liebman
En conséquence, afin d’obtenir des informations interprétables biologiquement à partir de ces données, la mise en place de processus d’analyse va de pair avec l’utilisation de logiciels à jour, efficaces pour la gestion de données à grande échelle.
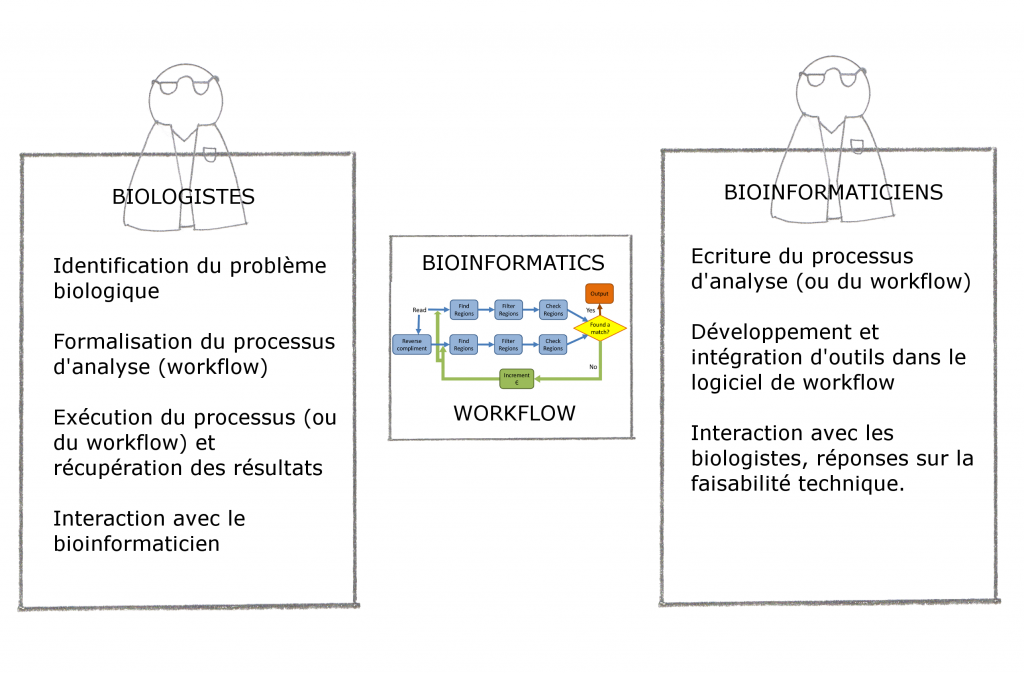
En outre, les logiciels doivent aussi simplifier cette gestion de données et fournir des outils d’analyses bioinformatiques accessibles mais aussi reproductibles pour tous types d’utilisateurs – dont les biologistes eux-mêmes. C’est dans ce cadre que s’insèrent l’automatisation de processus (ou « Workflow ») et le développement de « Workflow management system », ou logiciel de Workflow, pour la bioinformatique.
Workflow ou automatisation de processus : définition
Le logiciel de Workflow s’inscrit aussi dans une logique de pérennisation des processus analytiques, qui a pour but de sortir de la logique « projet » (dans le sens ponctuel) en créant des processus d’analyses génériques.
Un logiciel de Workflow est un outil permettant d’exécuter un ensemble de processus de façon automatique. Ces « pipelines » sont très présents en bioinformatique (à défaut d’être tres utilisés) car ils permettent aux chercheurs en biologie d’analyser leurs données (issues de séquencages, génotypages) de façon relativement transparente et (quasiment) sans l’aide d’informaticiens (denrées rares dans la recherche).
Toutefois, il convient de distinguer deux sortes de logiciel de Workflow :
– Les logiciels de Workflow qui permettent aux chercheurs de manipuler leurs données et exécuter leurs analyses sans posséder de connaissances en écriture de scripts ou en bases de données. Les données sont rapatriées au sein du logiciel de Workflow, permettant l’exécution d’un ensemble de tâches, à travers des modules pré-installés. En séquençage, le Workflow permet de convertir des séquences en formats divers, les filtrer ou les assembler… Le logiciel de Workflow ISYS (2001), BioMOBY, Taverna et plus particulièrement Galaxy (qui fera l’objet d’un prochain post ici) entrent dans cette catégorie.
– Les logiciels de Workflow qui assurent un accès direct à des composants (installés sur le serveur) et/ou aux données génomiques sans passer par un rapatriement préalable des données. WildFire, Pegasys ou Ergatis (ce dernier sera décrit dans un prochain post) font partie de cette catégorie. De manière générale ces logiciels de Workflow sont plus difficiles à prendre en main mais sont évidemment plus flexibles.
Pour résumer, quel que soit le logiciel de Workflow utilisé il permet :
– D’automatiser des processus d’analyse (idéalement répétitifs) en les reliant dans un pipeline
– De lancer des analyses sur des architectures matérielles complexes telles des grilles de calculs (voir l’article sur Grisbi) ou des serveurs
– De formaliser le processus d’analyse en vue d’une publication scientifique
Philosophie générale : interaction et exemple de workflow
Pour finir cette introduction, nous vous proposons deux schémas : la place du logiciel Workflow et un exemple concret.
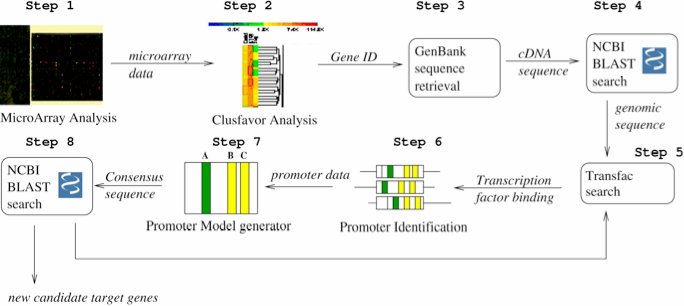
- Figure 1.3 : Exemple de conception d’un Workflow d’identification de promoteurs (extrait de « Accelerating the scientific exploration process with scientific workflows« )
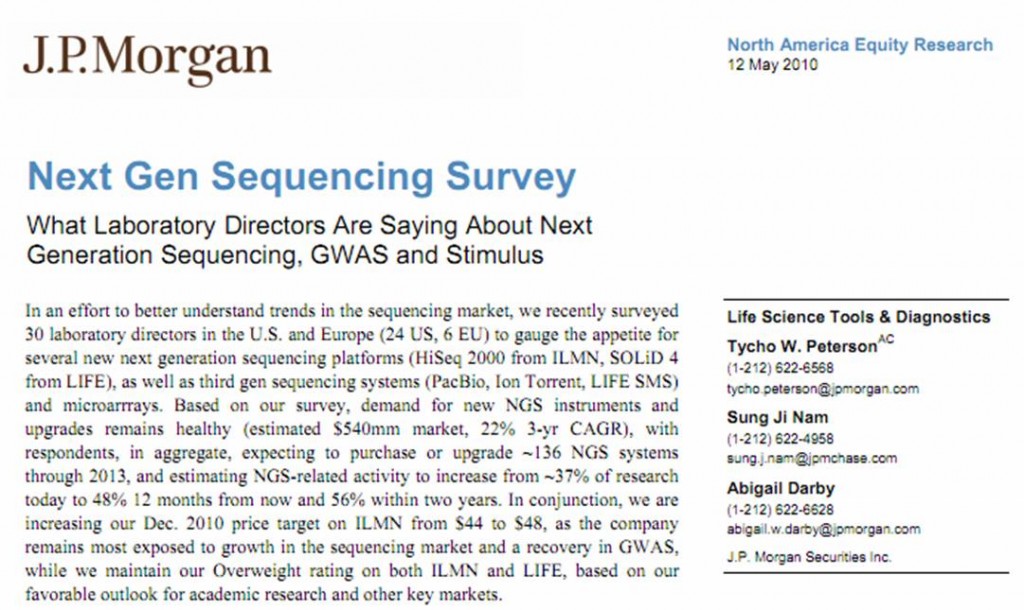
Voici un excellent rapport (toujours d’actualité) réalisé par J.P. Morgan, une holding leader dans la banque d’investissement. Ce rapport traite de la part prise par les différentes applications NGS, il compare les différents séquenceurs haut-débit… cette enquête a été réalisée en interrogeant 30 laboratoires (dont 24 Américains), 50 % de ces laboratoires sont publics. ..
Ce rapport permet d’entrevoir ce que seront les prochaines demandes de financements des laboratoires d’une part, d’autre part, il permet d’entrevoir les possibilités offertes par ce type de technologies encore très consommatrices de fonds et de temps humain. Les technologies disponibles sont de plus en plus diverses… Pour beaucoup de laboratoires il s’agira d’effectuer le choix technologique le plus adapté à leur champ d’application. Avec un petit étonnement, on apprend que l’application la plus développée au sein des laboratoires, concerne l’expression de gènes (mRNA expression profiling)… le séquençage de novo n’arrivant qu’en 7ème place des applications les plus développées.
(le rapport est disponible en cliquant sur l’image ci-dessus)
 De retour sur les chemins de traverses pour aborder un sujet qui est finalement assez peu développé dans le monde scientifique latin : la fraude (le puritanisme anglo-saxon oblige à s’intéresser de près à tout manquement à la morale…scientifique). Evidemment il semble utile de définir les contours de ce qui est considéré comme une fraude scientifique. Le chercheur dans le champ scientifique est en quête de vérité en vue de permettre une accumulation de savoir fiable disponible pour l’ensemble de la communauté humaine. Tout manquement (volontaire ?) à l’obligation de loyauté face à la vérité scientifique peut être considéré comme frauduleux. Ainsi, ces fraudes majeures peuvent être classées en trois grandes catégories :
De retour sur les chemins de traverses pour aborder un sujet qui est finalement assez peu développé dans le monde scientifique latin : la fraude (le puritanisme anglo-saxon oblige à s’intéresser de près à tout manquement à la morale…scientifique). Evidemment il semble utile de définir les contours de ce qui est considéré comme une fraude scientifique. Le chercheur dans le champ scientifique est en quête de vérité en vue de permettre une accumulation de savoir fiable disponible pour l’ensemble de la communauté humaine. Tout manquement (volontaire ?) à l’obligation de loyauté face à la vérité scientifique peut être considéré comme frauduleux. Ainsi, ces fraudes majeures peuvent être classées en trois grandes catégories :
– la fabrication de données
– la falsification de données
– le plagiat
Ces catégories constituent la zone « noire », avec une prévalence certainement faible, un grand nombre de faits peuvent quant à eux, être plus difficiles à classifier et caractériser, c’est ce que l’on classe dans la zone « grise ». A titre d’exemples, les publications « salami », les co-signatures honorifiques, les soumissions multiples semblent quant à eux des actes beaucoup plus fréquents.
Il existe assez peu d’études descriptives et analytiques sur la thématique de la fraude scientifique. Martinson et al. (Nature, 2005) est une des rares études interrogeant un nombre significatif de chercheurs (distinguant les jeunes chercheurs des chercheurs confirmés). Avec cette publication que nous vous laissons le soin de lire, se trouve légèrement étendue la notion de fraude scientifique. Ainsi tombe sous la qualification de comportement frauduleux, les faits et actes suivants :
– la falsification de données
– l’ignorance des principaux aspects liés à la vie humaine (définition un peu floue)
– la non divulgation des conséquences liées à la recherche privée en lien avec un produit commercial(isable)
– les éventuelles connivences entre étudiants, sujets de recherche ou client
– l’utilisation d’idées qui ne sont pas de soi sans en avoir obtenu la permission ou sans nommer la paternité de celles-ci
– l’utilisation non autorisée d’informations confidentielles dans le cadre de ses propres recherches
– omettre de présenter des données qui contredisent ses propres recherches antérieures
– contourner certains aspects mineurs des exigences liées à l’homme comme sujet d’étude
– la surabondance d’utilisation de données erronées ou d’interprétations douteuses de données
– la modification de la conception, de la méthodologie ou des résultats d’une étude en réponse à la pression provenant d’une source de financement
– éditer les mêmes données ou les mêmes résultats dans deux ou plusieurs publications
– la rétention d’informations (particulièrement concernant l’aspect méthodologique d’une publication)
– omettre les observations ou les points de données paraissant de prime abord fallacieux (cela simplifie la démonstration finalement)
– la tenue inadéquate des dossiers relatifs aux projets de recherche (le cahier de laboratoire comme « un excellent atout juridique pour prouver une antériorité voire être une très bonne base pour la rédaction d’un brevet »)
Avant Martinson, Larivée et Baruffaldi (1993) et Swazey (1993) ont décrit, étudié et calculé des prévalences de cas de fraudes scientifiques. Larivée après avoir étudié 1000 articles de cinq grands domaines scientifiques (sciences de la terre, sciences pures, sciences humaines,sciences de la vie et sciences de la santé) détecte 230 cas de fraudes (rien que ça).
Il est temps d’aborder brièvement les raisons pour lesquelles le cherche se corrompt en flirtant avec la fraude : pression, compétitivité, besoin de performance (finalement les mêmes raisons qui poussent un cycliste à prendre des substances améliorant ses performances).
Quelques articles font état de l’exception française concernant la fraude scientifique. Souvent, dans notre pays ce problème donne lieu à des bafouilles peu audibles, peu étayées… un exemple de cet état de fait peut être porté par le document sonore d’une intervention d’Alain Prochiantz au Collège de France (autant demander à un cyclisme de faire une conférence sur le dopage).
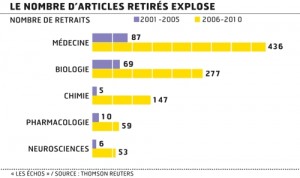
Les exemples de fraudes scientifiques sont malgré tout à la fois légions, les exemples de collusions aussi. Quelques articles aux graphes repris de multiples fois (l’un des plus utilisé est présenté ci-contre) tendent à démontrer que le nombre d’article retirés explose sans réellement chercher à comprendre si ceux-ci sont retirés du fait de l’efficacité de la censure des comités de lecture ou si les chercheurs fraudeurs amateurs sont de plus nombreux à éprouver le système…
Les études où les conclusions sont dictées par l’inclination d’un chercheur pour son financier sont très fréquentes dans la littérature et certainement sur-représentées sur internet (nous pouvons exhumer l’article de Nature de 1988, Human basophil degranulation triggered by very dilute antiserum against IgE (abusivement associée à « la mémoire de l’eau » cet article d’un chercheur financé par Boiron, crédite en partie le modèle de dilution pratiquée en homéopathie).
Les chartes d’éthique telles que celle de l’Institut Pasteur à Paris constituent un garde-fou nécessaire mais insuffisant. En effet, ce type de chartes sont incitatives rarement coercitives. Finalement, face à la possibilité de tricher avec la vérité, le chercheur est face à sa conscience.
La fraude scientifique peut avoir des conséquences directes, en effet, la réputation des chercheurs porteurs d’un projet est en lien direct pour les bailleurs de fonds (particulièrement pour ce qui concerne les fonds publics) avec la qualité de leur propre recherche. Le plus grave sont les conséquences de la fraude sur la science elle-même, l’exemple cité par Serge Larivée est relativement saisissant : « la fraude peut en outre affecter le travail d’autres chercheurs qui auraient construit tout un corpus de connaissances à partir de données frauduleuses. Le cas relativement récent de Gupta est à cet égard exemplaire. Avec 124 autres chercheurs, ce paléontologue de l’Université de Chandigarh, au Penjab, a publié de 1969 à 1988 près de 450 articles sur l’étude des fossiles de l’Himalaya, dont 300 sont soit truffés d’anomalies et d’invraisemblances, soit de pures fictions. Certains soutiennent que cette fraude porte peu à conséquence dans la mesure où la plupart des géologues spécialistes de l’Himalaya ne tenaient plus compte des publications de Gupta depuis belle lurette. Par contre, les chercheurs qui avaient intégré de bonne foi les «découvertes» de Gupta dans leur corpus de connaissances ont été lourdement escroqués puisqu’ils sont maintenant en possession de données inutilisables. » Bien évidemment, une autre conséquence de ce type de fraudes, trouvant échos à raison dans les médias, ont des conséquence sur la santé humaine. Imaginons (toute ressemblance avec un épisode faisant intervenir un médicament du laboratoire Servier est fortuite) que des scientifiques soient amenés à cautionner frauduleusement un médicament en masquant la toxicité d’une molécule… imaginons que des études sur l’incidence des ondes émanant de téléphones sur la santé humaine soient financées par un fabriquant de téléphones portables (à ce jour une dizaine d’études de ce type sont recensées)… le conflit d’intérêt étant bien entendu au cœur de nombreuses fraudes de grande ampleur.
Le scientifique face à ce type de choix : négocier sa carrière ou atteindre la vérité (ce pourquoi il fait ce métier) peut se corrompre en oubliant que l’on ne négocie pas avec la vérité qui a une exigence de sincérité. L’exigence prime sur l’excellence !
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages