Cette article fait suite à notre post sur l’intervention du professeur Arnaud Fontanet de l’Institut Pasteur sur le Coronavirus COVID-19.
Dans sa présentation, le professeur Fontanet renvoie vers trois sites web qui permettent de mieux comprendre le coronavirus.
Chacun dans leur contexte (observation/simulation/étude), ces sites montre la rapidité avec laquelle les chercheurs peuvent développer des outils bioinformatiques de data visualisation pertinents pour la communauté.
Ceci étant bien sur rendu possible à partir du moment où le partage de données épidémiologiques, génétiques, génomiques (…) est effectué.
Pour observer :
Coronavirus COVID-19 Global Cases by Johns Hopkins CSSE
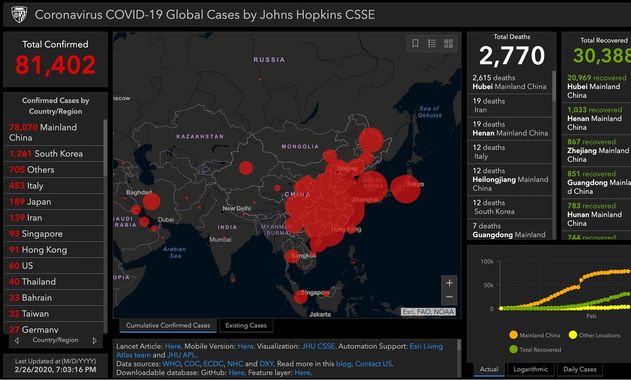
L’université John Hopkins maintient une carte avec des données en temps réel sur le nombre de patients diagnostiqués avec le nouveau coronavirus, le nombre de patients décédés et le nombre de patients guéris. Ces chiffres sont basés sur des informations provenant, entre autres, de l’Organisation mondiale de la santé (OMS) et du Centre européen de prévention et de contrôle des maladies (ECDC). Il peut y avoir de légères différences dans les chiffres réels .
Pour connaître les derniers chiffres confirmés, nous renvoyons aux sites web de l’OMS et de l’ECDC
Github – entrepôt de données : https://github.com/CSSEGISandData/COVID-19
Pour anticiper :
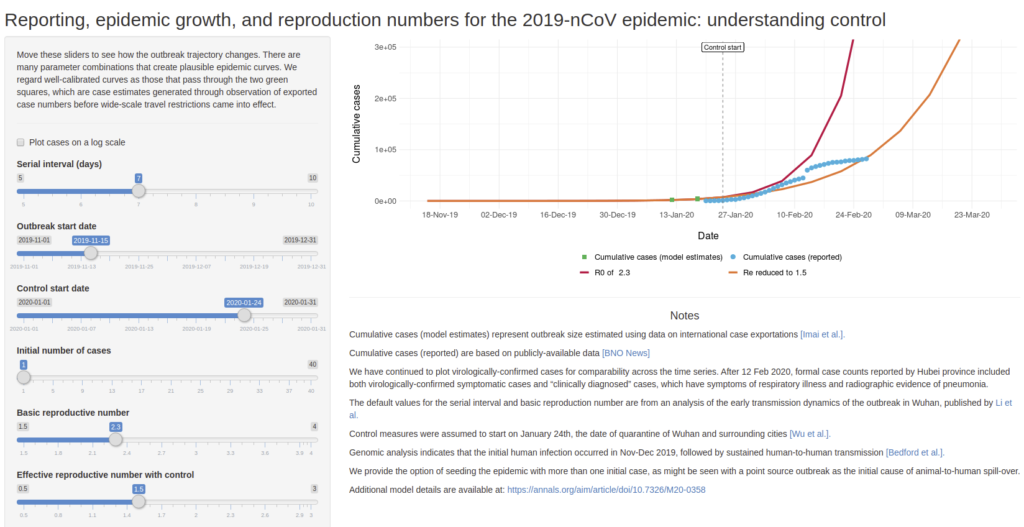
Permet de simuler des scénario de croissance de l’épidémie de COVID-19 en faisant varier quelques paramètres comme :
Serial interval (days) : nombre de jours avant de tomber malade
Outbreak start date : date de début de la maladie
Control start date : date de mise en place de controle (quarantaine, confinement,…)
Initial number of cases : nombre de cas initialement détectés
Basic reproductive number : nombre de personne à leur tour infecté par un malade si aucun contrôle n’est mis en place
Effective reproductive number with control : nombre de personnes à leur tour infecté par un malade si un contrôle est mis en place
Développé par Ashleigh Tuite et David Fisman, Dalla Lana School of Public Health, Université de Toronto
Pour étudier :
Genomic epidemiology of novel coronavirus (HCoV-19)
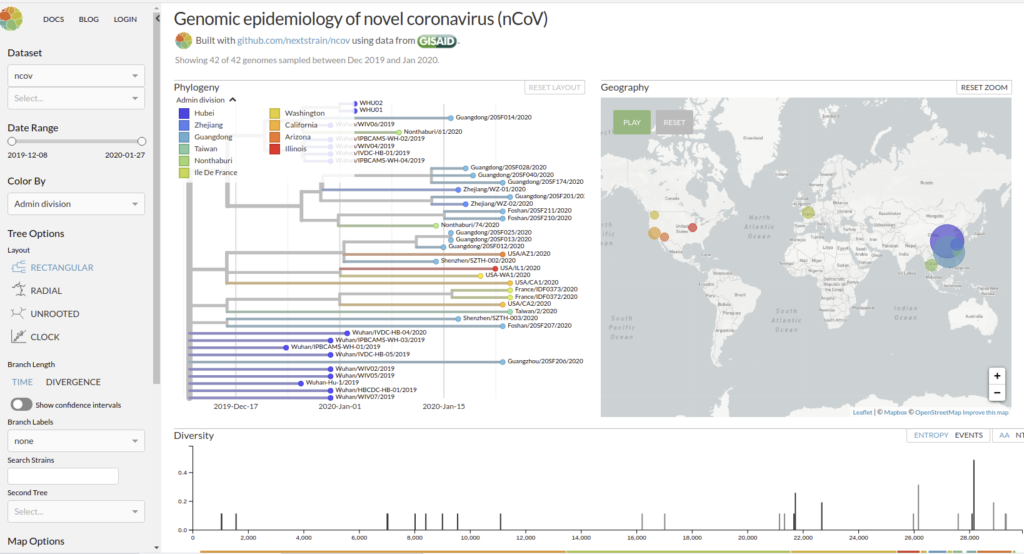
Nextstrain est un projet à open-source visant à exploiter le potentiel scientifique et de santé publique des données sur le génome des agents pathogènes. ils fournissent une vue continuellement mise à jour des données accessibles au public ainsi que de puissants outils d’analyse et de visualisation à l’usage de la communauté. L’objectif est d’aider à la compréhension épidémiologique et d’améliorer la réponse aux épidémies.
Il permet de visualiser les divergences phylogeniques entre les différentes génomes de COVDIR-19 séquencés à ce jour [ 20/02/2020 ]
En savoir plus : Hadfield et al., Nextstrain: real-time tracking of pathogen evolution, Bioinformatics (2018)
Github de l’application : https://github.com/nextstrain/ncov
Des explications précises et instructives sur l’état actuel (au 20 février 2020) des connaissances concernant l’épidémie provoquée par ce nouveau Coronavirus (COVID-19) – Durée : 35Min
Le professeur Arnaud Fontanet revient en détail sur l’histoire de ce virus, de la découverte des premiers cas, à l’enquête sur son mode de transmission jusqu’à son séquençage extrêmement rapide.
Il donne également beaucoup d’informations sur la durée d’incubation, la contagion, les symptômes associés, les mesures sanitaires et le travail des épidémiologistes et des chercheurs pour endiguer la propagation.
Les conséquences économiques ainsi qu’un parallèle pas inintéressant avec la grippe saisonnière permettront, pour certains, de mettre en perspective cette épidémie par rapport à notre monde actuel…
Cette intervention fait partie du MOOC de l’Institut Pasteur « Virus émergents et réémergents ».
Selon une étude : La consommation (modérée) de vin rouge liée à une meilleure santé intestinale…
Une étude du King’s College de Londres tend à montrer que les personnes qui boivent du vin rouge présenteraient une plus grande diversité de microbiote intestinal (un signe de santé intestinale) que les buveurs de vin non rouge.
Dans un article publié le 28 août dans la revue Gastroenterology, une équipe de chercheurs du Department of Twin Research & Genetic Epidemiology au King’s College de Londres a étudié l’effet de la bière, du cidre, du vin rouge, du vin blanc et des spiritueux sur le microbiote intestinal et sur la santé qui en découle chez un groupe de 916 jumelles britanniques. Pourquoi des jumelles? Pour que le fond génétique soit identique, donc la différence de microbiote intestinal entres les jumelles sera en grande partie dû à l’environnement.
Ils ont constaté que le microbiote intestinal des buveurs de vin rouge était plus diversifié que celui des buveurs de vin non rouge. Ceci n’a pas été observé avec la consommation de vin blanc, de bière ou de spiritueux.
La première auteure de l’étude, Caroline Le Roy, du King’s College de Londres, a déclaré : « Bien que nous connaissions depuis longtemps les bienfaits inexpliqués du vin rouge sur la santé cardiaque, cette étude montre qu’une consommation modérée de vin rouge est associée à une plus grande diversité et à un microbiote intestinal plus sain qui explique en partie ses effets bénéfiques sur la santé. »
Le microbiome est la collection de micro-organismes dans un environnement et joue un rôle important dans la santé humaine. Un déséquilibre entre les « bons » microbes et les « mauvais » microbes dans l’intestin peut entraîner des effets néfastes sur la santé, comme un affaiblissement du système immunitaire, un gain de poids ou un taux de cholestérol élevé.
Le microbiote intestinal d’une personne ayant un nombre plus élevé d’espèces bactériennes différentes peut-être considéré comme un marqueur de la santé intestinale.
L’équipe a observé que le microbiote intestinal des consommateurs de vin rouge contenait un plus grand nombre d’espèces bactériennes différentes que celui des non-consommateurs. Ce résultat a également été observé dans trois cohortes différentes au Royaume-Uni, aux États-Unis et aux Pays-Bas. Les auteurs ont tenu compte de facteurs tels que l’âge, le poids, le régime alimentaire régulier et le statut socio-économique des participants et ont continué à voir l’association.
Les auteurs croient que la raison principale de cette association est due aux nombreux polyphénols présents dans le vin rouge. Les polyphénols sont des produits chimiques de défense naturellement présents dans de nombreux fruits et légumes. Ils ont de nombreuses propriétés bénéfiques (y compris des antioxydants) et agissent principalement comme un carburant pour les microbes présents dans notre système.
L’auteur principal, le professeur Tim Spector du King’s College de Londres, a déclaré : « Il s’agit de l’une des plus importantes études jamais réalisées sur les effets du vin rouge sur l’intestin de près de trois mille personnes dans trois pays différents et elle montre que les niveaux élevés de polyphénols dans la peau du raisin pourraient être responsables d’une grande partie des bienfaits controversés pour la santé lorsqu’ils sont utilisés avec modération. »
« Bien que nous ayons observé une association entre la consommation de vin rouge et la diversité du microbiote intestinal, boire du vin rouge rarement, comme une fois toutes les deux semaines, semble suffisant pour observer un effet. Si vous devez choisir une boisson alcoolisée aujourd’hui, c’est le vin rouge qu’il faut choisir, car il semble exercer un effet bénéfique sur vous et sur vos microbes intestinaux, ce qui peut aussi aider à réduire le poids et le risque de maladies cardiaques. Cependant, il est toujours conseillé de consommer de l’alcool avec modération, vous n’avez pas à boire du vin rouge, et vous n’avez pas à commencer à en boire si vous ne buvez pas », a ajouté le Dr Le Roy.
Effectivement, comme dans toute étude, corrélation n’est pas raison! Ainsi et même si la catégorie socio-économique est prise en compte dans l’étude, il pourrait exister d’autres facteurs, non mesurés dans cette étude, qui expliqueraient en partie la bonne santé microbienne des individus. Rappelons que ça n’est pas l’alcool qui est associé avec une meilleure santé intestinale mais d’autres composants du vin rouge (l’hypothèse étant que ce sont les polyphénols), que l’on trouvera aisément dans d’autres aliments (fruits, légumes, noix, cacao…) .
Emplacement de la publication originale :
https://www.sciencedirect.com/science/article/abs/pii/S0016508519412444
L’abus d’alcool est dangereux pour la santé, consommez avec modération
Comment réagiriez vous si l’on vous « offrait » votre génotypage complet, ouvrant la possibilité de prédire d’éventuelles maladies, réactions à certains médicaments etc. ? Certes, dans le cas de l’Estonie (contrairement à ce que propose des sociétés privées telles 23andMe) il s’agit d’une démarche s’inscrivant en santé publique pour le meilleur du bien public et non pour le pire du bien privé… Néanmoins, l’ampleur de la cohorte humaine visée n’est pas sans poser des questions.
L’Estonie a lancé un programme visant à recruter et à génotyper 100.000 nouveaux participants à la biobanque (pour une population nationale totale de 1,316 million de citoyens estoniens !) dans le cadre de son programme national de médecine personnalisée. Cette biobanque comportait déjà 50.000 génotypes de citoyens estoniens. Le gouvernement veut développer un système de soins de santé en offrant à un maximum de ses résidents un génotypage scannant le génome qui sera traduit en rapports personnalisés. Ce rapport serait intégré à la pratique médicale quotidienne par l’entremise du portail national de cybersanté.
Cette biobanque a été initiée à trois fins (Leitsalu et al., International Journal of Epidemiology, 2015) :
- promouvoir le développement de la recherche génétique ;
- recueillir des informations sur l’état de santé de la population estonienne, ainsi que des informations génétiques
- utiliser les résultats de la recherche génétique pour améliorer la santé publique.

Au sein de l’article de Leitsalu et al., les auteurs abordent les forces et faiblesses de leur entreprise :
La principale force de la Biobanque estonienne consiste au fait qu’il s’agit d’une biobanque basée sur la population avec une base de données longitudinales et prospectives. Cela signifie qu’un large éventail de groupes d’âge et de phénotypes sont représentés. Alors que les populations urbaines ont généralement tendance à être surreprésentées, ce n’est pas le cas pour la cohorte de la Biobanque estonienne. La biobanque dispose d’ADN, de plasma et de globules blancs pour chaque donneur. Cela signifie qu’il est possible d’analyser les effets directs des variants de séquence sur le métabolisme. De plus, il est possible de transformer les cellules en lignées cellulaires ou en cellules souches pluripotentes induites (iPS) et de réaliser directement des expériences de biologie moléculaire ou de génétique. Une autre force est fournie par la HGRA (the Estonian Human Genes Research Act) ainsi que par le formulaire de consentement général qui permet de participer à un large éventail de projets de recherche sans avoir à communiquer de nouveau et à demander un nouveau consentement. La HGRA et le formulaire de consentement permettent également aux donneurs de demander la divulgation de leurs données génétiques, de leurs caractéristiques héréditaires et des risques génétiques obtenus à partir de la recherche génétique menée. Cela permettrait à terme de mener des projets sur les tests du génome personnel, la perception des risques et la gestion des risques en milieu industriel.
De plus, la HGRA permet à la Biobanque d’obtenir des renseignements supplémentaires en reliant les dossiers aux registres électroniques nationaux et aux principaux hôpitaux. Tous les registres sont reliés de façon centralisée par une infrastructure technique à l’échelle nationale qui permet l’échange sécurisé de données entre les bases de données. La HGRA a également imposé des restrictions sur les activités de l’EGCUT (Estonian Genome Center of the University of Tartu) et les données collectées dans la Biobanque estonienne. La participation devait être entièrement volontaire – seules les personnes intéressées pour rejoindre la Biobanque estonienne, après en avoir entendu parler soit lors d’événements promotionnels spéciaux, soit par les médias, soit par des amis, soit au cabinet du médecin de famille ou à l’hôpital, sont recrutées. L’EGCUT n’a pas été autorisé à envoyer les lettres d’invitation à leur adresse domiciliaire. Par conséquent, la biobanque ne représente pas un échantillon aléatoire classique et pourrait être sujette à un biais de recrutement. Une proportion considérable de la population recrutée pourrait toutefois compenser ce biais. Par conséquent, bien qu’elle ne soit pas aléatoire sur le plan classique, la cohorte peut quand même être considérée comme représentative de la population. Bien que le recrutement était ouvert à tous, il y a une disproportion d’Estoniens ethniques et de Russes ethniques dans la biobanque, les Estoniens étant surreprésentés (81% dans la biobanque contre 70% dans la population générale) et les Russes sous-représentés (16% dans la biobanque contre 25% dans la population générale). Une autre faiblesse est la profondeur limitée de certains sous-questionnaires. Par exemple, un questionnaire relativement bref sur la fréquence des aliments a été administré sans information détaillée sur l’apport en énergie ou en nutriments ; les mesures des traits glycémiques à jeun, comme le niveau d’insuline, ne sont disponibles que pour un nombre limité d’échantillons. La profondeur limitée des données recueillies peut parfois limiter le nombre de projets dans lesquels les données peuvent être utilisées. Toutefois, des questionnaires plus complets auraient exigé des durées d’entrevue encore plus longues et auraient coûté beaucoup plus cher, ce qui aurait pu entraîner une réduction de la taille de la cohorte.
Le pays dispose de nombreuses solutions numériques sécurisées incorporées dans les fonctions gouvernementales qui relient les diverses bases de données du pays par des voies cryptées de bout en bout. Un site Web a été créé dans le cadre du projet, afin que les Estoniens puissent se porter volontaires et donner leur consentement à être génotypés. La génération des données est assurée par l’institut de génomique de l’Université de Tartu (aujourd’hui les 50.000 premiers génotypages ont été réalisés et analysés, les 100.000 pousseront à une population estonienne à 10 % génotypée)
Les efforts internationaux ont permis d’identifier des milliers d’associations entre les variants génétiques et les maladies, ou traits génétiques, et de créer des cartes des variations uniques au sein des populations.
« Aujourd’hui, nous avons suffisamment de connaissances sur le risque génétique des maladies complexes et la variabilité interindividuelle des effets des médicaments pour commencer à utiliser systématiquement ces informations dans les soins de santé au quotidien « , a déclaré Jevgeni Ossinovski, ministre de la Santé et du Travail. « En coopération avec l’Institut national pour le développement de la santé et l’Université de Tartu, nous allons permettre à 100.000 autres personnes de rejoindre la biobanque estonienne, afin de stimuler le développement de la médecine personnalisée en Estonie et de contribuer ainsi à l’avancement des soins de santé préventifs. «
Le gouvernement estonien a alloué 5 millions d’euros au programme au cours de l’année 2018. Le projet sera coordonné par l’Institut national pour le développement de la santé, dont la tâche est d’élaborer et de mettre en œuvre des procédures et des principes pour la mise en œuvre efficace de la recherche scientifique dans la pratique médicale.
Andres Metspalu, directeur du Centre estonien du génome à l’Université de Tartu, se félicite de l’initiative du ministère des Affaires sociales d’augmenter le nombre de participants à la biobanque. « Nous sommes heureux qu’avec le soutien de ce projet, les résultats des travaux à long terme du centre de génomique seront transférés en médecine pratique et donneront un nouvel élan à nos recherches futures. L’Université contribuera également à la création d’un système de rétroaction pour les participants de la biobanque, et à la formation des professionnels de la santé pour qu’ils puissent donner aux patients une rétroaction fondée sur l’information génétique« .
Le projet sera mis en œuvre sur la base de la loi estonienne sur la recherche sur les gènes humains et du même formulaire de consentement général qui a été utilisé pour les 50.000 premiers participants. Le prélèvement officiel d’échantillons a débuté le 2 avril 2018. A voir si l’expérience relativement pionnière ,à cette échelle, menée en Estonie, fera école ou tâche d’huile dans d’autres pays de l’Union Européenne !
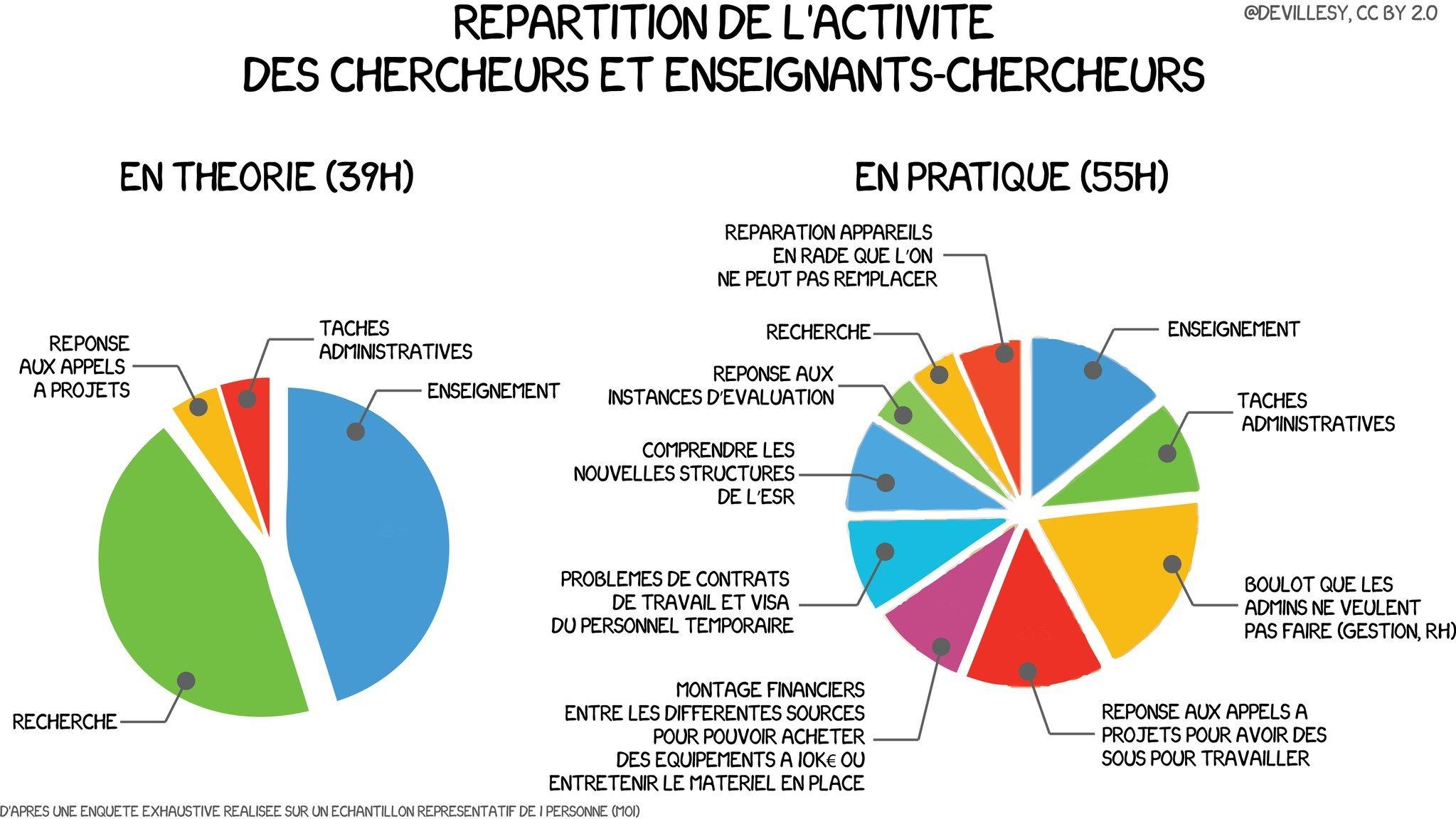
Avec humour et sans complètement tomber dans la caricature extrême, cette infographie met en lumière le travail protéiforme des chercheurs & enseignants-chercheurs, ces hybrides qui naviguent entre laboratoires et amphithéâtres. Plombés par des tâches administratives… il faut dire qu’ils sont bien aidés par du personnel administratif parfois un peu mono-tâche (à café) soucieux de faire coller son volume horaire pratique à la théorie, les chercheurs et enseignants-chercheurs sont les rares personnes à devoir chercher leur propre budget pour répondre à l’injonction d’accroître le corpus de connaissances, en étant tour à tour : évalués, évaluateur, éditeur, vulgarisateur, secrétaire, encadrant de stagiaire de thésards, professeur, RH…
Tout ceci m’a remis en mémoire, ce dessin de Charb issu des cahiers pédagogiques…
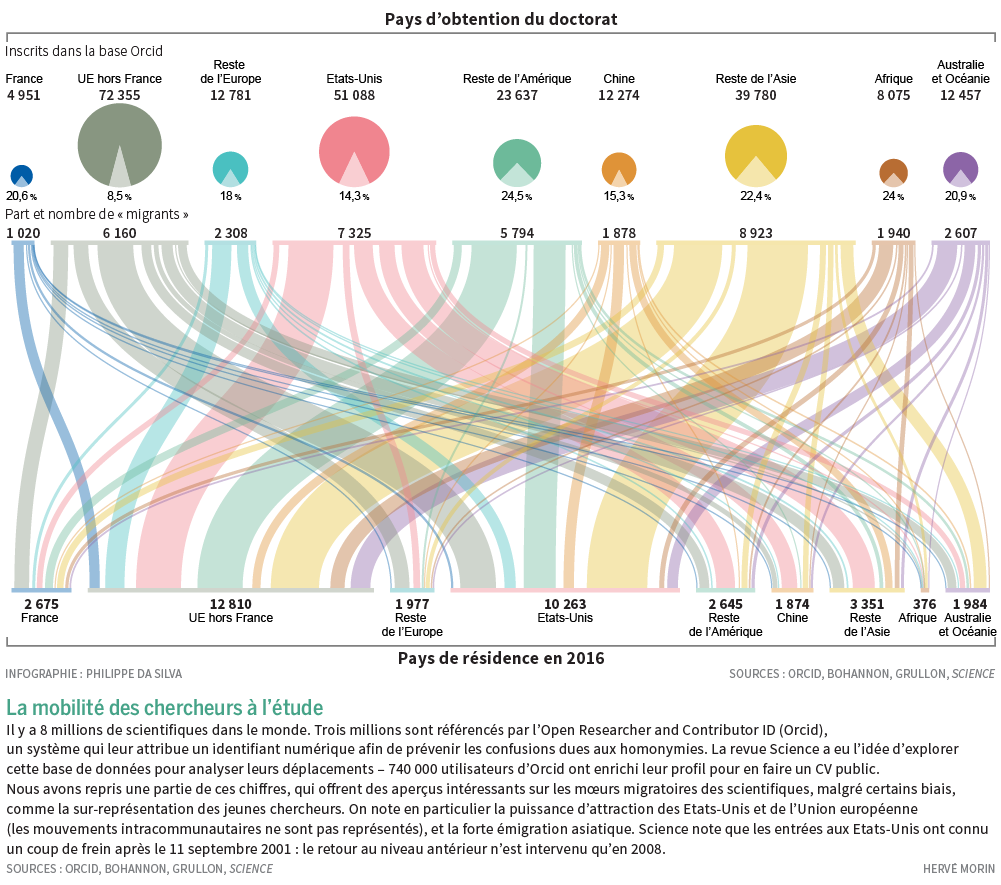
 Voici une infographie tirée du Monde Science et Techno du 23/05/2017. Celle-ci a été permise grâce au numéro ORCID qui permet d’identifier, de façon non ambiguë tout chercheur contributeur d’une publication scientifique. Selon l’Open Researcher and Contributor ID, « ORCID est une organisation à but non lucratif qui a pour objectif d’aider à créer un monde dans lequel tous les intervenants dans les domaines de la recherche, de l’université et de l’innovation sont identifiés de manière unique et sont reliés à leurs contributions et à leurs affiliations, au-delà des limites des disciplines, des frontières et des époques. » Concernant les migrations de chercheurs, l’Union Européenne et les Etat-Unis restent attractifs. En France, un cinquième des chercheurs ayant obtenu leur diplôme sur le territoire, émigre, ce qui est plutôt beaucoup… une question peut se poser : est ce que la proportion de chercheurs quittant leur pays d’obtention de doctorat ne serait pas inversement proportionnelle à l’intérêt de ce pays pour sa propre recherche scientifique…?
Voici une infographie tirée du Monde Science et Techno du 23/05/2017. Celle-ci a été permise grâce au numéro ORCID qui permet d’identifier, de façon non ambiguë tout chercheur contributeur d’une publication scientifique. Selon l’Open Researcher and Contributor ID, « ORCID est une organisation à but non lucratif qui a pour objectif d’aider à créer un monde dans lequel tous les intervenants dans les domaines de la recherche, de l’université et de l’innovation sont identifiés de manière unique et sont reliés à leurs contributions et à leurs affiliations, au-delà des limites des disciplines, des frontières et des époques. » Concernant les migrations de chercheurs, l’Union Européenne et les Etat-Unis restent attractifs. En France, un cinquième des chercheurs ayant obtenu leur diplôme sur le territoire, émigre, ce qui est plutôt beaucoup… une question peut se poser : est ce que la proportion de chercheurs quittant leur pays d’obtention de doctorat ne serait pas inversement proportionnelle à l’intérêt de ce pays pour sa propre recherche scientifique…?
Ce « marqueur » – le taux d’émigration des chercheurs diplômés du pays qu’ils quittent- peut, en même temps, être lié à une bonne santé d’un système éducatif et de formation.
En effet, plus les étudiants diplômés seront perçus comme bien formés plus ils auront de facilités à le quitter, attirés par des pays dont la recherche est plus dynamique (entendons par là des pays rémunérant mieux, offrant de meilleurs capacités d’accueil) que celui dans lequel ils ont obtenu leur diplôme. En définitive, cette proportion, ce marqueur peuvent en quelque sorte être liés à une distorsion entre deux capacités pour un pays : celui de former et celui de réaliser une recherche de haut niveau permettant de garder les chercheurs que ce même pays a formé.
 Vous pouvez tout aussi bien lire cet article en sur le site du Monde : http://www.lemonde.fr/sciences/. Il s’agit du point de vue proposé par Guillaume Miquelard-Garnier, cofondateur du think-tank l’Alambic et maître de conférences au CNAM.
Vous pouvez tout aussi bien lire cet article en sur le site du Monde : http://www.lemonde.fr/sciences/. Il s’agit du point de vue proposé par Guillaume Miquelard-Garnier, cofondateur du think-tank l’Alambic et maître de conférences au CNAM.
Les grandes questions médiatiques du moment concernant l’enseignement supérieur et la recherche, qu’il s’agisse par exemple du campus Paris-Saclay, des partenariats public-privé à l’université, ou des MOOCs [Massive Open Online Courses, cours en ligne ouverts et massifs], laissent souvent de côté une problématique pourtant essentielle : quelle politique de recrutement à court ou moyen terme envisage-t-on pour la recherche académique française, et subséquemment, qui pour faire la recherche en France ?
Historiquement, le système français était plutôt fondé sur des crédits dits récurrents (attribués directement aux laboratoires et répartis ensuite entre chercheurs). Le fonctionnement typique d’un laboratoire était un assemblage de petites équipes « pyramidales » de permanents (un chercheur senior, deux ou trois chercheurs junior) avec un recrutement plutôt jeune, et donc relativement peu d’étudiants ou de chercheurs « précaires » (attaché temporaire d’enseignement et de recherche, post-doctorants…).
Si l’on préfère, le fonctionnement se faisait avec un ratio permanents/non-permanents élevé. Dans ce système, le chercheur junior est celui qui est en charge de faire la recherche au quotidien, d’obtenir les résultats et d’encadrer de près, avant de, plus expérimenté, migrer vers des activités de mentorat scientifique de l’équipe (rôle du chercheur senior).
Ce fonctionnement était à l’opposé de celui, par exemple, des Etats-Unis. La recherche y est financée exclusivement ou très majoritairement sur projet, c’est-à-dire par « appels d’offres » ou « appels à projets ». Les chercheurs, principalement à titre individuel, décrivent leurs idées sous forme de projets à des agences gouvernementales ou des industriels, une sélection étant ensuite effectuée par des panels d’experts et l’argent réparti en fonction de ces choix.
Dans ce système, il y a peu de permanents, beaucoup de non-permanents recrutés sur les budgets issus des appels à projets pour la durée de ceux-ci, et un fonctionnement « individuel » (chaque chercheur, junior ou senior, gère son propre groupe de doctorants et post-doctorants, en fonction de son budget). Le chercheur est, dès son recrutement (aux USA, la tenure track), un chef de groupe-chef de projets, dont le rôle est de définir les grandes orientations intellectuelles, de trouverles financements, de les répartir et de recruter. La recherche proprement dite est alors très majoritairement effectuée par les non-permanents.
SITUATION ALARMANTE
Il n’est pas, ici, question de débattre des avantages et inconvénients des deux systèmes. Le premier favorise le mandarinat et un système de « rente scientifique » quand le second amène une tendance à l’effet Matthieu (6 % des chercheurs américains monopolisent 28 % des financements) et mise beaucoup pour le recrutement des précaires sur un fort attrait des pays asiatiques qui n’est peut-être pas éternel.
Toutefois, il faut constater que, depuis une quinzaine d’années au moins, et encore plus depuis la mise en place de l’Agence nationale de la recherche (ANR) en 2005 et les réformes engagées par Valérie Pécresse et poursuivies jusqu’à aujourd’hui, le système français est en mutation.
Les recrutements sont devenus plus tardifs, autour de 33 ans en moyenne pour un maître de conférences ou un chargé de recherches, soit typiquement cinq ou six années après l’obtention de la thèse (pour les sciences dures). Les financements récurrents ont été largement diminués au profit des financements par projet. Les appels à projets, principalement par le biais de l’ANR ou européens, ont développé les recrutements de post-doctorants. Les initiatives pourpromouvoir l’excellence individuelle des chercheurs se sont multipliées (la prime d’excellence scientifique pérennisée même si rebaptisée, les bourses jeunes chercheurs nationales ou européennes sur un modèle de tenure track ou servant à financer un groupe de recherche indépendant).
Or, plus récemment, la révision générale des politiques publiques (RGPP) conduit, malgré l’autonomie des universités, à un effondrement alarmant des recrutements de chercheurs et enseignants-chercheurs permanents à l’université et dans les établissements publics à caractère scientifique et technologique (EPST). En trois ans, le CNRS est passé de 400 chercheurs recrutés par an à 300 (soit d’environ 350 à 280 jeunes chercheurs). Cette année, on compte au total 1 430 postes de maîtres de conférences ouverts au concours alors qu’il y en avait encore 1 700 il y a deux ans et 2 000 il y a cinq ans. Cette situation a conduit le conseil scientifique du CNRS à s’alarmer récemment, et ne devrait pas s’améliorer à la suite des énormes problèmes financiers de bon nombre d’universités, dont le symbole est Versailles-Saint-Quentin.
En parallèle, le budget de l’ANR alloué aux projets est passé de plus de 600 millions d’euros en 2010 à moins de 500 actuellement, le nombre de projets financés de 1 300 en 2010 à très certainement moins de 1 000 en 2014.
A cela s’ajoutent les effets liés à la loi Sauvadet de 2012. Cette loi favorisant la titularisation des personnels contractuels après six ans passés dans la fonction publique a jeté un froid dans certains laboratoires et certaines disciplines scientifiques (notamment celles qui recrutaient plutôt à 35 ans qu’à 31…). Les budgets ne permettant que rarement ces « cdisations » non planifiées, les ressources humaines des organismes sont aujourd’hui très craintives et rendent difficile l’embauche d’un post-doctorant dès la quatrième voire la troisième année.
La baisse du budget de l’ANR, censée s’accompagner d’une revalorisation des financements récurrents, a d’ailleurs probablement et principalement servi àfinancer ces titularisations non anticipées dans la mesure où l’argent n’est en tout cas pas arrivé jusqu’aux laboratoires. En lien avec cette loi Sauvadet et cette résorption de la « précarité » dans l’enseignement supérieur et la recherche, on peut également souligner que les règles de l’ANR concernant l’embauche de contractuels ont été rendues plus drastiques : il faut aujourd’hui trois permanents à temps plein sur un projet pour recruter un non-permanent à temps plein pour la durée de celui-ci.
AU MILIEU DU GUÉ
Nous nous trouvons donc aujourd’hui au milieu du gué, et l’on se demande si nous y sommes arrivés de façon réfléchie ou simplement par suite de tiraillements successifs et aléatoires vers les directions opposées prises par ces deux systèmes.
Des financements récurrents qui ont quasiment disparu mais également des financements sur projets qui s’effondrent. Des permanents qui ont de moins en moins de temps à consacrer à la pratique de la recherche pour en passer de plus en plus à la gestion (de projets, de groupes) pour les plus talentueux ou chanceux, et à la lutte pour l’obtention des crédits nécessaires à leur activité pour les autres. Et de moins en moins de docteurs non permanents dans les laboratoires et de plus en plus de difficultés à les financer ou plus simplement à les attirer (puisqu’on ne peut honnêtement plus rien leur promettre et que les salaires proposés ne sont toujours pas compétitifs).
La question se pose alors simplement : qui, en dehors des quelques doctorants passionnés qui pourront encore être financés (et alors que les difficultés d’insertion des docteurs sont toujours récurrentes en France, ce qui est tout sauf une incitation à envisager le doctorat comme un choix de carrière judicieux), fera demain de la recherche dans les laboratoires publics en France ?
Point de vue : LE MONDE SCIENCE ET TECHNO | 05.05.2014 à 17h00 • Mis à jour le 07.05.2014 à 13h57
 Parmi les technologies dédiées à la génomique, l’ « Optical Mapping » fait figure d’outil qualifiable d’alternatif. Cette approche repose sur une représentation graphique des sites de restrictions enzymatiques au travers d’un génome complet.
Parmi les technologies dédiées à la génomique, l’ « Optical Mapping » fait figure d’outil qualifiable d’alternatif. Cette approche repose sur une représentation graphique des sites de restrictions enzymatiques au travers d’un génome complet.
Les applications concernent aussi bien la génomique comparative (détection des délétions, insertions, inversions ou translocations), que le typage de souches (comparaison des cartes de restrictions). Aussi, conjuguée aux technologies de séquençage à haut-débit, elle permet également de répondre aux illusions fréquentes de l’obtention d’un « draft » de génome d’intérêt, nouvellement séquencé. Actuellement, OpGEN est la seule société proposant une solution semi-automatisée de cette technologie.
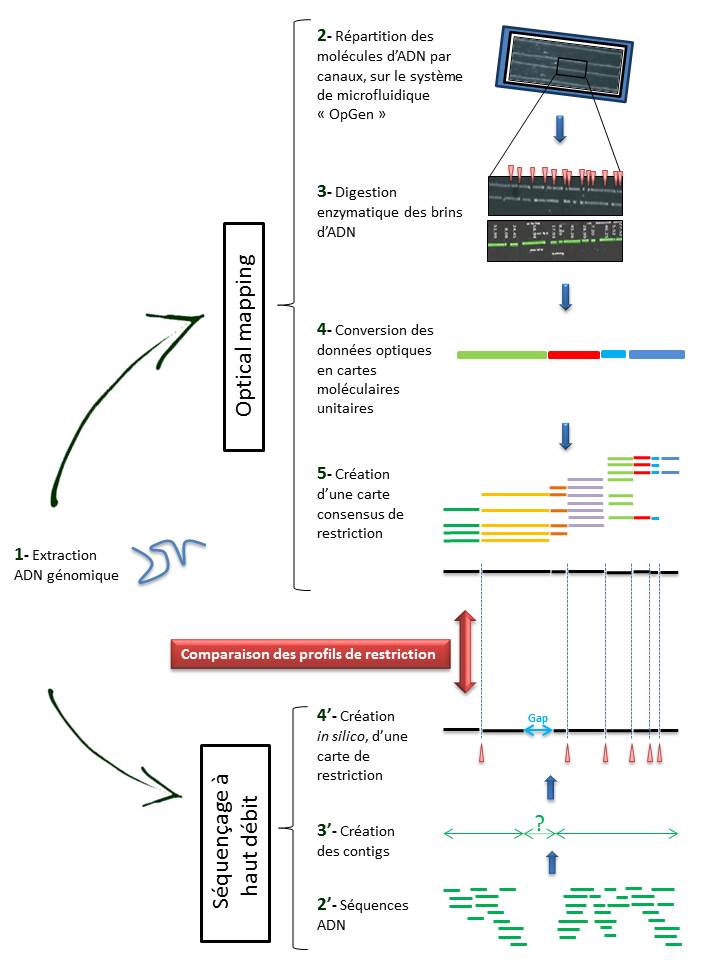
Techniquement, l’ « Optical mapping » consiste en (Cf fig ci-dessous):
– Une immobilisation des fragments d’ADN génomique extraits (1) au sein de canaux intégrés dans un support dédié (Argus System – OpGen) (2).
– Chaque molécule subit une digestion enzymatique (endonucléase de restriction) générant des sites de clivage, symbolisés ci dessous par les espaces (3).
– Le logiciel d’analyse (MapSolver) convertit ces données optiques en cartes moléculaires unitaires (4), qui alignées, fournissent une carte de restriction consensus du génome étudié (5).
L’utilisation de cette méthode, dans la perspective d’un assemblage efficace de génome, ne cesse de croître. En effet, elle permet de pallier les limites des NGS (Homopolymères, zones de génome peu ou non couvert) qui ne permettent bien souvent d’aboutir qu’à un nombre restreint de contigs (3′).
Il convient alors de créer une carte de restriction, in silico, de ces contigs (4′), à leur tour alignés sur l’ « optical map » du génome, sur la base des sites de clivage. Cette comparaison permet alors de positionner les contigs entre eux, de les orienter et de mettre en lumière les hypothétiques gaps. Le scaffold des contigs ainsi établi, associé à un séquençage Sanger des gaps permettent ainsi d’aboutir à un « draft » du génome étudié.
L’ « optical mapping » apparait comme un outil fiable et utile dans l’assemblage de génome, d’autant qu’il fait appel à une technique différente, indépendante mais à la fois très complémentaire au séquençage à haut débit.
 A l’aube des années 2000, la génomique appliquée au domaine végétal fait l’objet d’une mobilisation internationale de grande ampleur comme en témoignent les programmes « Zygia » et « Gabi » en Allemagne, « Plant Genome Initiative » aux États-Unis, ou encore « Rice Genome Research Program » au Japon, qui poursuivent des objectifs analogues. Il devient primordial d’identifier les gènes et leur fonction jouant notamment un rôle décisif dans la production végétal (Rusticité, résistance aux maladies, aux herbicides, etc…).
A l’aube des années 2000, la génomique appliquée au domaine végétal fait l’objet d’une mobilisation internationale de grande ampleur comme en témoignent les programmes « Zygia » et « Gabi » en Allemagne, « Plant Genome Initiative » aux États-Unis, ou encore « Rice Genome Research Program » au Japon, qui poursuivent des objectifs analogues. Il devient primordial d’identifier les gènes et leur fonction jouant notamment un rôle décisif dans la production végétal (Rusticité, résistance aux maladies, aux herbicides, etc…).
Cette période est également marquée par l’achèvement du séquençage du génome de la plante modèle Arabidopsis thaliana, étape majeure dans la recherche en biologie végétale.
Simultanément, des collections de mutants d’insertions (T-DNA) chez A. thaliana sont créés au sein de nombreux groupes (SALK, GABI-Kat, Syngenta, INRA Versailles, etc…), et elles émergent notamment au travers du projet « Genoplante« , programme fédérateur en génomique végétale (Groupement d’Intérêt Scientifique regroupant à la fois des organismes publics tel que l’INRA, CNRS, Cirad, IRD et de puissants partenaires privés tel que Biogemma, Rhône-Poulenc Santé végétale et animale et Bioplante). L’idée est donc d’utiliser ces banques de mutants comme outils pour la génomique fonctionnelle appliquée à la plante modèle.
A l’époque, les solutions proposées pour l’identification des positions d’insertion du T-DNA au sein du génome sont nombreuses ( « Tail-PCR », « Inverse PCR », « Kanamycin Rescue » ). Néanmoins, ces approches restent fastidieuses: En plus de présenter certaines étapes techniques limitantes, elles sont également très chronophages.
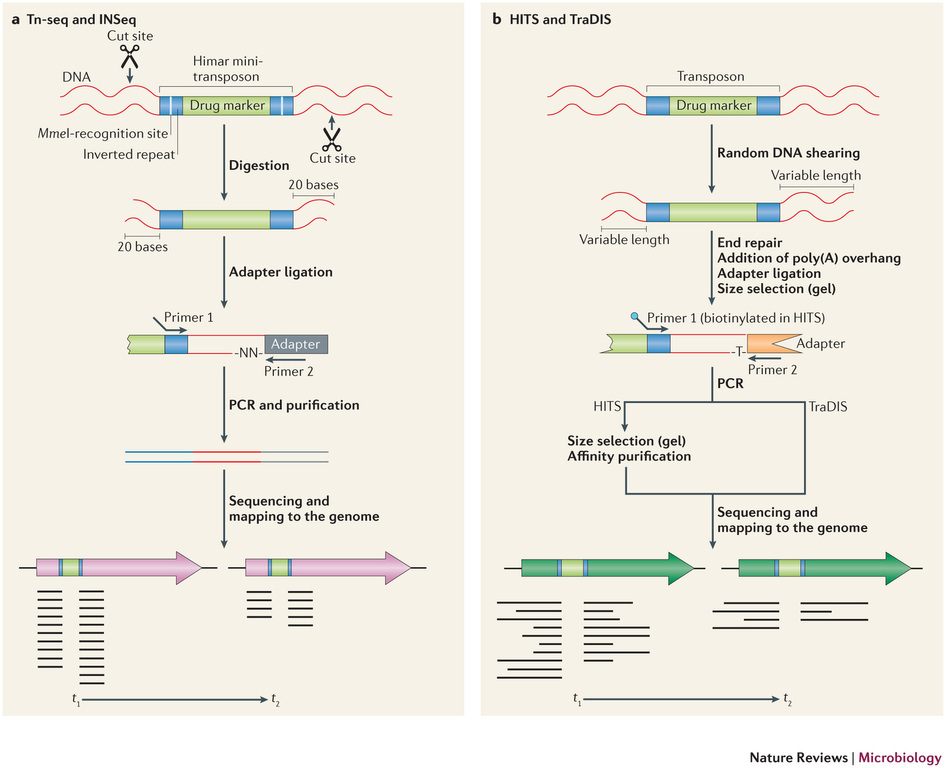
Récemment, de nombreuses études ont commencé a démontrer l’énorme potentiel du séquençage à haut-débit dans l’identification des sites d’insertion de transposons. Le terme générique « Tn-Seq », pour « Transposon-Sequencing », est une variante du séquençage d’amplicons ciblés (Target-seq) et peut se décliner selon quatre méthodes comme illustrées ci-dessous (Tim van Opijnen and Andrew Camilli, Nature reviews – Microbiology (2013 July)). Elles dépendent notamment de la procédure de préparation de librairie de séquençage employée:
– Le »Tn-seq » et « INSeq » (respectivement pour « Transposon sequencing » et « Insertion sequencing ») sont deux approches très similaires reposant sur un séquençage d’amplicons obtenus à partir d’un couple d’oligos dont l’un cible le transposon. Seule la méthode de purification varie (Gel PAGE pour « INSeq » et Gel agarose pour « Tn-Seq)
– Le « HITS » et « TraDIS » (respectivement pour « High-throughput insertion tracking by deep sequencing » et « Transposon-directed insertion site sequencing ») sont également deux méthodes très similaires notamment en amont de l’étape de PCR de librairie.
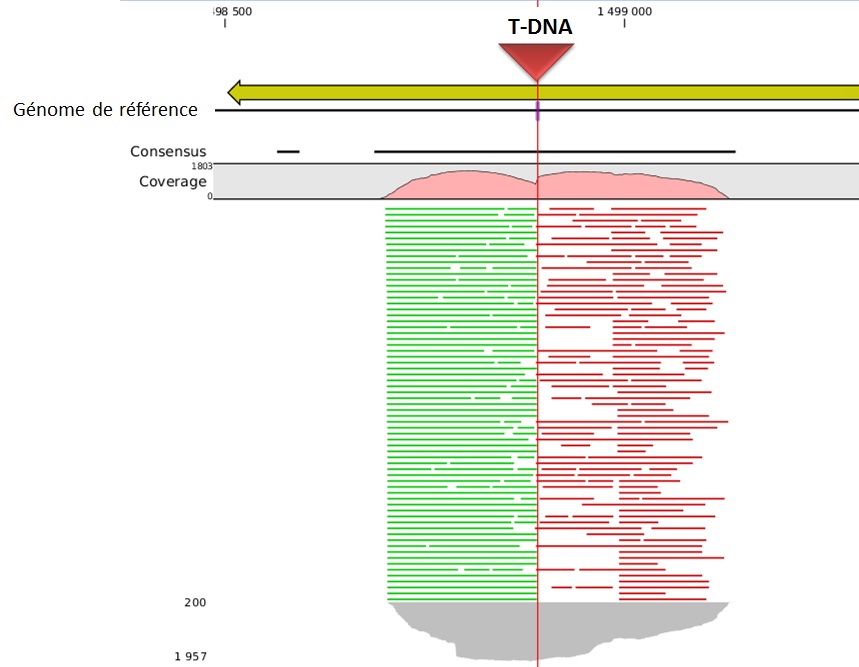
L’alignement des données de séquençage (.fastq) sur le génome de référence, permet ainsi d’identifier la position du site d’insertion. L’illustration met en évidence les « reads » issus de la PCR de librairie ciblant les régions flanquantes au Transposon (« En vert » la bordure gauche, « en rouge » la bordure droite). Sur la base de cette méthode, il devient donc aisé d’identifier le nombre d’insertion potentielle.
L’utilisation des technologies de séquençage à haut-débit pour l’identification des sites d’insertion de T-DNA dans les banques de mutants révolutionnent les méthodes de criblage. Tout en s’affranchissant de techniques fastidieuses, cette approche de Tn-seq présente à la fois l’avantage de pouvoir gérer simultanément un très grand nombre d’échantillons (barcoding), à des coûts réduits et dans un délai des plus respectables.
 Le séquençage du génome humain pour 30$, c’est la promesse faite par David Weitz, co-fondateur de GnuBio au cours de l’année 2010. Trois ans plus tard, la start-up vient de lancer en béta-test son nouveau procédé de séquençage à haut débit. Il s’agit du premier système entièrement intégré (amplification des cibles, enrichissement, séquençage et analyse) qui propose pour le moment une application de target-sequencing destinée aux cliniciens et dédiée au diagnostic moléculaire.
Le séquençage du génome humain pour 30$, c’est la promesse faite par David Weitz, co-fondateur de GnuBio au cours de l’année 2010. Trois ans plus tard, la start-up vient de lancer en béta-test son nouveau procédé de séquençage à haut débit. Il s’agit du premier système entièrement intégré (amplification des cibles, enrichissement, séquençage et analyse) qui propose pour le moment une application de target-sequencing destinée aux cliniciens et dédiée au diagnostic moléculaire.
En 2010, David Weitz et son équipe de l’Université d’Harvard ambitionnent de développer une nouvelle technologie de séquençage à haut-débit, alliant les technologies de biologie moléculaire aux procédés de microfluidique développés quelques années plus tôt (2004) au sein de la société RainDance technologies.
Cette nouvelle approche repose sur la capacité à générer des gouttes de l’ordre du picolitre et pouvant être déplacée sur une puce microfluidique. Ces gouttes peuvent renfermer soit un couple d’amorces, des adaptateurs, ou tout autre type de réactifs nécessaires aux étapes de préparation de librairie et de séquençage (séquençage par hybridation-ligation, type SOLiD avec une fidélité de 99.99%). Dès lors, leurs quantités utilisées au sein de ces picogouttes sont considérablement revues à la baisse, ce qui constitue le point clé à une réduction des coûts de séquençage et donc la perspective d’un séquençage de génome humain à 30$, selon David Weitz.
Les projets de GnuBio sont désormais d’élargir le champs d’applications de leur séquenceur à l’analyse transcriptomique (RNA-seq), l’étude de la méthylation (ChiP-seq) ou encore le séquençage de génome entier. La société ambitionne une commercialisation de leur équipement au cours de l’année 2014.
A suivre…
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages