En pleine guerre froide des compagnies de biotechnologies, développeuses et vendeuses de matériel de séquençage… une fuite de documents est organisée. Le tout pour fournir aux personnes prescriptrices ou décideuses d’un futur achat de séquenceur, les arguments pour orienter ou justifier leur choix… parfois même leur mauvaise foi.

En cliquant sur la volute nucléique (depuis le 30/08/2012 – ce lien est censuré par Illumina- cf. commentaires de ce même article) ci-dessus vous aurez accès à un vrai-faux document confidentiel fourni par Illumina. Ce document assez bien réalisé a devancé celui de Life Technologies présenté dans notre article précédent. Il met l’accent sur le bénéfice, au niveau de la qualité des données générées, que procure un MiSeq (Illumina) par rapport à un PGM (Ion Torrent, Life Technologies)
Un document paru dans la Ion Community (site de partage autour de la technologie Ion Torrent et Ion Proton de Life Technologies) commence à faire parler de lui. En effet, des scientifiques de Life Technologies montrent et démontrent que le MiSeq d’Illumina produit (aussi) des erreurs au niveau des homopolymères.
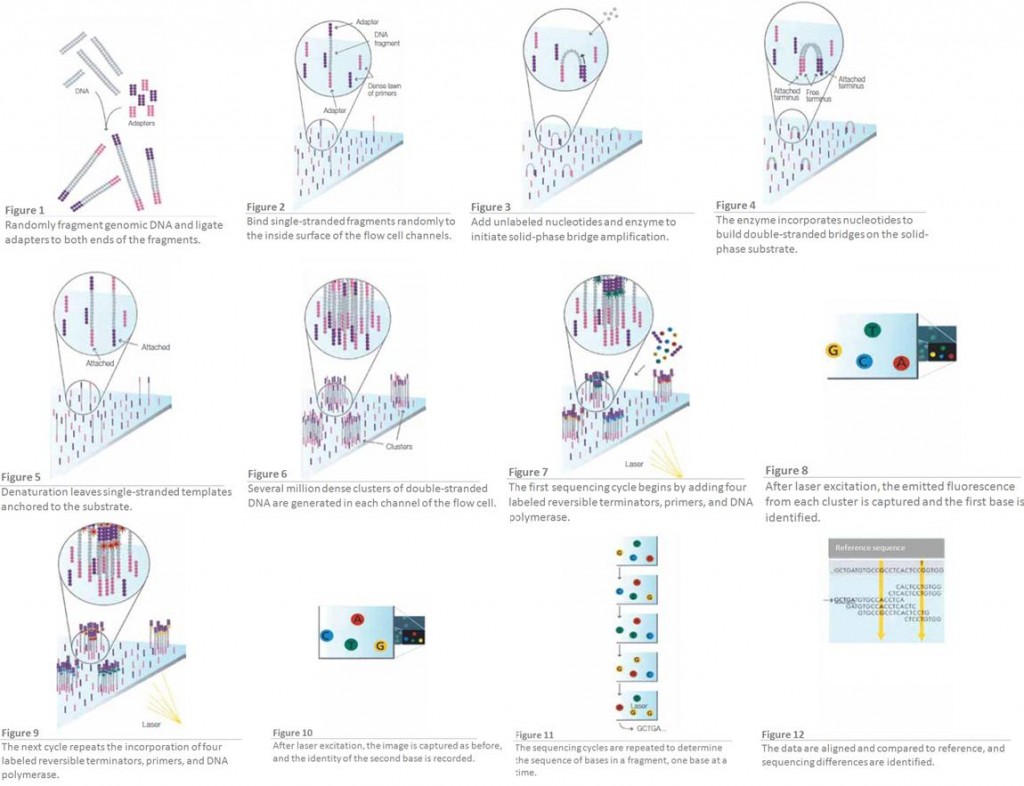
Nous savions depuis longtemps que les séquenceurs de type 454 ou Ion Torrent montraient des biais quant à détermination correcte de ce type de séquences… Ici, pour la première fois les méthodes de SBS (Sequencing By Syntesis) dont le principe pour la technologie Illumina est rappelée par le schéma disponible ci-dessous, montrent des biais étonnement importants pour la juste détermination de séquences adjacentes d’homopolymères (particulièrement concernant les régions GC % riches). La note qui met le feu dans l’argumentaire commercial d’Illumina est disponible ici-même. Une hypothèse quant à la génération de ce type d’erreurs est formulée.
Nous vous laissons le soin de lire la note, intéressante malgré la partialité de la source. La démonstration quoique scientifique, montre l’âpreté de la bataille qui se joue entre les acteurs majeurs du séquençage de paillasse de deuxième génération.
 Cet article est l’occasion de mettre en avant un site plutôt fourni, SEQanswers… un jeu de mots pour une communauté d’utilisateurs de technologie de séquençage haut-débit. Ce site, un peu bouillon, fondé en 2007 par Eric Olivares (qui a travaillé pour Pacific Biosciences) s’adresse aux biologistes moléculaires plus qu’aux bio-informaticiens. Malgré tout, le lien que nous vous présentons ici, renvoie sur la partie Wiki du site SEQanswers. Cette page liste et ordonne en fonction de leurs domaines d’applications les logiciels (gratuits et commerciaux) utiles pour le devenir de vos reads produits par séquençage haut-débit : http://seqanswers.com/wiki/Software/
Cet article est l’occasion de mettre en avant un site plutôt fourni, SEQanswers… un jeu de mots pour une communauté d’utilisateurs de technologie de séquençage haut-débit. Ce site, un peu bouillon, fondé en 2007 par Eric Olivares (qui a travaillé pour Pacific Biosciences) s’adresse aux biologistes moléculaires plus qu’aux bio-informaticiens. Malgré tout, le lien que nous vous présentons ici, renvoie sur la partie Wiki du site SEQanswers. Cette page liste et ordonne en fonction de leurs domaines d’applications les logiciels (gratuits et commerciaux) utiles pour le devenir de vos reads produits par séquençage haut-débit : http://seqanswers.com/wiki/Software/
Cette page est plutôt bien renseignée et vous donnera un large choix de logiciels : vous y trouverez des assemblers de novo, des logiciels pour réaliser du RNAseq (quantification), des logiciels permettant de trouver des pics après ChipSeq… peu de logiciels indispensables sont absents de cette liste qui comporte un peu moins de 500 logiciels…
La paléontologie, la science des fossiles et des traces de vie du passé, use de méthodes de biologie moléculaire de pointe qui pallient les effets du temps qui passe…
Afin d’introduire ce premier article traitant de paléogénomique, les moyens de la biologie moléculaire au service de la paléontologie, une vidéo amuse-bouche (Auteur(s) : Eva-Maria Geigl, Réalisation : Samia Serri, Production : Université Paris Diderot, Durée : 17 minutes 40 secondes) est disponible en usant du fameux clic gauche sur la capture d’image ci-dessous. Cette vidéo vaut surtout pour l’accent mis sur les précautions indispensables pour l’étude d’un échantillon précieux fossilisé… et dont l’ADN, peu abondant, peut être fragmenté. En outre, des mesures simples mais draconiennes permettent de limiter les sources de contaminations, quand l’ADN moderne peut polluer l’ADN fossile. Le port de sur-chausses, de masque et les changements de blouses, le non croisement des échantillons avant et après amplification sont autant de précautions mises en avant dans cette vidéo… une occasion de visiter virtuellement les laboratoire de l’Institut Jacques Monod.
L’une des problématiques liées à l’étude de l’ADN « fossile » réside dans sa faible quantité disponible. Plusieurs méthodes de biologie moléculaire ont été envisagées pour amplifier ce matériel génétique afin d’en permettre l’expertise. Une publication dans BMC Genomics de 2006, Assessment of whole genome amplification-induced bias throughhigh-throughput, massively parallel whole genome sequencing, relate la comparaison de 3 méthodes d’amplifications pan-génomiques (méthodes WGA pour Wide Genome Amplification) d’ADN qui pourra devenir ensuite la matrice suffisante d’un séquençage haut-débit.
– la PEP-PCR (Primer Extension Preamplification-PCR) : cette technique fait intervenir des amorces aléatoires aux conditions d’appariement à basse température (low melting temperature) qui initieront la PCR
référence : Zhang, L. et al. (1992) Whole genome amplification from a single cell: Implications for genetic analysis. Proc. Natl. Acad. Sci. USA 89, 5847
– la DOP-PCR (Degenerate Oligonucleotide Primed-PCR) : cette technique, quant à elle, fait intervenir des amorces semi-dégénérées (de type : CGACTCGAGNNNNNNATGTGG) qui ont une température d’hybridation supérieure à celles utilisées dans la PEP-PCR
référence : Telenius, H. et al. (1992) Degenerate oligonucleotide-primed PCR: general amplification of target DNA by a single degenerate primer. Genomics 13, 718.
L’utilisation d’une Taq PCR limite la taille des fragments néo-synthétisés qui ne dépassent guère 3 kb. En outre, ces deux techniques, à l’instar de ce qui peut être démontré dans la publication de BMC Genomics 2006 (mentionnée ci-dessus), induisent des erreurs de séquences accompagnées de nombreux biais d’amplification (certaines régions ne sont pas amplifiées au profit de régions qui deviennent, de fait, sur-représentées).
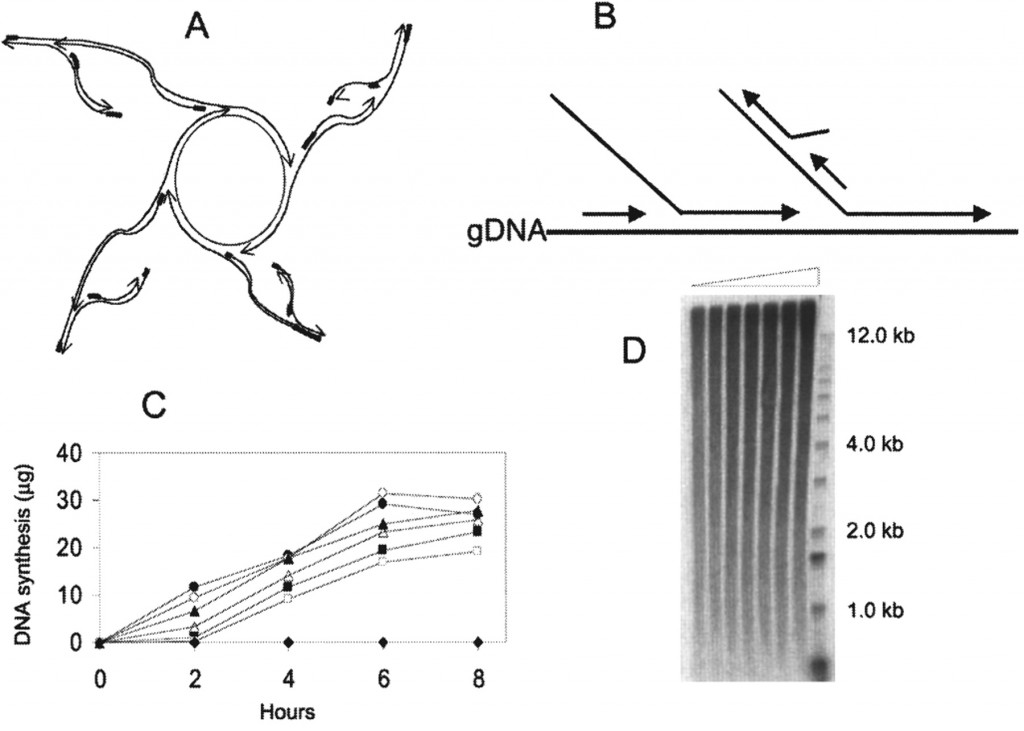
– la MDA (Multiple Displacement Amplification) : cette amplification iso-thermique fait intervenir des amorces aléatoires de type hexamères et une enzyme, la phi29. Le type d’amplification générée est schématisées sur la figure ci-dessous. L’enzyme surfe à partir du brin néo-synthétisé, déplace un brin complémentaire pour continuer sa synthèse. Ainsi, les brins générés par cette technique peuvent atteindre 100 kb. En outre, la phi29 possède une activité 3′ -> 5′ de relecture (proofreading) lui conférant un taux d’erreur 100 fois moindre que ceux constatés pour des Taq polymérases classiquement utilisées dans les techniques de PEP- ou DOP-PCR
source : Cold Spring Harb Protoc 2011.2011: pdb.prot5552 (la légende originale de la figure est disponible en cliquant sur celle-ci)
Ces techniques d’amplification pangénomique ont rendu possible l’étude d’ADN anciens et peu abondants et tout naturellement elles ont trouvé leur place dans la boîte à outils moléculaires des paléogénéticiens. Cependant, la révolution des séquençages haut-débit (dont nous avons abordé le sujet à plusieurs reprises) laisse entrevoir un nouveau champ des possibles pour l’étude des ADN fossiles. Au fond, des technologies telles que celle développée par Helicos Biosciences, trouvent ici un réel champ d’application à l’instar de ce que développe la publication True single-molecule DNA sequencing of a pleistocene horse bone de Genome Research, 2011- nulle nécessité d’amplifier la matrice de départ. Cette publication compare des technologies de séquençage de 2ème et 3ème générations (GaIIx et Helicos) appliquées au séquençage de l’ADN isolé à partir d’un os de cheval pleistocène conservé dans permafrost. Le séquençage « single molecule« , une chance pour la paléogénomique !
- des fiches logiciels (classement, descriptions et lien pointant vers l’hébergeur de ceux-ci)
- des fiches ressources (site internet, évènements, documents utiles…)
- mutualiser les compétences sur les logiciels
- promouvoir les développements internes
- animer une communauté autour du logiciel
- promouvoir l’usage des logiciels libres et la contribution à leur élaboration
 Noblegen Biosciences, start-up localisée dans le Massachusetts, ambitionne de commercialiser pour 2014 « optipore » (pour « optical detection » et « nanopore ») , un séquenceur de paillasse de troisième génération combinant nanotechnologies et un système de lecture optique permettant de réduire drastiquement les coûts de séquençage. L’objectif est de conquérir le marché des laboratoires cliniques dans la perspective de l’émergence d’une médecine personnalisée et de proposer ainsi un séquençage de génome humain complet à faible coût et dans un temps record. Dans cette course engagée, la société américaine se trouve en bonne position.
Noblegen Biosciences, start-up localisée dans le Massachusetts, ambitionne de commercialiser pour 2014 « optipore » (pour « optical detection » et « nanopore ») , un séquenceur de paillasse de troisième génération combinant nanotechnologies et un système de lecture optique permettant de réduire drastiquement les coûts de séquençage. L’objectif est de conquérir le marché des laboratoires cliniques dans la perspective de l’émergence d’une médecine personnalisée et de proposer ainsi un séquençage de génome humain complet à faible coût et dans un temps record. Dans cette course engagée, la société américaine se trouve en bonne position.
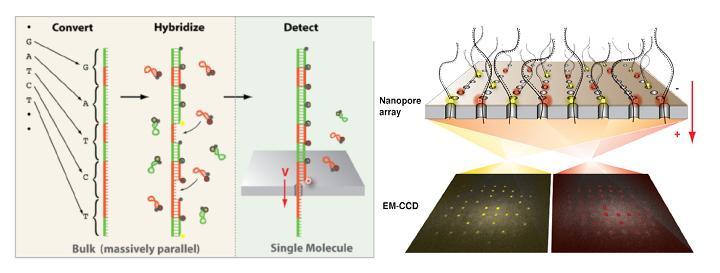
A l’instar du PGM Ion torrent, une puce en silicium constitue le coeur de la machine et renferme des centaines de nanopores.  La molécule d’ADN unique native destinée à être séquencée est tout d’abord convertie en une nouvelle molécule synthétique transformant chaque base par une séquence nucléotidique spécifique correspondante. A chacune des quatre signatures nucléotidiques correspond un « molecular beacon » complémentaire fluorescent initialement inactivé par un système de « quencher ». Leurs hybridations au brin néoformé aboutit à un ADN double brin. la molécule est dirigé vers les nanopores par des échanges ioniques et en raison de la taille des orifices, les « molecular beacon » sont contraints à se deshybrider libérant cette fois une fluorescence, lue par un capteur photographique de type CMOS, qui est traduite en séquence.
La molécule d’ADN unique native destinée à être séquencée est tout d’abord convertie en une nouvelle molécule synthétique transformant chaque base par une séquence nucléotidique spécifique correspondante. A chacune des quatre signatures nucléotidiques correspond un « molecular beacon » complémentaire fluorescent initialement inactivé par un système de « quencher ». Leurs hybridations au brin néoformé aboutit à un ADN double brin. la molécule est dirigé vers les nanopores par des échanges ioniques et en raison de la taille des orifices, les « molecular beacon » sont contraints à se deshybrider libérant cette fois une fluorescence, lue par un capteur photographique de type CMOS, qui est traduite en séquence.
L’ensemble des études et preuves de faisabilité sur lesquelles repose la technologie de séquençage « optipore » sont décrites et détaillées au travers de l’article libre d’accès ci dessous: A ce jour, peu de caractéristiques techniques liées à cette plateforme filtrent mais Frank Feist, cofondateur de la société, annonce une capacité de séquençage de 500 Gb/heure. De plus, en raison de l’étape de conversion initiale, la taille des fragments serait limitée à 200 bases.
A ce jour, peu de caractéristiques techniques liées à cette plateforme filtrent mais Frank Feist, cofondateur de la société, annonce une capacité de séquençage de 500 Gb/heure. De plus, en raison de l’étape de conversion initiale, la taille des fragments serait limitée à 200 bases.
Un soutien de 4,2 millions de dollars en septembre dernier de la part du National Human Genome Research Institute devrait permettre à la société américaine de conforter cette avancée prometteuse.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages