Au cours des dix dernières années, la génomique connait une avancée technologique indéniable au travers des différents procédés de séquençage à haut-débit de deuxième génération. Néanmoins, certaines limites techniques subsistent, notamment par rapport à la quantité d’ADN requit, impliquant donc son extraction à partir de plusieurs millions de cellules. Cette contrainte implique une dilution de l’information pour des cellules aux fonctions biologiques bien souvent hétérogènes jusqu’au sein d’un même tissu.
En attendant la démocratisation du séquençage de troisième génération (Séquençage ADN sans amplification clonale) aux caractéristiques techniques qui permettraient un virage vers la génomique à l’échelle de la cellule unique , des méthodes alternatives appliquées à la seconde génération se développent afin d’accéder à cette hétérogénéité cellulaire. Ces solutions s’accompagnent donc, en amont du séquençage, d’une inévitable amplification de l’ADN de la cellule, préalablement isolée soit microdissection laser, système de microfluidique (ex: C1 Fluidigm), cytométrie en flux, ou encore micropipettage.
Ce poste est donc l’occasion de présenter la méthode d’amplification MALBAC, pour Multiple Annealing and Looping Based Amplification Cycles (Science, Zong et al.).
La méthode MALBAC repose sur l’utilisation de primers spécifiques (séquence de 8 nucléotides variables s’hybridant aléatoirement sur l’échantillon, couplée à une séquence connue de 26 nucléotides) générant des amplicons aux extrémités complémentaires. Cette particularité favorise la formation d’une boucle, évitant ainsi aux brins néo-synthétisés de servir à nouveau de matrice à la PCR et d’engendrer un biais d’ amplification (contrairement à la MDA). Ces étapes d’amplifications quasi-linéaires sont répétées cinq fois, puis les amplicons sont amplifiés par PCR exponentielle classique, en amont du séquençage.

MALBAC favorise une meilleure couverture de séquençage ainsi qu’une amplification plus uniforme (cf représentation ci-dessous) . De par ses performances, elle surclasse les méthodes conventionnelles tel que PEP-PCR, DOP-PCR ou encore MDA, pour Multiple Displacement Amplification, méthode la plus répandue depuis dix ans. Jusqu’à 83% de couverture à 10X de profondeur contre 45% pour la MDA. Parmi les autres atouts, la quantité d’ADN initiale requise n’est que de 0.5pg (contre 1000pg pour la MDA) et la polymérase Bst associée connait un taux d’erreur de 1/10000 bases.
Les performances de cette méthode permettent ainsi de reconsidérer les études génomiques ciblant un matériel biologique rare. Parmi elles, l’analyse de cellules tumorales circulantes, de tissus microdisséqués, de cellules embryonnaires, de micro-organismes, de cellules foetales circulantes, , etc…
La paléontologie, la science des fossiles et des traces de vie du passé, use de méthodes de biologie moléculaire de pointe qui pallient les effets du temps qui passe…
Afin d’introduire ce premier article traitant de paléogénomique, les moyens de la biologie moléculaire au service de la paléontologie, une vidéo amuse-bouche (Auteur(s) : Eva-Maria Geigl, Réalisation : Samia Serri, Production : Université Paris Diderot, Durée : 17 minutes 40 secondes) est disponible en usant du fameux clic gauche sur la capture d’image ci-dessous. Cette vidéo vaut surtout pour l’accent mis sur les précautions indispensables pour l’étude d’un échantillon précieux fossilisé… et dont l’ADN, peu abondant, peut être fragmenté. En outre, des mesures simples mais draconiennes permettent de limiter les sources de contaminations, quand l’ADN moderne peut polluer l’ADN fossile. Le port de sur-chausses, de masque et les changements de blouses, le non croisement des échantillons avant et après amplification sont autant de précautions mises en avant dans cette vidéo… une occasion de visiter virtuellement les laboratoire de l’Institut Jacques Monod.
L’une des problématiques liées à l’étude de l’ADN « fossile » réside dans sa faible quantité disponible. Plusieurs méthodes de biologie moléculaire ont été envisagées pour amplifier ce matériel génétique afin d’en permettre l’expertise. Une publication dans BMC Genomics de 2006, Assessment of whole genome amplification-induced bias throughhigh-throughput, massively parallel whole genome sequencing, relate la comparaison de 3 méthodes d’amplifications pan-génomiques (méthodes WGA pour Wide Genome Amplification) d’ADN qui pourra devenir ensuite la matrice suffisante d’un séquençage haut-débit.
– la PEP-PCR (Primer Extension Preamplification-PCR) : cette technique fait intervenir des amorces aléatoires aux conditions d’appariement à basse température (low melting temperature) qui initieront la PCR
référence : Zhang, L. et al. (1992) Whole genome amplification from a single cell: Implications for genetic analysis. Proc. Natl. Acad. Sci. USA 89, 5847
– la DOP-PCR (Degenerate Oligonucleotide Primed-PCR) : cette technique, quant à elle, fait intervenir des amorces semi-dégénérées (de type : CGACTCGAGNNNNNNATGTGG) qui ont une température d’hybridation supérieure à celles utilisées dans la PEP-PCR
référence : Telenius, H. et al. (1992) Degenerate oligonucleotide-primed PCR: general amplification of target DNA by a single degenerate primer. Genomics 13, 718.
L’utilisation d’une Taq PCR limite la taille des fragments néo-synthétisés qui ne dépassent guère 3 kb. En outre, ces deux techniques, à l’instar de ce qui peut être démontré dans la publication de BMC Genomics 2006 (mentionnée ci-dessus), induisent des erreurs de séquences accompagnées de nombreux biais d’amplification (certaines régions ne sont pas amplifiées au profit de régions qui deviennent, de fait, sur-représentées).
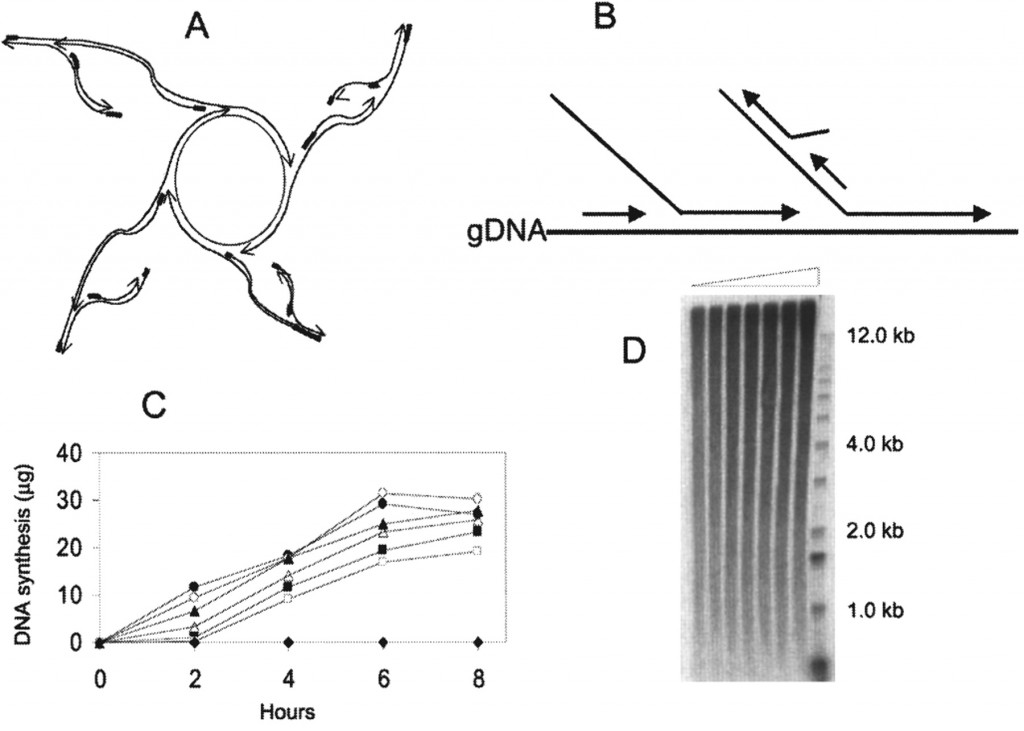
– la MDA (Multiple Displacement Amplification) : cette amplification iso-thermique fait intervenir des amorces aléatoires de type hexamères et une enzyme, la phi29. Le type d’amplification générée est schématisées sur la figure ci-dessous. L’enzyme surfe à partir du brin néo-synthétisé, déplace un brin complémentaire pour continuer sa synthèse. Ainsi, les brins générés par cette technique peuvent atteindre 100 kb. En outre, la phi29 possède une activité 3′ -> 5′ de relecture (proofreading) lui conférant un taux d’erreur 100 fois moindre que ceux constatés pour des Taq polymérases classiquement utilisées dans les techniques de PEP- ou DOP-PCR
source : Cold Spring Harb Protoc 2011.2011: pdb.prot5552 (la légende originale de la figure est disponible en cliquant sur celle-ci)
Ces techniques d’amplification pangénomique ont rendu possible l’étude d’ADN anciens et peu abondants et tout naturellement elles ont trouvé leur place dans la boîte à outils moléculaires des paléogénéticiens. Cependant, la révolution des séquençages haut-débit (dont nous avons abordé le sujet à plusieurs reprises) laisse entrevoir un nouveau champ des possibles pour l’étude des ADN fossiles. Au fond, des technologies telles que celle développée par Helicos Biosciences, trouvent ici un réel champ d’application à l’instar de ce que développe la publication True single-molecule DNA sequencing of a pleistocene horse bone de Genome Research, 2011- nulle nécessité d’amplifier la matrice de départ. Cette publication compare des technologies de séquençage de 2ème et 3ème générations (GaIIx et Helicos) appliquées au séquençage de l’ADN isolé à partir d’un os de cheval pleistocène conservé dans permafrost. Le séquençage « single molecule« , une chance pour la paléogénomique !
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages