 Que feriez vous avec un séquenceur qui tient dans la paume de votre main… ? Alors qu’il y a quelques années était annoncée l’arrivée de troisième génération de séquenceur, toujours plus sensibles (permettant de séquencer l’ADN natif, non pré-amplifié comme cela peut être le cas dans les technologies de séquençage de 2ème génération), générant des reads toujours plus longs, entachés de beaucoup plus d’erreurs… C’est aujourd’hui, avec le séquençage Minion de Nanopore que se pose réellement la question du changement d’applications qu’induirait le fait de posséder ce type de technologies.
Que feriez vous avec un séquenceur qui tient dans la paume de votre main… ? Alors qu’il y a quelques années était annoncée l’arrivée de troisième génération de séquenceur, toujours plus sensibles (permettant de séquencer l’ADN natif, non pré-amplifié comme cela peut être le cas dans les technologies de séquençage de 2ème génération), générant des reads toujours plus longs, entachés de beaucoup plus d’erreurs… C’est aujourd’hui, avec le séquençage Minion de Nanopore que se pose réellement la question du changement d’applications qu’induirait le fait de posséder ce type de technologies.
Cet article paru ce mois-ci dans la revue Médecine/Sciences, invite à réfléchir sur les conséquences de l’introduction de cette technologie en milieu hospitalier : Séquençage par nanopores – Perspectives d’applications en santé humaine essaie de faire le tour de la question.

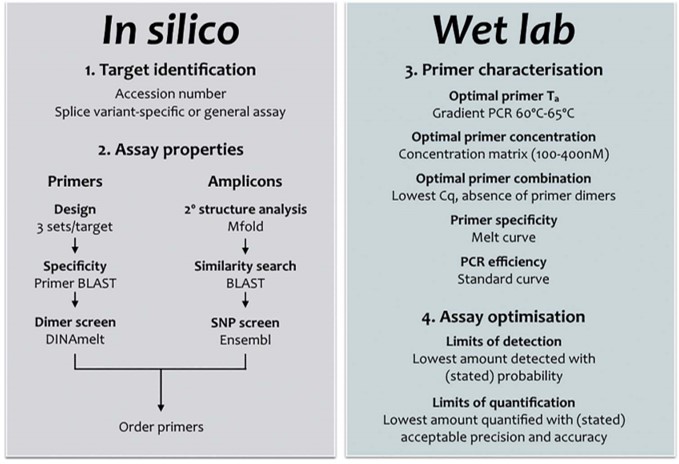
La qPCR est une méthode permettant de doser la quantité d’acides nucléiques ciblés introduits dans une réaction de PCR. Pour des raisons de rapidité, de sensibilité et de coût, souvent l’option de travailler avec un agent intercalant (sans sonde) est choisie. La bonne vieille qPCR SybGreen nécessitant le seul design d’une paire d’amorces…
Simple ? Pas nécessairement, car cette approche, certainement plus que la version qPCR Taqman nécessite un travail in silico et de validation/optimisation expérimentales comme passages obligés. C’est ce que montre la publication de Stephen Bustin et Jim Huggett dans Biomolecular Detection and Quantification. Cette publication incontournable pour les férus de qPCR SybrGreen est un beau travail pour lequel la publication vous est mise à disposition en cliquant ci-dessous. On attend ardemment une déclinaison Taqman, HRM, MolecularBeacon de ce type de revues permettant de formaliser des procédures visant à optimiser l’approche d’optimisation.
La quantification par qPCR SybrGreen suppose une relation linéaire entre le logarithme de la quantité initiale introduite en PCR et la valeur Cq obtenue lors de l’amplification. Ceci permet de calculer l’efficacité d’amplification d’un test et de borner ses limites de détection et de quantification. Les caractéristiques d’un test qPCR (bien) optimisé sont les suivantes:
• Une excellentissime spécificité révélée par un pic unique lors de l’établissement de la courbe de fusion
• Une efficacité d’amplification élevée (95-105%)
• Une courbe étalon linéaire (R2 > 0,980)
• Une bonne répétabilité
• Peu ou prou de dimères d’amorces
Pour paraphraser la conclusion de l’article, afin de finir par convaincre de lire cet « essentiel » de la qPCR :
La conception, le design d’une PCR est souvent au cœur de tout projet de recherche visant à quantifier les acides nucléiques. Il doit être réalisé avec soin, mais peut être simplifié en suivant un flux de travail simple, comme décrit ci-dessus (cf. diagramme workflow design qPCR).
Cela signifie généralement une spécificité absolue, l’absence de structures en épingle à cheveux ou de potentielles dimérisations croisées. Une bonne conception des essais doit tenir compte de la structure de l’amplicon (paramètre souvent négligé) et veiller à ce que les cibles de l’amorce soient exempts de structure secondaire. Il existe de nombreuses opinions et lignes directrices; une recherche sur Internet pour les termes « qPCR Assay Design » renvoie 695.000 pages. Cependant, bon nombre de ceux-ci sont basés sur des mythes ou peuvent être appropriés pour la PCR mais nécessitent des modifications subtiles (ou moins subtiles) pour être utilisés pour développer une qPCR. Chaque « nouveau » dosage doit être correctement validé, la validation in silico servant de filtre initial pour éliminer des designs ne permettant pas d’aboutir à une bonne qPCR. L’optimisation et la validation empirique sont une partie essentielle, mais souvent négligée, de toute expérience qPCR. Cela s’applique aussi bien aux essais nouvellement conçus qu’aux essais obtenus en reprenant des amorces issues d’une publication, par exemple. Avec tant d’essais prêts à l’emploi, on peut se demander pourquoi quelqu’un voudrait se donner la peine de concevoir un autre essai. D’autant plus que l’on a l’impression que la conception de son propre test est beaucoup plus complexe et peu commode que de simplement l’acheter à un fournisseur commercial, qui en tout cas aura validé chacun de ses tests. Cette perception est erronée pour deux raisons:
1° il se peut que les amorces commerciales ou les conditions d’analyse n’aient pas été validées ou optimisées de façon expérimentale.
2° on ne peut pas présumer qu’un ensemble d’amorces produira les mêmes résultats dans des conditions expérimentales différentes puisque la performance du dosage peut varier selon les méthodes d’extraction utilisées pour purifier les acides nucléiques.
 Voici une initiative originale conciliant deux tendances du moment : étude d’un microbiote & le concept de smart city. Saugrenu ? Science tendance ? Projet formaté pour la vulgarisation, la valorisation médiatique ? Quoi qu’il en soit, ce projet nous est plutôt bien vendu, à grand renfort d’infographies, de photographies, de vidéos un peu inquiétantes de personnes en train de réaliser des prélèvements de surface dans le métro. Derrière la communication, l’idée du projet est séduisante : utiliser des données biologiques, en l’occurrence des profils de l’ADNr 16S ou des séquençages génomes entiers définissant un microbiote pour améliorer nos écosystèmes urbains. Il est vrai qu’à une époque où l’on conçoit du mobilier urbain volontairement inconfortable pour ne pas que le passant puisse faire autre chose que passer, il ne semble pas intuitif d’aller chasser le microbiote pour concevoir une ville moins idiote. Même si la ville est le terrain de tous les paradoxes, il est captivant d’observer que cet environnement quotidien, banal, renferme une part de mystère… mystère qui serait à l’origine des odeurs du métro ? quelles espèces bactériennes propres à New York, Paris ou Rome sont à découvrir… quelle ville a le microbiote le plus diversifié ? quelle est celle qui aura le privilège d’héberger le plus de bactéries résistantes aux antibiotiques, la plus propre ou la plus sale ?
Voici une initiative originale conciliant deux tendances du moment : étude d’un microbiote & le concept de smart city. Saugrenu ? Science tendance ? Projet formaté pour la vulgarisation, la valorisation médiatique ? Quoi qu’il en soit, ce projet nous est plutôt bien vendu, à grand renfort d’infographies, de photographies, de vidéos un peu inquiétantes de personnes en train de réaliser des prélèvements de surface dans le métro. Derrière la communication, l’idée du projet est séduisante : utiliser des données biologiques, en l’occurrence des profils de l’ADNr 16S ou des séquençages génomes entiers définissant un microbiote pour améliorer nos écosystèmes urbains. Il est vrai qu’à une époque où l’on conçoit du mobilier urbain volontairement inconfortable pour ne pas que le passant puisse faire autre chose que passer, il ne semble pas intuitif d’aller chasser le microbiote pour concevoir une ville moins idiote. Même si la ville est le terrain de tous les paradoxes, il est captivant d’observer que cet environnement quotidien, banal, renferme une part de mystère… mystère qui serait à l’origine des odeurs du métro ? quelles espèces bactériennes propres à New York, Paris ou Rome sont à découvrir… quelle ville a le microbiote le plus diversifié ? quelle est celle qui aura le privilège d’héberger le plus de bactéries résistantes aux antibiotiques, la plus propre ou la plus sale ?

En juillet 2017, Stockholm accueillera la 3ème conférence annuelle « Metagenomics and Metadesign of Subways and Urban Biomes (MetaSUB)« , qui rassemblera des chercheurs dédiés à la cartographie du métagénome urbain de plus de 67 villes à travers le monde (dont Paris et Marseille). Ce projet ambitieux a débuté il y a deux ans, alors que le professeur Christopher Mason et son équipe ont réalisé la première étude sur la microflore de surface, dressant le microbiome de la ville de New York. Avec ces données moléculaires essentiellement basées sur le séquençage haut-débit 16S, le projet a étudié la façon dont ce «microbiome urbain» change avec des variables telles que la météo, la propreté, les matériaux de construction et même les niveaux socio-économiques du quartier. L’équipe a cherché à établir des profils de microbiotes dans le métro, à identifier les bio-menaces potentielles et à fournir des données complémentaires qui peut être utilisé par la ville pour créer une «ville intelligente», c’est-à-dire qui agrège des données hétérogènes pour améliorer son urbanisme.
Près de la moitié (48%) de l’ADN séquencé ne correspondait pas aux organismes connus, soulignant qu’un microbiota incognita entoure les usagers du métro. Le projet du métro de New York n’était que le début, ce qui a conduit à la création d’un consortium international de laboratoires pour établir une « cartographie » mondiale de microbiomes dans les systèmes de transport en commun notamment. En cliquant sur la capture d’écran ci-dessous, on pourra se faire une idée de la communication « Metasubienne » qui n’a rien à envier à celle de Tara Oceans ou de MetaHit (qui sont toutes trois, d’excellentes communications autour d’un projet et d’un consortium).
Voici en quelques mots les promesses annoncées dans l’article Geospatial Resolution of Human and Bacterial Diversity with City-Scale Metagenomics (Cell Syst, 2016) :
« La région métropolitaine de New York City (NYC) est un endroit idéal pour entreprendre une étude métagénomique à grande échelle car c’est la plus grande et la plus dense des États-Unis; 8,2 millions de personnes vivent sur une masse continentale de seulement 755 km2. En outre, le métro de New York est le plus grand système de transport en commun dans le monde (par le nombre de stations), qui s’étend sur plus de 406 km et utilisé par 1,7 milliard de personnes par an. Ce vaste écosystème urbain est une ressource précieuse qui nécessite un suivi pour le maintenir et le sécuriser contre les actes de bioterrorisme, les perturbations environnementales ou les épidémies. Ainsi, nous avons cherché à caractériser le métagénome de NYC en examinant le matériel génétique des microorganismes et d’autres ADN présents dans, autour et au-dessous de New York, en mettant l’accent sur les métros et les zones publiques très empruntés. Nous envisageons cela comme une première étape vers l’identification des menaces biologiques potentielles, la protection de la santé des New-Yorkais et la mise à disposition de données moléculaires qui pourront être utilisée par la ville pour créer une «ville intelligente», c’est-à-dire celle qui utilise des données de grande dimension pour améliorer l’urbanisme, la gestion de l’environnement bâti, des transports en commun et de la santé humaine. »
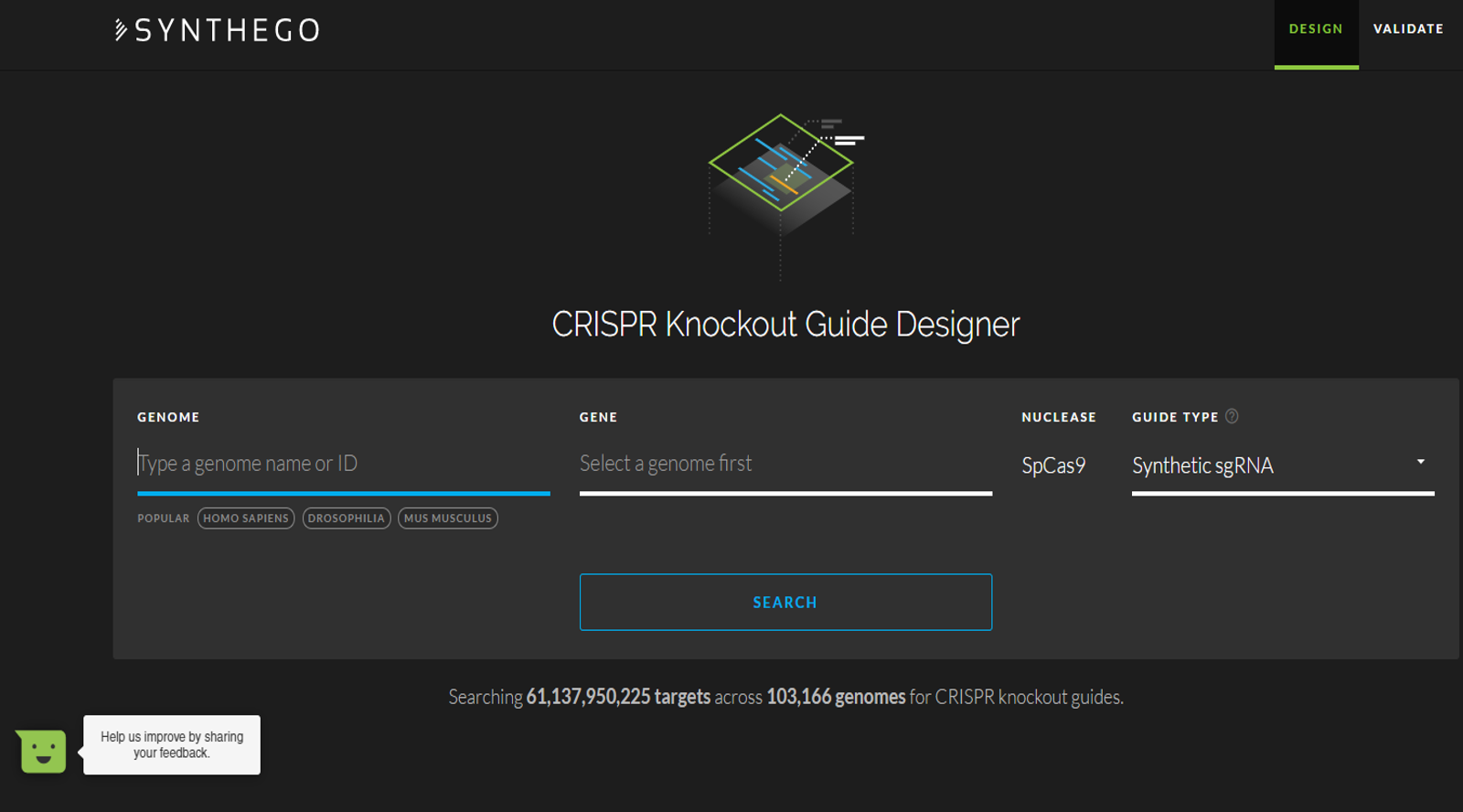
Voici un outil simplissime, rapide, en ligne, permettant de designer et de qualifier informatiquement des sgRNA, dans un contexte de KO réalisé par CRISPR/Cas9.
Vraiment pas mal !
Cliquez sur la capture d’écran ci-dessous et tentez votre design.
 Le London Calling a été l’occasion pour Oxford Nanopore Technologies (ONT) de frimer un peu avec des annonces et une gamme de séquenceurs ciblant des marchés très différents. Cette technologie de rupture risque d’être un tsunami technologique pour finir par déferler dans nos vies, car avec un séquenceur qui tient dans la poche et se connecte à un smartphone, ce qui était hier science fiction devient réalité.
Le London Calling a été l’occasion pour Oxford Nanopore Technologies (ONT) de frimer un peu avec des annonces et une gamme de séquenceurs ciblant des marchés très différents. Cette technologie de rupture risque d’être un tsunami technologique pour finir par déferler dans nos vies, car avec un séquenceur qui tient dans la poche et se connecte à un smartphone, ce qui était hier science fiction devient réalité.
ONT a fourni, en ce début de mois de mai 2017, informations concernant les développements technologiques et les perspectives de commercialisation. L’une d’elles, concerne une nouvelle « flow cell » pour MinIon, appelée Flongle (Flow Cell Dongle) pour les applications de diagnostics cliniques. Le développement du Flongle aidera également le travail de l’entreprise sur SmidgION, un séquenceur miniature avec de petites flow cells alimentées par un téléphone mobile (voir la photo ci-dessous).
Clive Brown, responsable technologique d’ONT, a donc présenté diverses mises à jour lors du « London Calling 2017 », le grand barnum des utilisateurs de la technologie, en ce début de mois de mai. En ce qui concerne le séquenceur MinION, Brown a déclaré que la technologie est d’ores et déjà capable de délivrer plus de 20 Gb de données par run de 48 h. (Actuellement, les utilisateurs sont plus autour des 15 Gb, ce rendement plus limité serait partiellement lié à la préparation de la bibliothèque, en particulier la quantification appropriée de la taille et de la quantité d’ADN de départ, selon Brown).

En mars, la société avait parlé d’une nouvelle méthode de séquençage, appelée séquençage 1D2, où les deux brins d’une librairie bicaténaire sont poussés par nanopore séquentiellement sans être physiquement connectés. La méthode contourne un brevet détenu par Pacific Biosciences et aboutit à des lectures plus précises que la seule lecture 1D, où seul un brin de la librairie constituée est séquencé. ONT prévoit de publier le kit de séquençage 1D2 courant mois de mai 2017, ainsi qu’une nouvelle chimie appelée R9.5. Les kits 2D ne sont plus disponibles et l’entreprise vient juste d’interrompre les anciennes cellules R9.4.
Dans l’ensemble, la précision de lecture brute est maintenant supérieure à 90 % pour la chimie R9.4 et supérieure à 95 % pour la R9.5 (en mode 1D2), ces deux chimies tournant à 450 bases / min.
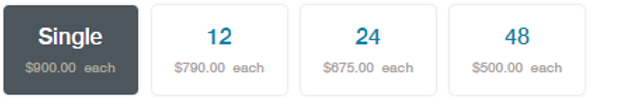
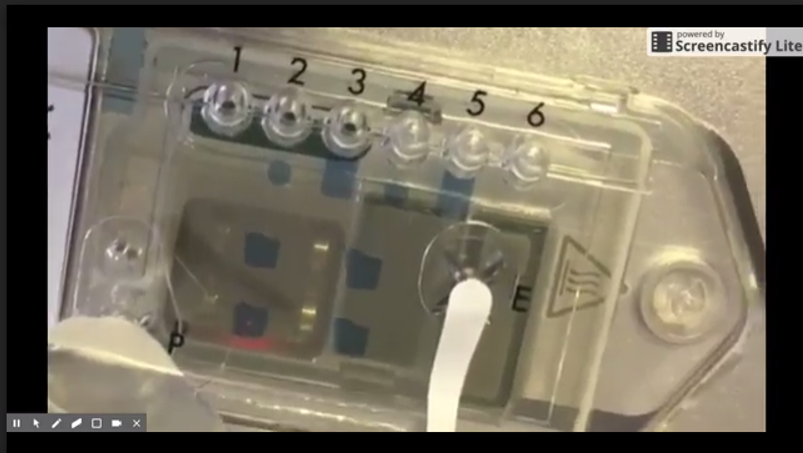
Ci-dessus, les prix actuels de la chimie 9.5. Là où l’on constate que le prix décroit drastiquement avec la quantité commandée… je pense qu’une mutualisation ou la création d’une centrale d’achats s’imposent ! Avec cette nouvelle chimie,ONT a diffusé une nouvelle version, la 1.6, de son logiciel MinKnow ainsi que de son basecaller Albacore (maintenant en 1.1). En outre, ONT a récemment commercialisé des kits de séquençage direct d’ARN et diffusé un pipeline pour réaliser le profil de résistance aux antibiotiques appelé ARMA (an analysis workflow for identification of antibiotic-resistant microorganisms in real time). À partir d’août, la société prévoit d’envoyer des flow cells à la température ambiante, des développements sont en cours pour allonger leur date limite d’exploitation (aujourd’hui une flow cell reçue doit être utilisée sous 8 semaines). Toujours visant le marché de l’utra-portabilité, en visant la décentralisation de l’acte de séquençage, ONT développe également un petit module de calcul pour le basecalling qui ferait environ la moitié de la taille du séquenceur MinION et pouvant y être directement connecté. Sur ce volet de la portabilité, le dispositif de préparation d’échantillons, VolTrax (voir vidéo ci-dessous), d’Oxford Nanopore est maintenant entre les mains de plus de 50 utilisateurs qui ont récemment reçu leurs premiers kits. La société développe actuellement un kit à base de transposase rapide et un kit d’indexation 4-plex rapide.
Les chercheurs de l’entreprise travaillent déjà sur une nouvelle version, VolTrax V2, qui devrait être disponible à la fin de 2017. Cette version permettra la PCR, la quantification des échantillons et le contrôle de la qualité des échantillons, tout en utilisant les mêmes aimants et appareils de chauffage que la version actuelle. Il sera également capable de gérer plus d’échantillons et d’exécuter des protocoles de préparation d’échantillons plus complexes. Il sera livré avec un chargeur de réactif avec des réactifs lyophilisés (lyophilisés, c’est mieux, parce que le séquenceur qui tient dans la main c’est bien, mais s’il vous faut un congélateur pour réaliser, sur le terrain, la moindre réaction…)
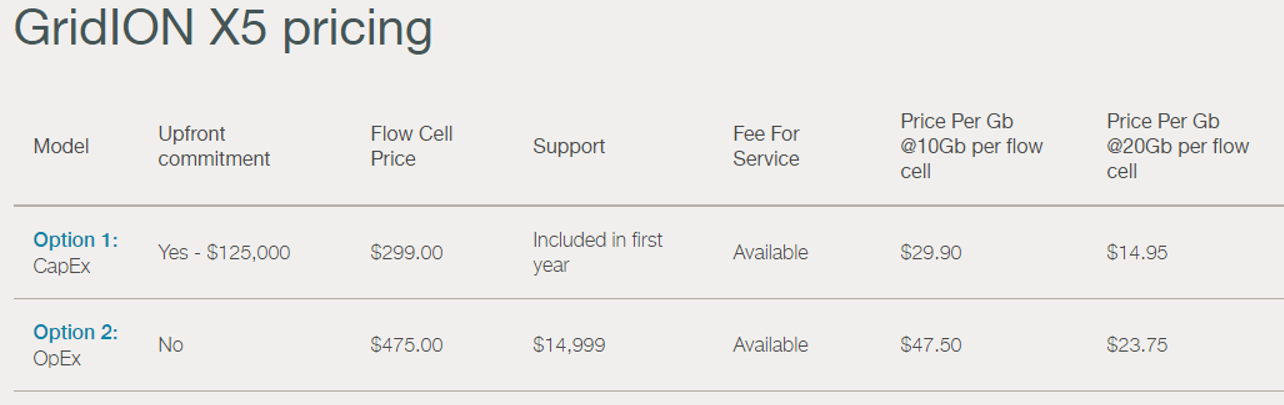
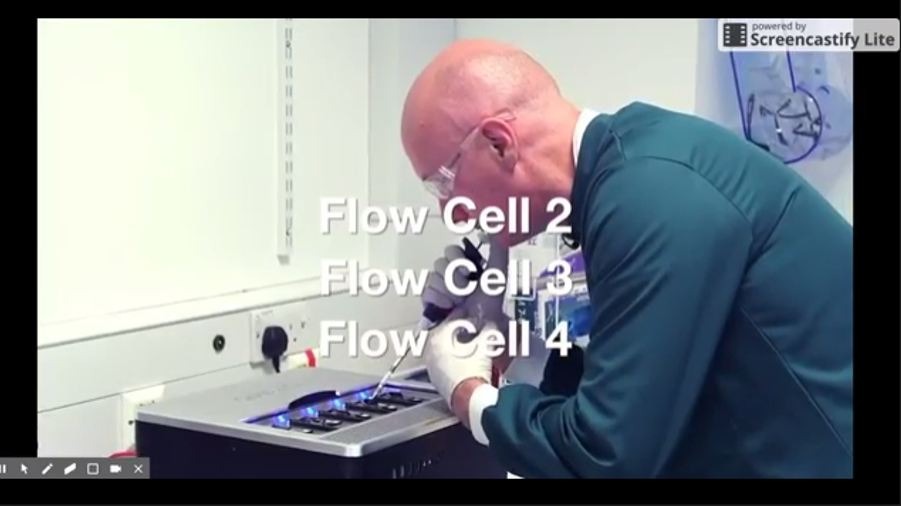
En mars, Oxford Nanopore a annoncé le lancement du GridIon X5, un séquenceur nanopore de bureau (et non plus de poche comme le MinION) pouvant permettre de travailler jusqu’à 5 flow cells à la fois et disposera d’un débit de 100 Gb par run de 48 heures. La plate-forme est livrée avec un cluster local de calcul haute performance qui permet le basecalling en temps réel ainsi que l’analyse des données. La première unité a été expédiée en début de semaine (en atteste la photo ci-dessous)

La société a récemment utilisé le GridIon pour séquencer un génome humain à 20 X, en utilisant 5 flow cells et la chimie R9.4 (en mode 1D).
Le GridION X5 permet d’exécuter simultanément ou individuellement jusqu’à cinq expériences; Les utilisateurs peuvent choisir d’utiliser tout ou partie de cette ressource à tout moment. La version actuelle de la chimie et du logiciel permet de générer jusqu’à 100 Gb de données pendant une exécution GridION X5 et le module de calcul peut analyser ces données en temps réel.
En utilisant la même technologie de base que le MinION et PromethION, le GridION X5 offre la possibilité de séquencer ADN et ARN en temps réel (dans la vidéo ci-dessous Clive Brown se transforme en Pipetman pour la promotion du GridION) :
Oxford Nanopore devait envoyer il y a peu les premiers consommables pour PromethION permettant de délivrer 50 Gb et pourraient en générer jusqu’à 120 Gb /jour dans un futur proche. En vitesse de croisière cette configuration sera capable de générer plus de données que le NovaSeq d’Illumina. Du lourd, du gros et du très petit : de quoi satisfaire les plateformes de séquençages et bientôt certainement des utilisateurs non touchés actuellement par la technologie aujourd’hui, puisque la version ci-dessous permet de préparer une librairie et de la séquencer avec quelques breloques qui tiennent dans le creux d’une main !

 Un slogan, une baseline : « nous avons cartographié le monde, maintenant cartographions la santé humaine » annonce la volonté de Google de persévérer dans le domaine de la santé humaine. C’est ainsi qu’Alphabet le conglomérat appartenant à et détenant Google tout à la fois, propose de constituer une cohorte humaine phénotypiquement caractérisée le plus finement possible : Verily (anciennement Google Life Sciences) une filiale d’Alphabet spécialisée dans la santé, a annoncé mercredi 19 avril qu’elle souhaitait recruter 10 000 volontaires pour son projet Baseline, annoncé en 2014 et déjà testé sur une centaine de volontaires. S’adjoignant des chercheurs du monde académique avec la participation de l’université de Duke (Caroline du Nord) et de l’université Stanford, Google vise à collecter des données de santé très précises sur ces personnes pendant plusieurs années. Assurément le nombre de personnes visées est pour l’instant moindre que la célèbre cohorte Nurses’ Health Study débutée en 1976 et dénombrant 280.000 participants, l’innovation consiste en la qualité des données collectées par l’intermédiaire de capteurs connectés. Les études épidémiologiques faisant intervenir des cohortes ne sont certes pas nouvelles, elles sont un outil formidable, grandes pourvoyeuses de résultats scientifiques valorisables (la célèbre cohorte de Framingham enregistre plusieurs centaines de publications). Dans le cas précis du projet Baseline, ce qui est nouveau est la promotion de ce type d’approche par et pour aussi un peu, une entité privée. Google, enfin Alphabet, peu importe finalement, est un conglomérat qui possède notamment la société de biotechnologie Calico, et qui a des liens capitalistiques avec 23andMe et possède donc encore Verily dont certains projets consistent en :
Un slogan, une baseline : « nous avons cartographié le monde, maintenant cartographions la santé humaine » annonce la volonté de Google de persévérer dans le domaine de la santé humaine. C’est ainsi qu’Alphabet le conglomérat appartenant à et détenant Google tout à la fois, propose de constituer une cohorte humaine phénotypiquement caractérisée le plus finement possible : Verily (anciennement Google Life Sciences) une filiale d’Alphabet spécialisée dans la santé, a annoncé mercredi 19 avril qu’elle souhaitait recruter 10 000 volontaires pour son projet Baseline, annoncé en 2014 et déjà testé sur une centaine de volontaires. S’adjoignant des chercheurs du monde académique avec la participation de l’université de Duke (Caroline du Nord) et de l’université Stanford, Google vise à collecter des données de santé très précises sur ces personnes pendant plusieurs années. Assurément le nombre de personnes visées est pour l’instant moindre que la célèbre cohorte Nurses’ Health Study débutée en 1976 et dénombrant 280.000 participants, l’innovation consiste en la qualité des données collectées par l’intermédiaire de capteurs connectés. Les études épidémiologiques faisant intervenir des cohortes ne sont certes pas nouvelles, elles sont un outil formidable, grandes pourvoyeuses de résultats scientifiques valorisables (la célèbre cohorte de Framingham enregistre plusieurs centaines de publications). Dans le cas précis du projet Baseline, ce qui est nouveau est la promotion de ce type d’approche par et pour aussi un peu, une entité privée. Google, enfin Alphabet, peu importe finalement, est un conglomérat qui possède notamment la société de biotechnologie Calico, et qui a des liens capitalistiques avec 23andMe et possède donc encore Verily dont certains projets consistent en :
- des lentilles de contact permettant de contrôler le niveau de glucose chez les personnes diabétiques
- des cuillères pour les personnes ayant des tremblements, par exemple atteintes de la maladie de Parkinson (projet Liftware)
- une plateforme permettant la détection de maladie par l’intermédiaire de nanoparticules
- un bracelet connecté permettant de suivre des paramètres liés à la santé
Certains de ces objets connectés auront une exposition majeure du fait de leur positionnement central dans la cohorte de Baseline. Ainsi une montre, au design contestable, complétera la panoplie de capteurs associée au projet. Le fameux capteur d’activités nocturnes qui se faufile jusque sous la couette peut laisser perplexe… Ces objets connectés, pourvoyeurs de données, de beaucoup de données, nécessiteront les méthodologies que Google s’attache à développer pour traiter de façon automatisée et optimale la manne visée par la cohorte Baseline ! En outre, ces objets trouveront un écho, à n’en pas douter, auprès de futurs consommateurs bien au-delà des personnes constituant la cohorte initiale. En effet, les modèles établis sur les personnes, population référente constitutive de la cohorte (pour lesquelles sont à disposition l’intégralité des données phénotypiques) pourront être appliqués à de futures personnes dotées de la batterie de capteurs mais n’ayant pour autant pas été caractérisées finement. La cohorte servirait à établir un modèle, modèle qui serait appliqué à de futurs utilisateurs des solutions connectées proposées par Google. Ainsi des prédictions de l’état de santé et pourquoi pas la mise en place d’un système d’alerte… pouvant aller jusqu’à signifier la nécessité de consulter pourraient trouver une application commerciale.
L’objectif qu’Alphabet revendique est de cartographier la santé humaine, cet objectif passe par la création d’une immense base de données avec la santé pour finalité, permettant, grâce aux nouvelles technologies, d’«explorer la santé en profondeur». Après anonymisation comme garantie de sécurité des données, cette base de données est destinée à être transmise à des chercheurs. Pour recruter ses volontaires et les inciter à rejoindre le projet (la stratégie de recrutement peut laisser entrevoir d’ailleurs quelques biais de recrutement), Verily assure qu’il s’agit d’une manière « de participer à la création de cette carte de la santé humaine, et de laisser durablement une trace», en contribuant à son échelle à la recherche médicale. Ces futurs participants pourront partager leurs données avec leur médecin. Verily cherche des personnes nord-américaines, en bonne santé qui seront suivies et invitées à subir, pendant deux jours, chaque année, une batterie de tests médicaux. Ces bilans médicaux accompagnés des données des bio-capteurs dont ils seront dotés seront agrégés sous forme de « Big Data », matière première dont Google et ses méthodologies sont devenus experts…
Plusieurs questions peuvent être soulevées :
- est ce que Google, entreprise commerciale, finira par monétiser ces données comme matière première à une recherche scientifique ?
- est ce que la stratégie de Google ne serait pas multiple : (i) faire la promotion de ces outils d’analyses sur une matière première (dont Google serait propriétaire) tout en (ii) faisant la promotion de ces objets connectés, surfant sur un marché de la e-santé, marché en pleine expansion, tout en (iii) rentabilisant son investissement pour « partager » ses données recrutées moyennant des contreparties soumises au secret ?
Par le passé, Google Health est – était- un service Internet d’archivage de dossiers médicaux pour les internautes américains, mis en place par Google en mars 2008. L’avantage reste celui de laisser le pouvoir aux pharmaciens américains de mettre à jour automatiquement les traitements ou encore de trouver un spécialiste pour traiter des maladies adaptées. Google fermera ce service le 1er janvier 2012, faute d’un nombre insuffisant d’utilisateurs. La volonté de Google de développer une offre trouvant des applications en santé, médecine de précision, auxiliaire personnel de santé n’est donc pas nouvelle. Aujourd’hui, dans un contexte américain où les fonds publics alloués à la recherche médicale se tarissent (lire l’article de Nature sur la coupe budgétaire sans précédents de Donald Trump), le temps des partenariats public/privé est peut être venu. Dans ce contexte, même si la démarche de Google est nappée de marketing, des projets tels que Baseline pourraient être favorablement accueillis par la communauté scientifique.
 Le développement des réseaux sociaux sur internet nous a permis d’appréhender la notion du « quand c’est gratuit, c’est toi le produit » en faisant référence, à des Facebook parmi d’autres, qui monnaient les informations personnelles des adhérents à leur service à des fins de profilage marketing. L’avènement des technologies à haut-débit en génomique a vu, il y a une dizaine d’années, émerger le concept de médecine personnalisée. Depuis, ce concept a évolué vers celui de médecine de précision (certainement plus consensuel et moins égotique). A partir de 2005, des sociétés privées se sont appropriées ce concept pour le faire déborder de la sphère de la recherche ou de la clinique pour proposer l’exploitation de profils génomiques à des fins de génomique récréative (23andMe, 2006). Un retour à l’égotisme en quelque sorte.
Le développement des réseaux sociaux sur internet nous a permis d’appréhender la notion du « quand c’est gratuit, c’est toi le produit » en faisant référence, à des Facebook parmi d’autres, qui monnaient les informations personnelles des adhérents à leur service à des fins de profilage marketing. L’avènement des technologies à haut-débit en génomique a vu, il y a une dizaine d’années, émerger le concept de médecine personnalisée. Depuis, ce concept a évolué vers celui de médecine de précision (certainement plus consensuel et moins égotique). A partir de 2005, des sociétés privées se sont appropriées ce concept pour le faire déborder de la sphère de la recherche ou de la clinique pour proposer l’exploitation de profils génomiques à des fins de génomique récréative (23andMe, 2006). Un retour à l’égotisme en quelque sorte.
23andMe a d’ailleurs un business model assez osé, consistant à vendre à des clients, leurs profils génomiques (données de puce à ADN) en échange de quelques indications concernant leur « pedigree génomique » (lire: 23 chromosomes, la NSA et moi) et quelques informations hautement critiquables. Ces dernières consistaient en des probabilités de susceptibilité à certaines afflictions (ce point là a suscité l’émoi de la FDA et ce service est aujourd’hui interrompu). En clair, 23andMe subventionne sa population de référence, ses bases de données génomiques par ses propres clients. En substance ceci constitue une évolution par rapport à Facebook, si l’on était taquin on pourrait abréger ceci en un : « vous payez pour devenir un produit ». Aujourd’hui, 23andMe habille le tout d’un « On average, a customer who consents to research contributes to over 230 studies« , se faire génotyper pour le bien de la recherche… pas loin de la servitude volontaire nappée de bon sentiment. Il est temps ici de rappeler ce que disait il y a peu un des plus grands promoteurs de la génomique personnelle : « Si je devais conseiller un jeune Craig Venter, je dirais, réfléchissez bien avant que vous veniez déverser votre génome sur Internet« . De l’aveu même de Craig Venter donc, il ne faudrait pas s’emballer avant d’envisager hypothéquer 50 % du patrimoine de ses propres enfants nés ou à naître sans qu’eux-mêmes n’aient eu mots à dire.
Aujourd’hui, arrive sur ce marché en pleine croissance, un nouvel acteur, Genos Research (page d’accueil ci-dessus). Ce dernier propose un nouveau business model : vous financez à raison de 399 USD votre propre séquençage d’exome (délivrant en moyenne 50 x fois plus de mutations que celles génotypées par l’intermédiaire des puces Illumina, telles que l’utilise 23andMe). Vous devenez propriétaire de vos données, à l’inverse des solutions concurrentes pour lesquelles, en général, vous en déléguez la propriété et l’utilisation future. Vous devenez donc le promoteur de votre propre information génomique que vous pouvez monnayer sur le marché de la recherche -priez pour avoir une maladie rare, ceci devrait accroître votre la cote de cette information. En outre, Genos permet aux utilisateurs de partager leurs données à travers leur plate-forme, ou d’exporter leurs données séquencées afin qu’ils puissent les transmettre à d’autres systèmes analytiques ou des conseillers génétiques –un business entier est d’ailleurs à bâtir : analyste de données personnelles génomiques. Genos est une société qui a bourgeonné à partir de Complete Genomics, une société de service de séquençage haut-débit, et a complété un financement privé soutenu par des entreprises en mai 2016. La société a  été co-fondée par Mark Blumling (l’ancien fondateur d’Hyperion Therapeutics, rachetée en mai 2015 par Horizon Pharma plc) et Clifford Reid (l’ancien fondateur et PDG de Complete Genomics, devenu une société de l’incroyable BGI’s company).
été co-fondée par Mark Blumling (l’ancien fondateur d’Hyperion Therapeutics, rachetée en mai 2015 par Horizon Pharma plc) et Clifford Reid (l’ancien fondateur et PDG de Complete Genomics, devenu une société de l’incroyable BGI’s company).
Pour le moins sensibles, que deviennent vos données génomiques au gré des acquisitions, fusions de sociétés de biotechnologie ? Ces changements de propriétaire sont monnaie courante et peuvent vous faire perdre de vue vos données qui pourraient finir par être le nouveau pétrole de l’ère post Donald Trump.
 A moins de sortir d’une longue phase d’hibernation, tout un chacun aura eu les oreilles rebattues par les applications liées à la technologie CRIPSR/Cas9, la révolution de l’édition de gènes (comprenez modification). Plus facile à mettre en oeuvre que les technologies TALEN et autres nucléases doigt de zinc (voir la table ci-dessous), cette technologie est tout à la fois moins onéreuse et nettement plus efficace. Cette technologie est souvent comparée, du fait de sa précision d’utilisation, à une méthode de micro-chirurgie. Un nombre exponentiel d’articles scientifiques et non scientifiques font le panégyrique de cette révolution qui est la conséquences des travaux de Jennifer Doudna et Emmanuelle Charpentier. Ces dernières ont permis de configurer cette technologie à partir de l’élucidation d’un mécanisme de vaccination primitif bactérien (nous reviendrons sur ce point fondamental dans un futur article).
A moins de sortir d’une longue phase d’hibernation, tout un chacun aura eu les oreilles rebattues par les applications liées à la technologie CRIPSR/Cas9, la révolution de l’édition de gènes (comprenez modification). Plus facile à mettre en oeuvre que les technologies TALEN et autres nucléases doigt de zinc (voir la table ci-dessous), cette technologie est tout à la fois moins onéreuse et nettement plus efficace. Cette technologie est souvent comparée, du fait de sa précision d’utilisation, à une méthode de micro-chirurgie. Un nombre exponentiel d’articles scientifiques et non scientifiques font le panégyrique de cette révolution qui est la conséquences des travaux de Jennifer Doudna et Emmanuelle Charpentier. Ces dernières ont permis de configurer cette technologie à partir de l’élucidation d’un mécanisme de vaccination primitif bactérien (nous reviendrons sur ce point fondamental dans un futur article).
| Technologie | Première utilisation | Première application sur animaux vivants | Echelle de temps pour la mise en œuvre |
| Zinc finger nucleases | 1996 | 2002 | mois / année |
| TALENs | 2010-2011 | 2011 | semaines |
| CRISPR/Cas9 | 2012 | 2012-2013 | jours |
Donc non ! il ne s’agit pas ici uniquement de contribuer à l’enthousiasme, certes communicatif, de cette révolution dans le domaine des biotechnologies qui est lui-même friand de ces révolutions qui se succèdent les unes aux autres… Il s’agit dans ce premier article abordant CRISPR/Cas9 de proposer un début de décryptage de ce qui se cache derrière la terminologie de Gene Drive, application de CRISPR/Cas9. Cette terminologie est encore une fois peu aisée à traduire à mi-chemin entre le pilotage et le forçage génétique. Quoi qu’il en soit derrière l’appellation Gene Drive, se cache une technologie qui mérite que l’on s’arrête pour pousser plus avant la réflexion, tant l’enfer est pavé de bonnes intentions… Pour faire monter la pression, il faut savoir que James Clapper, le directeur du renseignement national des États-Unis, a placé la technologie CRISPR mais surtout sa formulation « Gene Drive » au niveau des armes de destruction massive… rien que ça.
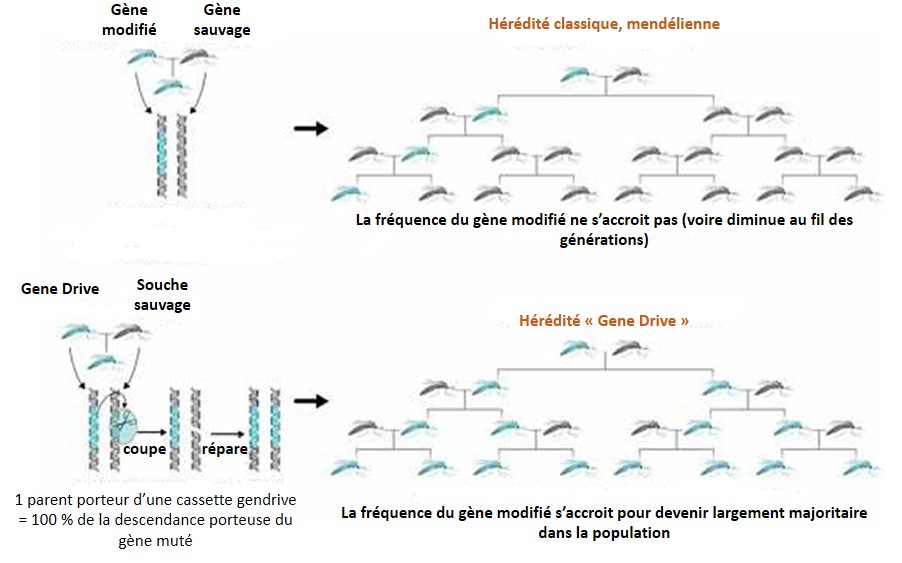
Gene Drive, technologiquement concentre dans une cassette, l’outil permettant d’accélérer sa propagation (les ciseaux permettant la microchirurgie sont embarqués et encodés au sein même du génome ciblé) ainsi que le gène que l’on souhaite voir introduire au niveau d’une espèce donnée, évidemment cette technologie s’intéresse principalement aux populations sauvages. Ainsi cette cassette possède sa propre capacité de propagation, échappant aux lois de Mendel. Correctement encodée au sein d’une partie de population « augmentée ou diminuée », une dizaine de générations d’individus se reproduisant de façon sexuée permet de faire en sorte que la cassette a contaminé l’intégralité ou l’extrême majorité de la population sauvage (voir le schéma ci-dessous). L’exemple souvent développé, sponsorisé par la Fondation Bill-et-Melinda-Gates, consiste à proposer aux dirigeants africains des anophèles modifiés par cette technologie pour être résistants au parasite, Plasmodium falciparum, l’agent du paludisme. Rappelons que 650 000 personnes meurent chaque année de la malaria (rapport OMS de 2013), principalement sur le continent africain. Toujours dans les bons coups, Bill, pour soigner son karma! Ce dernier oublie néanmoins que le moustique génétiquement modifié agent mutagène puisque porteur de la cassette Gene Drive ne s’arrête pas aux frontières… mais qu’importe le moyen technologique existe donc que ne serions nous pas de fâcheux obscurantistes à réfléchir aux conséquences de l’utilisation de cette technologie avant de l’employer la fleur au fusil. Les « obscures rabat-joie » souvent du côté des écologues s’opposent aux biologistes moléculaires, rapides prescripteurs d’une technologie prometteuse pour éradiquer un nombre substantiel de fléaux, quitte à modifier durablement la nature pour l’intérêt supérieur humain ou parfois même pour l’intérêt supérieur de quelques uns…
La technologie Gene Drive peut être utilisée pour :
• Éradiquer les maladies telles que le paludisme, la dengue, la fièvre jaune, virus du Nil occidental, la maladie du sommeil ainsi que beaucoup d’autres en modifiant les espèces d’insectes vecteurs des parasites, virus, bactéries causes de ces maladies
• Éradiquer les espèces envahissantes. Les dix premières espèces envahissantes aux États-Unis causent environ 42 milliards USD de dommages chaque année
• Certains évoquent avec beaucoup d’aplomb, l’utilisation de cette technologie pour une agriculture plus « durable » en inversant la résistance aux pesticides et aux herbicides. Cette fois c’est la plante résistante au glyphosate (la molécule du RoundUp) qui est ciblée.
Que de beaux et bons sentiments… en guise de promesses de cette technologie !
Les limites de la technologie :
• De « nombreuses » générations sont nécessaires pour répandre la mutation dans la population. La durée totale dépend du cycle de reproduction de l’organisme, du nombre d’individus porteurs de la cassette Gene Drive introduit initialement dans la population, de l’efficacité de la cassette et du flux génétique (transfert d’allèles d’une population à une autre). Par exemple, cela pourrait prendre quelques années pour modifier une population d’insectes. Si 10 individus d’une population étaient porteurs d’une cassette Gene Drive parmi une population constante de 100.000 organismes, il faudrait environ 16 générations – environ un an- pour se propager à 99% de la population sous de peu réalistes, conditions optimales.
• Cette technologie est inopérante sur des organismes ne reproduisant pas de façon sexuée comme les bactéries et les virus et aura des problèmes avec les espèces qui peuvent se reproduire de façon sexuée ou non, comme cela peut être le cas pour beaucoup de plantes.
• Certains types d’altérations devraient être réintroduites sans cesse. Par exemple, une cassette Gene Drive engendrant un trait qui est quelque peu nuisible à l’organisme finira par se « briser ». De même, une cassette engendrant une résistance aux herbicides inversée dans une mauvaise herbe aurait à lutter contre la sélection naturelle dans les zones où les herbicides ont été appliqués.
La technologie ouvre donc la boîte de Pandore de la modification génétique de population sauvage. Plus l’intervalle générationnel de la population cible sera court, plus l’objectif d’une population « panmutée » sera facile à atteindre. Ci-dessous vous pourrez avoir un excellent point concernant la technologie à l’aide du travail de « the National Academies of Sciences Engineering and Medicine ».
 Si vous vous intéressez au séquençage haut-débit, que vous souhaitez avoir un panorama des diverses technologies à disposition et que vous êtes friands de schémas de principe: la publication de Sara Goodwin, John D. McPherson & W. Richard McCombie. Cet article publié dans la revue Nature de mai 2016 promet de faire un retour en arrière sur 10 ans d’évolution du séquençage haut-débit. Elle parvient à tenir ses promesses et livre effectivement des schémas (avec la charte graphique « Nature ») très bien faits, très pédagogiques ! En outre, le tableau (Table 1 | Summary of NGS platforms) permet à tous les pourvoyeurs de projets nécessitant le recours à du séquençage, d’avoir un pense bête sous la main pour associer la bonne technologie à la question biologique qui leur incombe… Et comme vous êtes pressés, vous pourrez retrouver l’intégralité de cet article en vous promenant et cliquant sur l’image ci-dessous.
Si vous vous intéressez au séquençage haut-débit, que vous souhaitez avoir un panorama des diverses technologies à disposition et que vous êtes friands de schémas de principe: la publication de Sara Goodwin, John D. McPherson & W. Richard McCombie. Cet article publié dans la revue Nature de mai 2016 promet de faire un retour en arrière sur 10 ans d’évolution du séquençage haut-débit. Elle parvient à tenir ses promesses et livre effectivement des schémas (avec la charte graphique « Nature ») très bien faits, très pédagogiques ! En outre, le tableau (Table 1 | Summary of NGS platforms) permet à tous les pourvoyeurs de projets nécessitant le recours à du séquençage, d’avoir un pense bête sous la main pour associer la bonne technologie à la question biologique qui leur incombe… Et comme vous êtes pressés, vous pourrez retrouver l’intégralité de cet article en vous promenant et cliquant sur l’image ci-dessous.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages










Bioproduction: la France pas si mal que ça !
La carte obtenue par le LEEM – le syndicat du milieu pharmaceutique qui s’est substitué en 2002 au Syndicat national de l’industrie pharmaceutique (SNIP)- fait apparaître un regroupement de l’activité autour de quatre bassins principaux :
– la région Rhône-Alpes
– la vallée de la Seine
– l’Alsace
– le département du Nord
L’état des lieux de 2014 du LEEM est plutôt évocateur :
– La demande en biomédicaments est passée de 5 à 15 % entre 2000 et 2012.
– La France dispose notamment de capacités de production significatives dans le domaine des vaccins, des produits dérivés du sang, des hormones et des produits pour le diagnostic.
– 13 000 personnes sont employées dans la bioproduction
– Les formations françaises en biotechnologies sont reconnues au niveau international. Il en va de même pour le processus de reconversion des industries chimiques vers le vivant. Cette synergie de compétences est un atout.
Outre ces éléments visant à saluer une filière qui a su prendre un bon élan, d’un point de vue plus pratique, ce post est aussi l’occasion de fournir une liste de ces entreprises qui recherchent des profils plutôt jeunes et plutôt bien formés. Cette liste est évidemment non exhaustive et tirée de l’excellent recensement de l’Industrie Pharma Magazine.