Que feriez vous avec un séquenceur qui tient dans la paume de votre main… ? Alors qu’il y a quelques années était annoncée l’arrivée de troisième génération de séquenceur, toujours plus sensibles (permettant de séquencer l’ADN natif, non pré-amplifié comme cela peut être le cas dans les technologies de séquençage de 2ème génération), générant des reads toujours plus longs, entachés de beaucoup plus d’erreurs… C’est aujourd’hui, avec le séquençage Minion de Nanopore que se pose réellement la question du changement d’applications qu’induirait le fait de posséder ce type de technologies.
Que feriez vous avec un séquenceur qui tient dans la paume de votre main… ? Alors qu’il y a quelques années était annoncée l’arrivée de troisième génération de séquenceur, toujours plus sensibles (permettant de séquencer l’ADN natif, non pré-amplifié comme cela peut être le cas dans les technologies de séquençage de 2ème génération), générant des reads toujours plus longs, entachés de beaucoup plus d’erreurs… C’est aujourd’hui, avec le séquençage Minion de Nanopore que se pose réellement la question du changement d’applications qu’induirait le fait de posséder ce type de technologies.
Cet article paru ce mois-ci dans la revue Médecine/Sciences, invite à réfléchir sur les conséquences de l’introduction de cette technologie en milieu hospitalier : Séquençage par nanopores – Perspectives d’applications en santé humaine essaie de faire le tour de la question.
 Le London Calling a été l’occasion pour Oxford Nanopore Technologies (ONT) de frimer un peu avec des annonces et une gamme de séquenceurs ciblant des marchés très différents. Cette technologie de rupture risque d’être un tsunami technologique pour finir par déferler dans nos vies, car avec un séquenceur qui tient dans la poche et se connecte à un smartphone, ce qui était hier science fiction devient réalité.
Le London Calling a été l’occasion pour Oxford Nanopore Technologies (ONT) de frimer un peu avec des annonces et une gamme de séquenceurs ciblant des marchés très différents. Cette technologie de rupture risque d’être un tsunami technologique pour finir par déferler dans nos vies, car avec un séquenceur qui tient dans la poche et se connecte à un smartphone, ce qui était hier science fiction devient réalité.
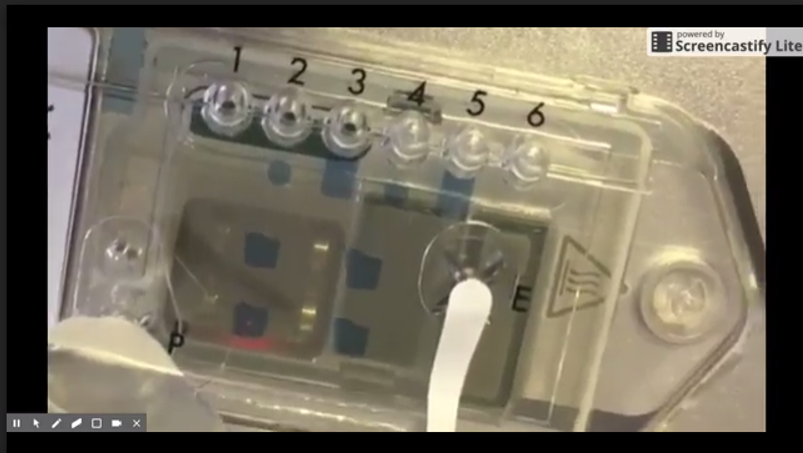
ONT a fourni, en ce début de mois de mai 2017, informations concernant les développements technologiques et les perspectives de commercialisation. L’une d’elles, concerne une nouvelle « flow cell » pour MinIon, appelée Flongle (Flow Cell Dongle) pour les applications de diagnostics cliniques. Le développement du Flongle aidera également le travail de l’entreprise sur SmidgION, un séquenceur miniature avec de petites flow cells alimentées par un téléphone mobile (voir la photo ci-dessous).
Clive Brown, responsable technologique d’ONT, a donc présenté diverses mises à jour lors du « London Calling 2017 », le grand barnum des utilisateurs de la technologie, en ce début de mois de mai. En ce qui concerne le séquenceur MinION, Brown a déclaré que la technologie est d’ores et déjà capable de délivrer plus de 20 Gb de données par run de 48 h. (Actuellement, les utilisateurs sont plus autour des 15 Gb, ce rendement plus limité serait partiellement lié à la préparation de la bibliothèque, en particulier la quantification appropriée de la taille et de la quantité d’ADN de départ, selon Brown).

En mars, la société avait parlé d’une nouvelle méthode de séquençage, appelée séquençage 1D2, où les deux brins d’une librairie bicaténaire sont poussés par nanopore séquentiellement sans être physiquement connectés. La méthode contourne un brevet détenu par Pacific Biosciences et aboutit à des lectures plus précises que la seule lecture 1D, où seul un brin de la librairie constituée est séquencé. ONT prévoit de publier le kit de séquençage 1D2 courant mois de mai 2017, ainsi qu’une nouvelle chimie appelée R9.5. Les kits 2D ne sont plus disponibles et l’entreprise vient juste d’interrompre les anciennes cellules R9.4.
Dans l’ensemble, la précision de lecture brute est maintenant supérieure à 90 % pour la chimie R9.4 et supérieure à 95 % pour la R9.5 (en mode 1D2), ces deux chimies tournant à 450 bases / min.
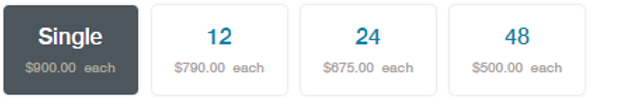
Ci-dessus, les prix actuels de la chimie 9.5. Là où l’on constate que le prix décroit drastiquement avec la quantité commandée… je pense qu’une mutualisation ou la création d’une centrale d’achats s’imposent ! Avec cette nouvelle chimie,ONT a diffusé une nouvelle version, la 1.6, de son logiciel MinKnow ainsi que de son basecaller Albacore (maintenant en 1.1). En outre, ONT a récemment commercialisé des kits de séquençage direct d’ARN et diffusé un pipeline pour réaliser le profil de résistance aux antibiotiques appelé ARMA (an analysis workflow for identification of antibiotic-resistant microorganisms in real time). À partir d’août, la société prévoit d’envoyer des flow cells à la température ambiante, des développements sont en cours pour allonger leur date limite d’exploitation (aujourd’hui une flow cell reçue doit être utilisée sous 8 semaines). Toujours visant le marché de l’utra-portabilité, en visant la décentralisation de l’acte de séquençage, ONT développe également un petit module de calcul pour le basecalling qui ferait environ la moitié de la taille du séquenceur MinION et pouvant y être directement connecté. Sur ce volet de la portabilité, le dispositif de préparation d’échantillons, VolTrax (voir vidéo ci-dessous), d’Oxford Nanopore est maintenant entre les mains de plus de 50 utilisateurs qui ont récemment reçu leurs premiers kits. La société développe actuellement un kit à base de transposase rapide et un kit d’indexation 4-plex rapide.
Les chercheurs de l’entreprise travaillent déjà sur une nouvelle version, VolTrax V2, qui devrait être disponible à la fin de 2017. Cette version permettra la PCR, la quantification des échantillons et le contrôle de la qualité des échantillons, tout en utilisant les mêmes aimants et appareils de chauffage que la version actuelle. Il sera également capable de gérer plus d’échantillons et d’exécuter des protocoles de préparation d’échantillons plus complexes. Il sera livré avec un chargeur de réactif avec des réactifs lyophilisés (lyophilisés, c’est mieux, parce que le séquenceur qui tient dans la main c’est bien, mais s’il vous faut un congélateur pour réaliser, sur le terrain, la moindre réaction…)
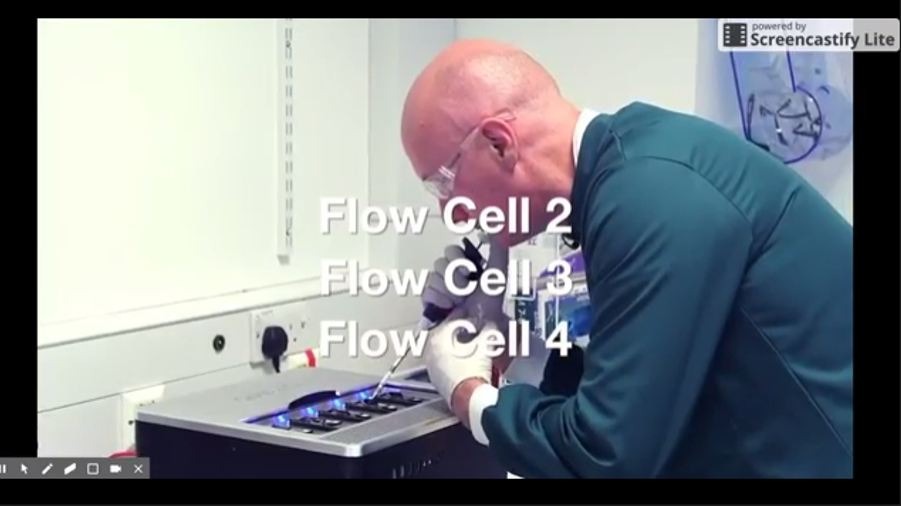
En mars, Oxford Nanopore a annoncé le lancement du GridIon X5, un séquenceur nanopore de bureau (et non plus de poche comme le MinION) pouvant permettre de travailler jusqu’à 5 flow cells à la fois et disposera d’un débit de 100 Gb par run de 48 heures. La plate-forme est livrée avec un cluster local de calcul haute performance qui permet le basecalling en temps réel ainsi que l’analyse des données. La première unité a été expédiée en début de semaine (en atteste la photo ci-dessous)

La société a récemment utilisé le GridIon pour séquencer un génome humain à 20 X, en utilisant 5 flow cells et la chimie R9.4 (en mode 1D).
Le GridION X5 permet d’exécuter simultanément ou individuellement jusqu’à cinq expériences; Les utilisateurs peuvent choisir d’utiliser tout ou partie de cette ressource à tout moment. La version actuelle de la chimie et du logiciel permet de générer jusqu’à 100 Gb de données pendant une exécution GridION X5 et le module de calcul peut analyser ces données en temps réel.
En utilisant la même technologie de base que le MinION et PromethION, le GridION X5 offre la possibilité de séquencer ADN et ARN en temps réel (dans la vidéo ci-dessous Clive Brown se transforme en Pipetman pour la promotion du GridION) :
Oxford Nanopore devait envoyer il y a peu les premiers consommables pour PromethION permettant de délivrer 50 Gb et pourraient en générer jusqu’à 120 Gb /jour dans un futur proche. En vitesse de croisière cette configuration sera capable de générer plus de données que le NovaSeq d’Illumina. Du lourd, du gros et du très petit : de quoi satisfaire les plateformes de séquençages et bientôt certainement des utilisateurs non touchés actuellement par la technologie aujourd’hui, puisque la version ci-dessous permet de préparer une librairie et de la séquencer avec quelques breloques qui tiennent dans le creux d’une main !

 Après de longues péripéties où il s’agissait de ralentir la progression d’une molécule d’ADN à travers un nanopore, après quelques investissements (de la part d’Illumina, essentiellement) il semblerait que la société, Oxford Nanopore s’apprête à vendre les deux produits dont elle fait la promotion depuis plusieurs mois. Ces deux produits n’existaient alors que dans les couloirs de l’AGBT (en 2012, parce que cette année il semble qu’Oxford Nanopore fasse profil bas à l’AGBT2013… à moins que…) et sur le site internet de la société encore un peu britannique.
Après de longues péripéties où il s’agissait de ralentir la progression d’une molécule d’ADN à travers un nanopore, après quelques investissements (de la part d’Illumina, essentiellement) il semblerait que la société, Oxford Nanopore s’apprête à vendre les deux produits dont elle fait la promotion depuis plusieurs mois. Ces deux produits n’existaient alors que dans les couloirs de l’AGBT (en 2012, parce que cette année il semble qu’Oxford Nanopore fasse profil bas à l’AGBT2013… à moins que…) et sur le site internet de la société encore un peu britannique.
Quelques médias d’outre-Manche évoquent cette fameuse technologie de séquençage en l’affublant du qualificatif de « future grande invention britannique » : lire à ce sujet la page web de « The Raconter » : Britain greatest inventions ». Cette invention est associée à la seule médecine personnalisée comme pour envisager le futur marché du séquençage haut-débit.
Les voyants semblent donc au vert pour Oxford Nanopore. Pour préparer le terrain, sort en ce début d’année, un article de Nature Methods : « disruptive nanopores« . Un titre qui fait écho à celui de Forbes (février 2012) repris dans notre image ci-dessus.
Nicole Rusk, rédactrice en chef à Nature Methods, vient avancer les principales caractéristiques du futur produit :
– des reads entre 10 et 100 kb
– des taux d’erreurs entre 1 et 4 %
– la possibilité de connaître les bases méthylées
– la possibilité de séquencer directement l’ARN
– la méthode est non destructive

La technologie de séquençage de 3ème génération par nanopores promet de révolutionner plusieurs applications à commencer par le séquençage de novo en laissant peu d’espoir à la technologie de Pacific Biosciences. Elle devrait remplacer PacBio dans les stratégies de séquençage hybride (qui consiste « à coupler » un séquenceur de 2ème génération permettant d’être très profond et d’une technologie de 3ème génération permettant de générer des reads très longs ce qui permet au final un assemblage de meilleure qualité).
Après avoir engendré de la curiosité, de l’impatience, puis déçu avec une arrivée sans cesse repoussée, il semble qu’Oxford Nanopore doive prouver de l’efficacité de sa technologie. Ainsi, la société britannique a annoncé le 8 janvier 2013, une série d’accords avec plusieurs institutions telles que l’ Université de l’Illinois, l’Université Brown, l’Université de Stanford , l’Université de Boston, de Cambridge et de Southampton. Oxford Nanopore prend son temps ou rencontre des difficultés avec son exonucléase. Malgré ses dizaines de brevets, pour conserver sa crédibilité la société a dû communiquer pour convaincre de l’efficience de sa technologie en minimisant les difficultés de développements, en alimentant les tuyaux de communication avec des séquenceurs sorti de palettes graphiques loin d’être finalisés.
Ce retard de lancement s’apparente t’il à un gage de sérieux ou est-il la preuve que le séquençage par nanopores rencontre de grosses, très grosses difficultés de développement ? Réponse en 2013.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages