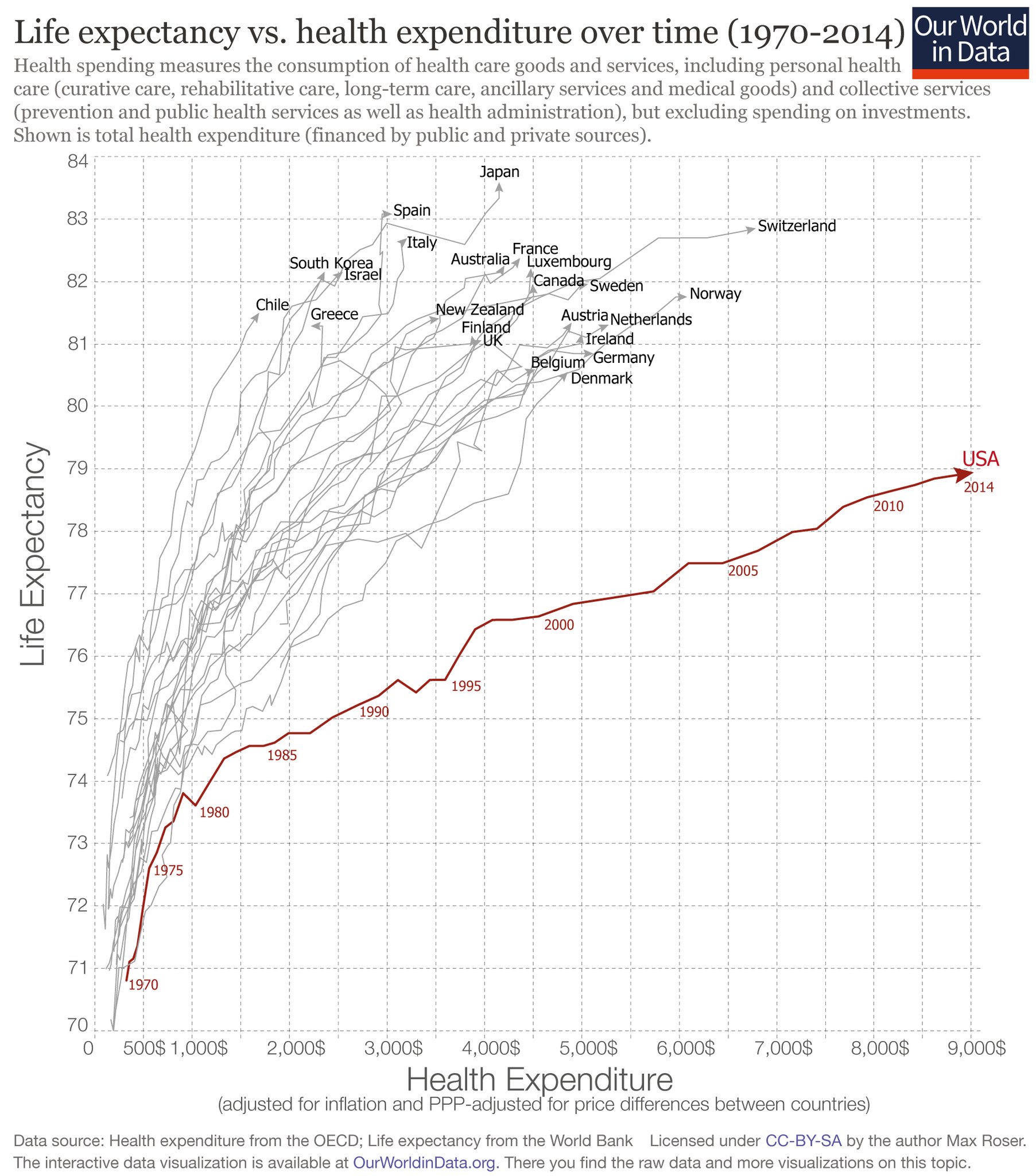
Des explications précises et instructives sur l’état actuel (au 20 février 2020) des connaissances concernant l’épidémie provoquée par ce nouveau Coronavirus (COVID-19) – Durée : 35Min
Le professeur Arnaud Fontanet revient en détail sur l’histoire de ce virus, de la découverte des premiers cas, à l’enquête sur son mode de transmission jusqu’à son séquençage extrêmement rapide.
Il donne également beaucoup d’informations sur la durée d’incubation, la contagion, les symptômes associés, les mesures sanitaires et le travail des épidémiologistes et des chercheurs pour endiguer la propagation.
Les conséquences économiques ainsi qu’un parallèle pas inintéressant avec la grippe saisonnière permettront, pour certains, de mettre en perspective cette épidémie par rapport à notre monde actuel…
Cette intervention fait partie du MOOC de l’Institut Pasteur « Virus émergents et réémergents ».
Selon une étude : La consommation (modérée) de vin rouge liée à une meilleure santé intestinale…
Une étude du King’s College de Londres tend à montrer que les personnes qui boivent du vin rouge présenteraient une plus grande diversité de microbiote intestinal (un signe de santé intestinale) que les buveurs de vin non rouge.
Dans un article publié le 28 août dans la revue Gastroenterology, une équipe de chercheurs du Department of Twin Research & Genetic Epidemiology au King’s College de Londres a étudié l’effet de la bière, du cidre, du vin rouge, du vin blanc et des spiritueux sur le microbiote intestinal et sur la santé qui en découle chez un groupe de 916 jumelles britanniques. Pourquoi des jumelles? Pour que le fond génétique soit identique, donc la différence de microbiote intestinal entres les jumelles sera en grande partie dû à l’environnement.
Ils ont constaté que le microbiote intestinal des buveurs de vin rouge était plus diversifié que celui des buveurs de vin non rouge. Ceci n’a pas été observé avec la consommation de vin blanc, de bière ou de spiritueux.
La première auteure de l’étude, Caroline Le Roy, du King’s College de Londres, a déclaré : « Bien que nous connaissions depuis longtemps les bienfaits inexpliqués du vin rouge sur la santé cardiaque, cette étude montre qu’une consommation modérée de vin rouge est associée à une plus grande diversité et à un microbiote intestinal plus sain qui explique en partie ses effets bénéfiques sur la santé. »
Le microbiome est la collection de micro-organismes dans un environnement et joue un rôle important dans la santé humaine. Un déséquilibre entre les « bons » microbes et les « mauvais » microbes dans l’intestin peut entraîner des effets néfastes sur la santé, comme un affaiblissement du système immunitaire, un gain de poids ou un taux de cholestérol élevé.
Le microbiote intestinal d’une personne ayant un nombre plus élevé d’espèces bactériennes différentes peut-être considéré comme un marqueur de la santé intestinale.
L’équipe a observé que le microbiote intestinal des consommateurs de vin rouge contenait un plus grand nombre d’espèces bactériennes différentes que celui des non-consommateurs. Ce résultat a également été observé dans trois cohortes différentes au Royaume-Uni, aux États-Unis et aux Pays-Bas. Les auteurs ont tenu compte de facteurs tels que l’âge, le poids, le régime alimentaire régulier et le statut socio-économique des participants et ont continué à voir l’association.
Les auteurs croient que la raison principale de cette association est due aux nombreux polyphénols présents dans le vin rouge. Les polyphénols sont des produits chimiques de défense naturellement présents dans de nombreux fruits et légumes. Ils ont de nombreuses propriétés bénéfiques (y compris des antioxydants) et agissent principalement comme un carburant pour les microbes présents dans notre système.
L’auteur principal, le professeur Tim Spector du King’s College de Londres, a déclaré : « Il s’agit de l’une des plus importantes études jamais réalisées sur les effets du vin rouge sur l’intestin de près de trois mille personnes dans trois pays différents et elle montre que les niveaux élevés de polyphénols dans la peau du raisin pourraient être responsables d’une grande partie des bienfaits controversés pour la santé lorsqu’ils sont utilisés avec modération. »
« Bien que nous ayons observé une association entre la consommation de vin rouge et la diversité du microbiote intestinal, boire du vin rouge rarement, comme une fois toutes les deux semaines, semble suffisant pour observer un effet. Si vous devez choisir une boisson alcoolisée aujourd’hui, c’est le vin rouge qu’il faut choisir, car il semble exercer un effet bénéfique sur vous et sur vos microbes intestinaux, ce qui peut aussi aider à réduire le poids et le risque de maladies cardiaques. Cependant, il est toujours conseillé de consommer de l’alcool avec modération, vous n’avez pas à boire du vin rouge, et vous n’avez pas à commencer à en boire si vous ne buvez pas », a ajouté le Dr Le Roy.
Effectivement, comme dans toute étude, corrélation n’est pas raison! Ainsi et même si la catégorie socio-économique est prise en compte dans l’étude, il pourrait exister d’autres facteurs, non mesurés dans cette étude, qui expliqueraient en partie la bonne santé microbienne des individus. Rappelons que ça n’est pas l’alcool qui est associé avec une meilleure santé intestinale mais d’autres composants du vin rouge (l’hypothèse étant que ce sont les polyphénols), que l’on trouvera aisément dans d’autres aliments (fruits, légumes, noix, cacao…) .
Emplacement de la publication originale :
https://www.sciencedirect.com/science/article/abs/pii/S0016508519412444
L’abus d’alcool est dangereux pour la santé, consommez avec modération
Comment réagiriez vous si l’on vous « offrait » votre génotypage complet, ouvrant la possibilité de prédire d’éventuelles maladies, réactions à certains médicaments etc. ? Certes, dans le cas de l’Estonie (contrairement à ce que propose des sociétés privées telles 23andMe) il s’agit d’une démarche s’inscrivant en santé publique pour le meilleur du bien public et non pour le pire du bien privé… Néanmoins, l’ampleur de la cohorte humaine visée n’est pas sans poser des questions.
L’Estonie a lancé un programme visant à recruter et à génotyper 100.000 nouveaux participants à la biobanque (pour une population nationale totale de 1,316 million de citoyens estoniens !) dans le cadre de son programme national de médecine personnalisée. Cette biobanque comportait déjà 50.000 génotypes de citoyens estoniens. Le gouvernement veut développer un système de soins de santé en offrant à un maximum de ses résidents un génotypage scannant le génome qui sera traduit en rapports personnalisés. Ce rapport serait intégré à la pratique médicale quotidienne par l’entremise du portail national de cybersanté.
Cette biobanque a été initiée à trois fins (Leitsalu et al., International Journal of Epidemiology, 2015) :
- promouvoir le développement de la recherche génétique ;
- recueillir des informations sur l’état de santé de la population estonienne, ainsi que des informations génétiques
- utiliser les résultats de la recherche génétique pour améliorer la santé publique.

Au sein de l’article de Leitsalu et al., les auteurs abordent les forces et faiblesses de leur entreprise :
La principale force de la Biobanque estonienne consiste au fait qu’il s’agit d’une biobanque basée sur la population avec une base de données longitudinales et prospectives. Cela signifie qu’un large éventail de groupes d’âge et de phénotypes sont représentés. Alors que les populations urbaines ont généralement tendance à être surreprésentées, ce n’est pas le cas pour la cohorte de la Biobanque estonienne. La biobanque dispose d’ADN, de plasma et de globules blancs pour chaque donneur. Cela signifie qu’il est possible d’analyser les effets directs des variants de séquence sur le métabolisme. De plus, il est possible de transformer les cellules en lignées cellulaires ou en cellules souches pluripotentes induites (iPS) et de réaliser directement des expériences de biologie moléculaire ou de génétique. Une autre force est fournie par la HGRA (the Estonian Human Genes Research Act) ainsi que par le formulaire de consentement général qui permet de participer à un large éventail de projets de recherche sans avoir à communiquer de nouveau et à demander un nouveau consentement. La HGRA et le formulaire de consentement permettent également aux donneurs de demander la divulgation de leurs données génétiques, de leurs caractéristiques héréditaires et des risques génétiques obtenus à partir de la recherche génétique menée. Cela permettrait à terme de mener des projets sur les tests du génome personnel, la perception des risques et la gestion des risques en milieu industriel.
De plus, la HGRA permet à la Biobanque d’obtenir des renseignements supplémentaires en reliant les dossiers aux registres électroniques nationaux et aux principaux hôpitaux. Tous les registres sont reliés de façon centralisée par une infrastructure technique à l’échelle nationale qui permet l’échange sécurisé de données entre les bases de données. La HGRA a également imposé des restrictions sur les activités de l’EGCUT (Estonian Genome Center of the University of Tartu) et les données collectées dans la Biobanque estonienne. La participation devait être entièrement volontaire – seules les personnes intéressées pour rejoindre la Biobanque estonienne, après en avoir entendu parler soit lors d’événements promotionnels spéciaux, soit par les médias, soit par des amis, soit au cabinet du médecin de famille ou à l’hôpital, sont recrutées. L’EGCUT n’a pas été autorisé à envoyer les lettres d’invitation à leur adresse domiciliaire. Par conséquent, la biobanque ne représente pas un échantillon aléatoire classique et pourrait être sujette à un biais de recrutement. Une proportion considérable de la population recrutée pourrait toutefois compenser ce biais. Par conséquent, bien qu’elle ne soit pas aléatoire sur le plan classique, la cohorte peut quand même être considérée comme représentative de la population. Bien que le recrutement était ouvert à tous, il y a une disproportion d’Estoniens ethniques et de Russes ethniques dans la biobanque, les Estoniens étant surreprésentés (81% dans la biobanque contre 70% dans la population générale) et les Russes sous-représentés (16% dans la biobanque contre 25% dans la population générale). Une autre faiblesse est la profondeur limitée de certains sous-questionnaires. Par exemple, un questionnaire relativement bref sur la fréquence des aliments a été administré sans information détaillée sur l’apport en énergie ou en nutriments ; les mesures des traits glycémiques à jeun, comme le niveau d’insuline, ne sont disponibles que pour un nombre limité d’échantillons. La profondeur limitée des données recueillies peut parfois limiter le nombre de projets dans lesquels les données peuvent être utilisées. Toutefois, des questionnaires plus complets auraient exigé des durées d’entrevue encore plus longues et auraient coûté beaucoup plus cher, ce qui aurait pu entraîner une réduction de la taille de la cohorte.
Le pays dispose de nombreuses solutions numériques sécurisées incorporées dans les fonctions gouvernementales qui relient les diverses bases de données du pays par des voies cryptées de bout en bout. Un site Web a été créé dans le cadre du projet, afin que les Estoniens puissent se porter volontaires et donner leur consentement à être génotypés. La génération des données est assurée par l’institut de génomique de l’Université de Tartu (aujourd’hui les 50.000 premiers génotypages ont été réalisés et analysés, les 100.000 pousseront à une population estonienne à 10 % génotypée)
Les efforts internationaux ont permis d’identifier des milliers d’associations entre les variants génétiques et les maladies, ou traits génétiques, et de créer des cartes des variations uniques au sein des populations.
« Aujourd’hui, nous avons suffisamment de connaissances sur le risque génétique des maladies complexes et la variabilité interindividuelle des effets des médicaments pour commencer à utiliser systématiquement ces informations dans les soins de santé au quotidien « , a déclaré Jevgeni Ossinovski, ministre de la Santé et du Travail. « En coopération avec l’Institut national pour le développement de la santé et l’Université de Tartu, nous allons permettre à 100.000 autres personnes de rejoindre la biobanque estonienne, afin de stimuler le développement de la médecine personnalisée en Estonie et de contribuer ainsi à l’avancement des soins de santé préventifs. «
Le gouvernement estonien a alloué 5 millions d’euros au programme au cours de l’année 2018. Le projet sera coordonné par l’Institut national pour le développement de la santé, dont la tâche est d’élaborer et de mettre en œuvre des procédures et des principes pour la mise en œuvre efficace de la recherche scientifique dans la pratique médicale.
Andres Metspalu, directeur du Centre estonien du génome à l’Université de Tartu, se félicite de l’initiative du ministère des Affaires sociales d’augmenter le nombre de participants à la biobanque. « Nous sommes heureux qu’avec le soutien de ce projet, les résultats des travaux à long terme du centre de génomique seront transférés en médecine pratique et donneront un nouvel élan à nos recherches futures. L’Université contribuera également à la création d’un système de rétroaction pour les participants de la biobanque, et à la formation des professionnels de la santé pour qu’ils puissent donner aux patients une rétroaction fondée sur l’information génétique« .
Le projet sera mis en œuvre sur la base de la loi estonienne sur la recherche sur les gènes humains et du même formulaire de consentement général qui a été utilisé pour les 50.000 premiers participants. Le prélèvement officiel d’échantillons a débuté le 2 avril 2018. A voir si l’expérience relativement pionnière ,à cette échelle, menée en Estonie, fera école ou tâche d’huile dans d’autres pays de l’Union Européenne !
 Un slogan, une baseline : « nous avons cartographié le monde, maintenant cartographions la santé humaine » annonce la volonté de Google de persévérer dans le domaine de la santé humaine. C’est ainsi qu’Alphabet le conglomérat appartenant à et détenant Google tout à la fois, propose de constituer une cohorte humaine phénotypiquement caractérisée le plus finement possible : Verily (anciennement Google Life Sciences) une filiale d’Alphabet spécialisée dans la santé, a annoncé mercredi 19 avril qu’elle souhaitait recruter 10 000 volontaires pour son projet Baseline, annoncé en 2014 et déjà testé sur une centaine de volontaires. S’adjoignant des chercheurs du monde académique avec la participation de l’université de Duke (Caroline du Nord) et de l’université Stanford, Google vise à collecter des données de santé très précises sur ces personnes pendant plusieurs années. Assurément le nombre de personnes visées est pour l’instant moindre que la célèbre cohorte Nurses’ Health Study débutée en 1976 et dénombrant 280.000 participants, l’innovation consiste en la qualité des données collectées par l’intermédiaire de capteurs connectés. Les études épidémiologiques faisant intervenir des cohortes ne sont certes pas nouvelles, elles sont un outil formidable, grandes pourvoyeuses de résultats scientifiques valorisables (la célèbre cohorte de Framingham enregistre plusieurs centaines de publications). Dans le cas précis du projet Baseline, ce qui est nouveau est la promotion de ce type d’approche par et pour aussi un peu, une entité privée. Google, enfin Alphabet, peu importe finalement, est un conglomérat qui possède notamment la société de biotechnologie Calico, et qui a des liens capitalistiques avec 23andMe et possède donc encore Verily dont certains projets consistent en :
Un slogan, une baseline : « nous avons cartographié le monde, maintenant cartographions la santé humaine » annonce la volonté de Google de persévérer dans le domaine de la santé humaine. C’est ainsi qu’Alphabet le conglomérat appartenant à et détenant Google tout à la fois, propose de constituer une cohorte humaine phénotypiquement caractérisée le plus finement possible : Verily (anciennement Google Life Sciences) une filiale d’Alphabet spécialisée dans la santé, a annoncé mercredi 19 avril qu’elle souhaitait recruter 10 000 volontaires pour son projet Baseline, annoncé en 2014 et déjà testé sur une centaine de volontaires. S’adjoignant des chercheurs du monde académique avec la participation de l’université de Duke (Caroline du Nord) et de l’université Stanford, Google vise à collecter des données de santé très précises sur ces personnes pendant plusieurs années. Assurément le nombre de personnes visées est pour l’instant moindre que la célèbre cohorte Nurses’ Health Study débutée en 1976 et dénombrant 280.000 participants, l’innovation consiste en la qualité des données collectées par l’intermédiaire de capteurs connectés. Les études épidémiologiques faisant intervenir des cohortes ne sont certes pas nouvelles, elles sont un outil formidable, grandes pourvoyeuses de résultats scientifiques valorisables (la célèbre cohorte de Framingham enregistre plusieurs centaines de publications). Dans le cas précis du projet Baseline, ce qui est nouveau est la promotion de ce type d’approche par et pour aussi un peu, une entité privée. Google, enfin Alphabet, peu importe finalement, est un conglomérat qui possède notamment la société de biotechnologie Calico, et qui a des liens capitalistiques avec 23andMe et possède donc encore Verily dont certains projets consistent en :
- des lentilles de contact permettant de contrôler le niveau de glucose chez les personnes diabétiques
- des cuillères pour les personnes ayant des tremblements, par exemple atteintes de la maladie de Parkinson (projet Liftware)
- une plateforme permettant la détection de maladie par l’intermédiaire de nanoparticules
- un bracelet connecté permettant de suivre des paramètres liés à la santé
Certains de ces objets connectés auront une exposition majeure du fait de leur positionnement central dans la cohorte de Baseline. Ainsi une montre, au design contestable, complétera la panoplie de capteurs associée au projet. Le fameux capteur d’activités nocturnes qui se faufile jusque sous la couette peut laisser perplexe… Ces objets connectés, pourvoyeurs de données, de beaucoup de données, nécessiteront les méthodologies que Google s’attache à développer pour traiter de façon automatisée et optimale la manne visée par la cohorte Baseline ! En outre, ces objets trouveront un écho, à n’en pas douter, auprès de futurs consommateurs bien au-delà des personnes constituant la cohorte initiale. En effet, les modèles établis sur les personnes, population référente constitutive de la cohorte (pour lesquelles sont à disposition l’intégralité des données phénotypiques) pourront être appliqués à de futures personnes dotées de la batterie de capteurs mais n’ayant pour autant pas été caractérisées finement. La cohorte servirait à établir un modèle, modèle qui serait appliqué à de futurs utilisateurs des solutions connectées proposées par Google. Ainsi des prédictions de l’état de santé et pourquoi pas la mise en place d’un système d’alerte… pouvant aller jusqu’à signifier la nécessité de consulter pourraient trouver une application commerciale.
L’objectif qu’Alphabet revendique est de cartographier la santé humaine, cet objectif passe par la création d’une immense base de données avec la santé pour finalité, permettant, grâce aux nouvelles technologies, d’«explorer la santé en profondeur». Après anonymisation comme garantie de sécurité des données, cette base de données est destinée à être transmise à des chercheurs. Pour recruter ses volontaires et les inciter à rejoindre le projet (la stratégie de recrutement peut laisser entrevoir d’ailleurs quelques biais de recrutement), Verily assure qu’il s’agit d’une manière « de participer à la création de cette carte de la santé humaine, et de laisser durablement une trace», en contribuant à son échelle à la recherche médicale. Ces futurs participants pourront partager leurs données avec leur médecin. Verily cherche des personnes nord-américaines, en bonne santé qui seront suivies et invitées à subir, pendant deux jours, chaque année, une batterie de tests médicaux. Ces bilans médicaux accompagnés des données des bio-capteurs dont ils seront dotés seront agrégés sous forme de « Big Data », matière première dont Google et ses méthodologies sont devenus experts…
Plusieurs questions peuvent être soulevées :
- est ce que Google, entreprise commerciale, finira par monétiser ces données comme matière première à une recherche scientifique ?
- est ce que la stratégie de Google ne serait pas multiple : (i) faire la promotion de ces outils d’analyses sur une matière première (dont Google serait propriétaire) tout en (ii) faisant la promotion de ces objets connectés, surfant sur un marché de la e-santé, marché en pleine expansion, tout en (iii) rentabilisant son investissement pour « partager » ses données recrutées moyennant des contreparties soumises au secret ?
Par le passé, Google Health est – était- un service Internet d’archivage de dossiers médicaux pour les internautes américains, mis en place par Google en mars 2008. L’avantage reste celui de laisser le pouvoir aux pharmaciens américains de mettre à jour automatiquement les traitements ou encore de trouver un spécialiste pour traiter des maladies adaptées. Google fermera ce service le 1er janvier 2012, faute d’un nombre insuffisant d’utilisateurs. La volonté de Google de développer une offre trouvant des applications en santé, médecine de précision, auxiliaire personnel de santé n’est donc pas nouvelle. Aujourd’hui, dans un contexte américain où les fonds publics alloués à la recherche médicale se tarissent (lire l’article de Nature sur la coupe budgétaire sans précédents de Donald Trump), le temps des partenariats public/privé est peut être venu. Dans ce contexte, même si la démarche de Google est nappée de marketing, des projets tels que Baseline pourraient être favorablement accueillis par la communauté scientifique.
Bien évidemment cette infographie vue d’ici vous parait assez peu lisible. Après avoir cliqué dessus, vous pourrez vous apercevoir qu’elle permet de s’y retrouver dans la jungle de ces molécules aux noms tous plus baroques… 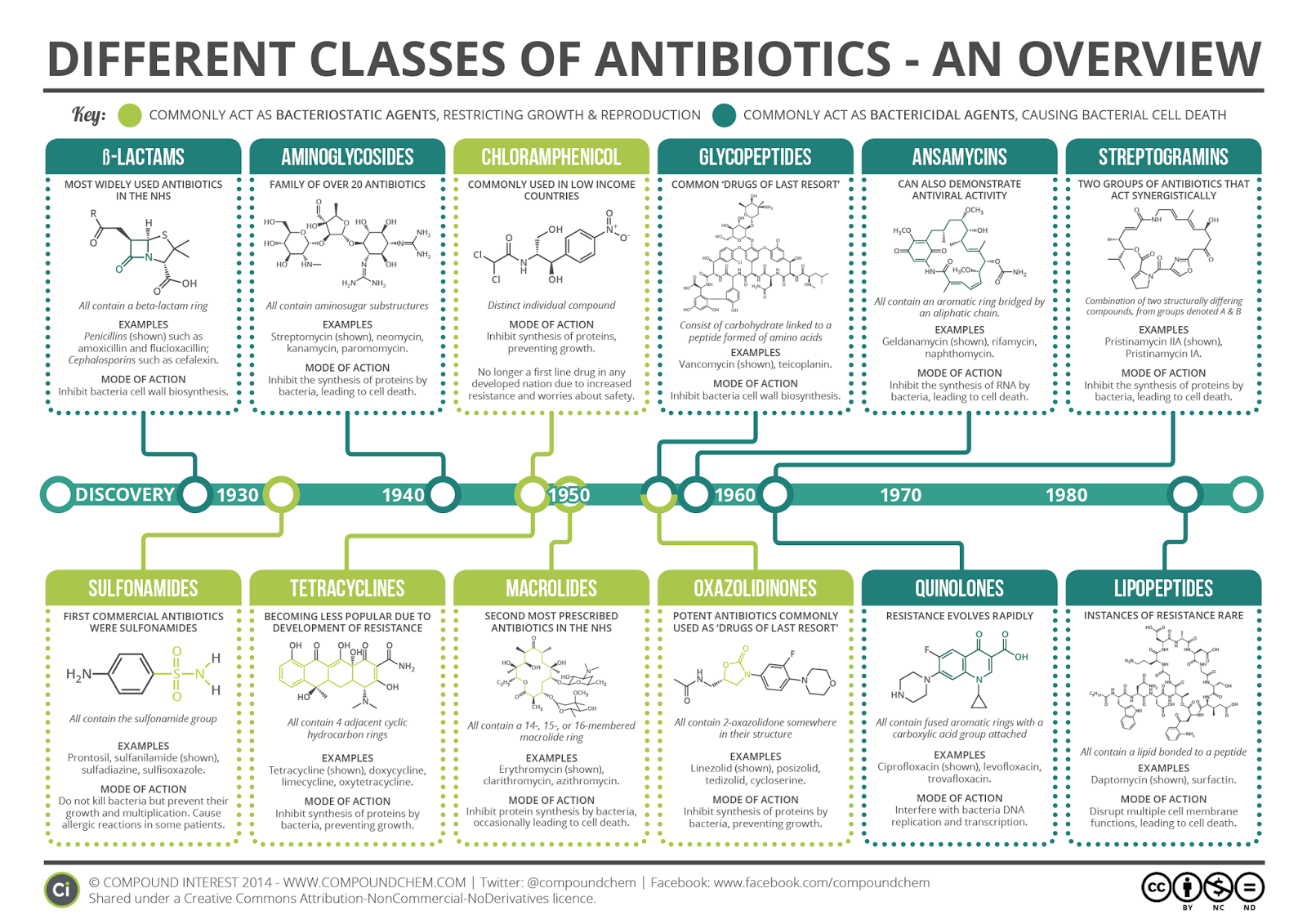
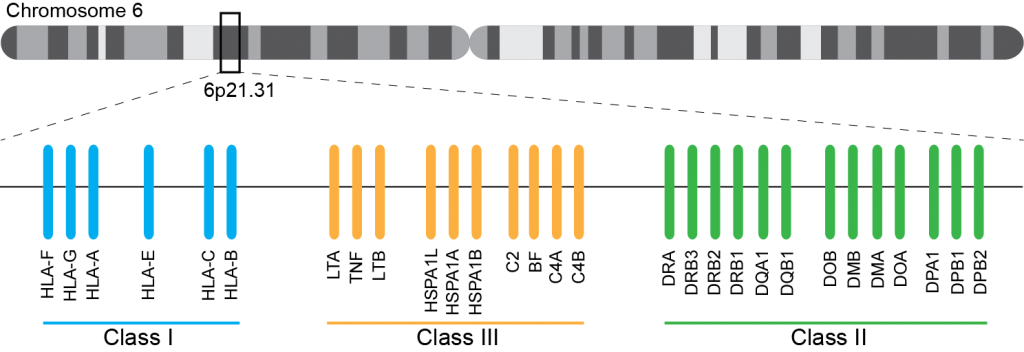
En cancérologie, l’allogreffe de moelle osseuse s’inscrit dans un parcours thérapeutique notamment comme traitement de consolidation après une chimiothérapie. Aussitôt, les notions de rejet ou d’acceptation du greffon apparaissent et il devient indispensable que les systèmes HLA (Human Leucocyt Antigens, découvert en 1950) du donneur et du receveur soient les plus proches possibles.
Ce système immunogène, situé sur le bras court du chromosome 6 chez l’homme, est caractérisé par son polygénisme et son polymorphisme qui sont à l’origine d’une grande variabilité interindividuelle et en fait le déterminant principal du résultat de greffe. L’ensemble des gènes HLA sont subdivisés en trois régions du chromosome 6 qui contiennent chacune de nombreux gènes avec ou sans fonction immunologique. On distingue ainsi la région CMH de classe I, de classe II, et de classe III.
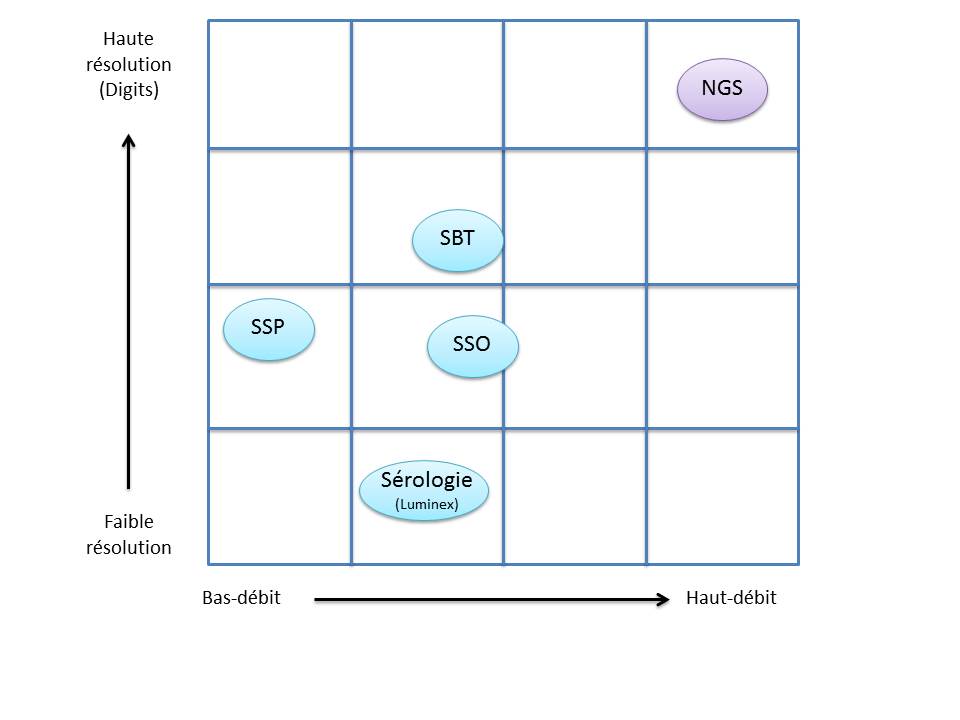
A ce jour, un rendu de typage est ciblé sur une portion génomique restreinte codant pour le HLA. Il s’agit de l’exon 2 et 3 des loci HLA-A, HLA-B et HLA-C (région I), l’exon 2 et 3 des loci HLA-DQ (DQ-A et DQ-B) et l’exon 2 pour HLA-DR (DRA et DRB1), où repose prés de 70% du polymorphisme. La région III ne renfermant pas de gènes intervenant dans la présentation antigénique.
C’est ainsi que différentes approches de typages ont été développées et l’avènement de la technique de PCR au milieu des années 1980 a pallié aux limites de résolution de la sérologie employée jusqu’alors. D’un typage rendu au niveau générique (2 Digits), la « PCR-SSO » (Sequence Specific Oligonucleotide) et la « PCR-SSP » (Sequence Specific Primer) développées dans les années 1990 ont permis d’accéder à un résultat allélique (4 Digits). Cette avancée technologique s’est poursuivie la décennie suivante avec la « PCR-SBT » (Sequence Based Typing) ou séquençage « Sanger » puis plus récemment avec la PCR en temps réel (ex: linkage). Toutes ces techniques de biologie moléculaire ont permis de mettre en avant le polymorphisme et la grande diversité génétique du HLA. Chaque année, de nombreux allèles sont découverts, alimentant continuellement la banque de données de référence IMGT.
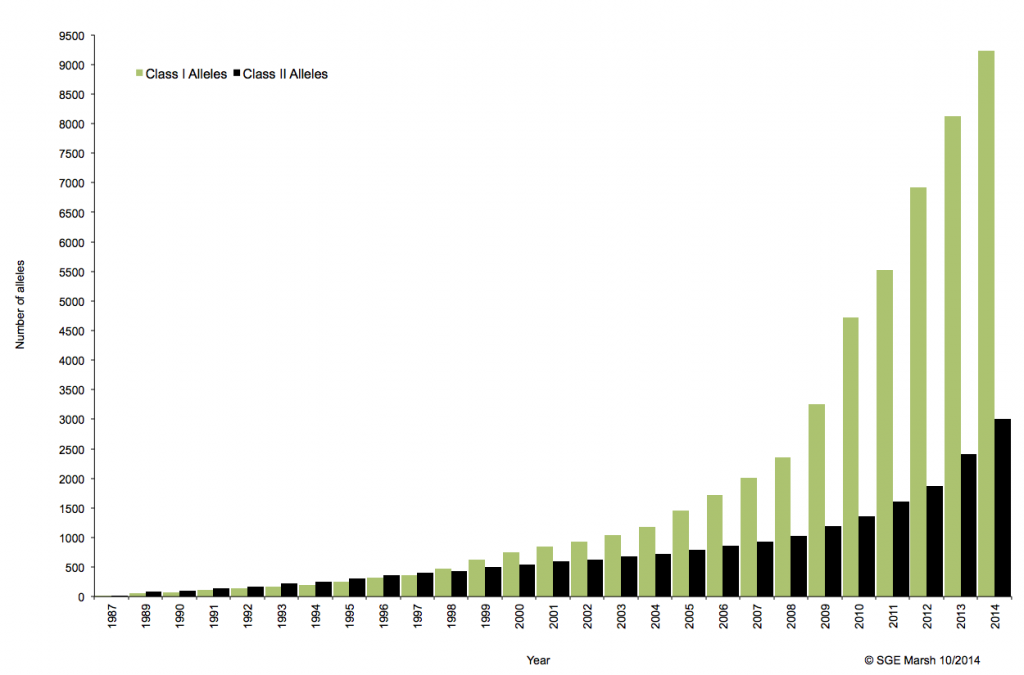
Associé à cette augmentation constante du nombre d’allèles typés, le nombre d’ambigüités croît et met progressivement en difficulté les technologies conventionnelles qui atteignent leurs limites. De plus, le pourcentage de réussite des allogreffes n’atteint environ que 50%. L’exploitation du reste de l’information génomique permettrait potentiellement d’améliorer cette performance par un typage plus affiné.
Le recours au séquençage nouvelle génération apparait donc inévitable. En plus de gérer les ambiguïtés par un séquençage allélique, le NGS permet de traiter simultanément de grandes quantités d’échantillons réduisant ainsi le coût unitaire, d’accéder à un niveau de résolution supérieur (4,6 ou 8 digits) tout en ayant la capacité de cibler des loci entier (Long Range PCR).
Ainsi, plusieurs stratégies existent avec leur solution technique adaptée à la préparation de la matrice d’ADN à séquencer:
– L’amplification ciblée des régions d’intérêt par PCR de fusion. Cette approche permets un gain de temps et une réduction des coûts en s’affranchissant des étapes de fragmentation, ligation, et autres purifications… .Par ailleurs elle n’est pas la mieux adapté dans le cas d’une couverture de séquençage de l’ensemble de la région génomique HLA.
– L’amplification par « Long Range PCR » permet une couverture complète des différents loci étudiés. Les fragments de plusieurs Kb subissent alors une fragmentation, une ligation des adaptateurs et indexation. Cette approche permet d’accéder à davantage d’informations (régions exoniques et introniques).
– La capture de séquences par hybridation. Même si cette solution est bien caractérisée elle n’est pas si efficace en terme de capture avec une disparité selon la taille des fragments.
– Le séquençage de génome entier ou d’exome. Cette approche est la moins biaisée et couvre tous les gènes du système HLA. Paradoxalement, l’analyse nécessite beaucoup trop de ressources pour une utilisation en routine et ne permets pas de traiter autant d’échantillons simultanément.
Plusieurs sociétés commerciales proposent des solutions clé en main depuis la préparation des échantillons (avec l’option « Long Range PCR » qui semble la plus plébiscité) jusqu’à l’analyse de résultats via leur logiciel dédié. Certaines sont en cours de validation de méthode pendant que d’autres tentent d’inonder le marché. Parmi elles, Gendx, Omixon, Illumina, One lambda, Life technologies, Immucor, etc…
L’exploitation des capacités et caractéristiques des solutions de séquençage à haut-débit permettrait d’affiner considérablement le typage HLA. Ainsi le décryptage de l’ensemble des régions codantes et non-codantes du génome d’intérêt représente un enjeu important dans la réussite des greffes. Par ailleurs, cette approche nécessitera une mise à jour considérable des banques de données (IMGT) avec une validation de nombreux nouveaux allèles.
 La toute puissante FDA (Food and Drug Administration) a calmé les ardeurs la société 23andMe en remettant en question la diffusion par la société californienne de tests génomiques personnels. (Étonnamment ?), la FDA ne semble pas réellement heurtée par le principe même d’une société de droit privé glanant des informations génomiques pour réaliser un commerce des plus lucratifs, motus concernant le devenir de ce type de données. En réalité, l’administration américaine, soucieuse du service rendu au consommateur, émet des doutes quant à la pertinence, quant à l’exploitation des résultats, après génotypages, des individus consentants, ainsi que sur la façon dont le tout est présenté au consommateur. Ainsi, 23andMe est mise en cause au niveau de la qualité de son service. Voici son seul « crime » aux yeux de la FDA!
La toute puissante FDA (Food and Drug Administration) a calmé les ardeurs la société 23andMe en remettant en question la diffusion par la société californienne de tests génomiques personnels. (Étonnamment ?), la FDA ne semble pas réellement heurtée par le principe même d’une société de droit privé glanant des informations génomiques pour réaliser un commerce des plus lucratifs, motus concernant le devenir de ce type de données. En réalité, l’administration américaine, soucieuse du service rendu au consommateur, émet des doutes quant à la pertinence, quant à l’exploitation des résultats, après génotypages, des individus consentants, ainsi que sur la façon dont le tout est présenté au consommateur. Ainsi, 23andMe est mise en cause au niveau de la qualité de son service. Voici son seul « crime » aux yeux de la FDA!
L’administration américaine lui reproche :
– malgré les sollicitations récurrentes de la part de la FDA, 23andMe n’a pas fait valider par l’administration ses dispositifs diagnostiques in vitro. Elle n’a cessé d’allonger la liste (stratégie commerciale du restaurant chinois) des maladies et caractères que leur système était susceptible de diagnostiquer -avant « sa mise en demeure », 23andMe proposait un menu comportant le « diagnostic » de 254 prédispositions à des « maladies » ou aptitudes diverses.
– au sujet des maladies multi-factorielles, telles que le cancer de l’ovaire ou du sein, les résultats rendus aux clients sont très probablement constitués d’un grand nombre de faux positifs et de faux négatifs.
la FDA souhaite que l’intégralité des assertions de 23andMe soit validée pour que la société continue de vendre son kit phare : health-related genetic tests.
La société qui comptait régner en maître sur le marché de la génomique récréative, a peut-être fini de jouer les apprentis sorciers (franchement peu probable !). En effet, si l’intérêt de certains de ces tests génomiques, en soi, n’est pas à remettre en question, leur encadrement législatif est à souhaiter. En outre, avant de valider des kits « diagnostiques », il serait appréciable de lever le quiproquo : les kits proposés par 23andMe sont ils des dispositifs de génomique récréative ou des dispositifs médicaux ? La libéralisation incontrôlée de ce type de marchés pourrait avoir des conséquences dramatiques… Si des prédispositions peuvent être inscrites dans notre code génétique, il n’en demeure pas moins qu’une large majorité de maladies peut être qualifiée de multifactorielle.
Se sentir à l’abri d’une maladie parce que 23andMe vous rend un résultat dans ce sens ou dans le cas contraire, sentir l’ombre de l’épée de Damoclès, peut avoir, pour le consommateur de ces tests, des conséquences graves.
La lettre d’avertissement du 22 novembre 2013, de la FDA adressée à Anne Wojciki, montre l’étendue de l’ambiguïté : LETTRE d’AVERTISSEMENT ainsi que la réponse de 23andMe.
Anne Wojcicki a déclaré que « 23andMe travaille activement avec la FDA pour s’assurer que la firme fournit des informations de haute qualité afin que les consommateurs puissent avoir confiance ».
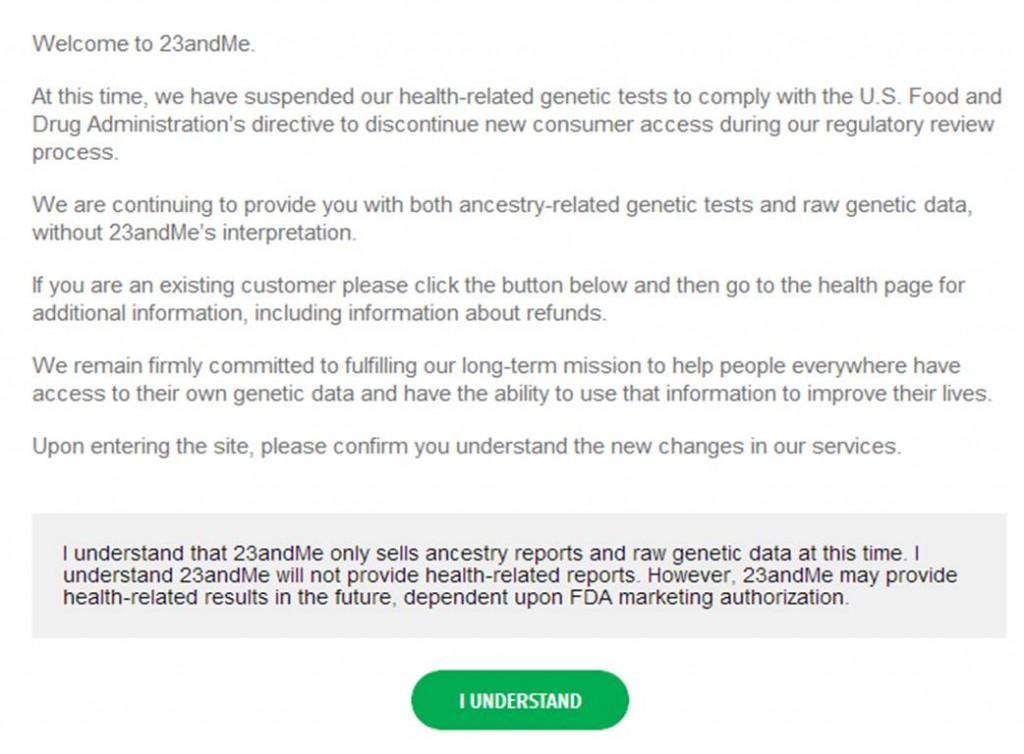
Ci-dessous, voici la nouvelle page qui vous accueille sur le site de 23andMe qui peut continuer à vendre ses tests phylogénétiques.
Comme le disait Stephen Hawking :
« J’ai remarqué que même les gens qui affirment que tout est prédestiné et que nous ne pouvons rien y changer regardent avant de traverser la rue. » (Trous noirs et bébés univers, Éditions Odile Jacob, 1994)
 Le séquençage du génome humain pour 30$, c’est la promesse faite par David Weitz, co-fondateur de GnuBio au cours de l’année 2010. Trois ans plus tard, la start-up vient de lancer en béta-test son nouveau procédé de séquençage à haut débit. Il s’agit du premier système entièrement intégré (amplification des cibles, enrichissement, séquençage et analyse) qui propose pour le moment une application de target-sequencing destinée aux cliniciens et dédiée au diagnostic moléculaire.
Le séquençage du génome humain pour 30$, c’est la promesse faite par David Weitz, co-fondateur de GnuBio au cours de l’année 2010. Trois ans plus tard, la start-up vient de lancer en béta-test son nouveau procédé de séquençage à haut débit. Il s’agit du premier système entièrement intégré (amplification des cibles, enrichissement, séquençage et analyse) qui propose pour le moment une application de target-sequencing destinée aux cliniciens et dédiée au diagnostic moléculaire.
En 2010, David Weitz et son équipe de l’Université d’Harvard ambitionnent de développer une nouvelle technologie de séquençage à haut-débit, alliant les technologies de biologie moléculaire aux procédés de microfluidique développés quelques années plus tôt (2004) au sein de la société RainDance technologies.
Cette nouvelle approche repose sur la capacité à générer des gouttes de l’ordre du picolitre et pouvant être déplacée sur une puce microfluidique. Ces gouttes peuvent renfermer soit un couple d’amorces, des adaptateurs, ou tout autre type de réactifs nécessaires aux étapes de préparation de librairie et de séquençage (séquençage par hybridation-ligation, type SOLiD avec une fidélité de 99.99%). Dès lors, leurs quantités utilisées au sein de ces picogouttes sont considérablement revues à la baisse, ce qui constitue le point clé à une réduction des coûts de séquençage et donc la perspective d’un séquençage de génome humain à 30$, selon David Weitz.
Les projets de GnuBio sont désormais d’élargir le champs d’applications de leur séquenceur à l’analyse transcriptomique (RNA-seq), l’étude de la méthylation (ChiP-seq) ou encore le séquençage de génome entier. La société ambitionne une commercialisation de leur équipement au cours de l’année 2014.
A suivre…
Bertrand Jordan a eu son doctorat en physique nucléaire à 1965 et a très bien fait de se convertir à la biologie moléculaire. Entre autres choses, il a réalisé en 1982 l’isolement du premier gène d’histocompatibilité humain, et en 2000, il a fondé la Génopole de Marseille. Bertrand Jordan est membre de l’Organisation européenne de biologie moléculaire (EMBO) ainsi que de l’organisation internationale HUGO (Human Genome Organisation).
Mais surtout, et c’est ce qui nous intéresse ici, l’auteur de plus de 150 publications scientifiques fait œuvre de vulgarisation et offre depuis plus de dix ans, des chroniques génomiques publiées au sein de la revue Médecine / Sciences. Ces chroniques sont accessibles gratuitement (vous pouvez y accéder directement sur le site de www.medecinesciences.org ou en cliquant sur l’image ci dessus en haut à gauche).
On peut vous conseiller de lire le point de vue de Bertrand Jordan sur les séquenceurs haut-débit dans sa chronique de mars 2010 intitulée : le boom des séquenceurs nouvelle génération, sans oublier le point de vue partagé (parfois longtemps après lui) par un nombre croissant de chercheurs sur les GWAS, dans sa chronique de mai 2009 : le déclin de l’empire des GWAS. C’est là tout l’intérêt de ces chroniques qui, bénéficiant d’un propos clair, simple, construit, argumenté et rare, permettent de prendre un peu de recul sur les applications d’une science qui n’ont pas toujours tenu leurs promesses. Ces chroniques peuvent être l’occasion de faire tomber le masque, de dégonfler l’air de ces vessies qui se prennent parfois pour des lanternes.
Bertrand Jordan, chroniqueur mais aussi auteur de quelques livres aux titres évocateurs, parfois mémorablement provocateurs :
Voyage autour du Génome : le tour du monde en 80 labos. Editions Inserm/John Libbey, Paris, 1993
Voyage au pays des gènes. Editions. Les Belles Lettres/ Inserm, Paris, 1995
Génétique et génome, la fin de l’innocence. Editions Flammarion, Paris, 1996
Les imposteurs de la génétique. Editions du Seuil, Paris, 2000 (Prix Roberval Grand Public 2000)
Le chant d’amour des concombres de mer. Editions du Seuil, Paris, 2002
Les marchands de clones. Editions du Seuil, Paris, 2003
Chroniques d’une séquence annoncée. Editions EDK, Paris, 2003
Le clonage, fantasmes et réalité. Essentiel Milan, Editions Milan, 2004
Thérapie génique : espoir ou illusion ? Editions Odile Jacob, Paris, 2007
L’humanité au pluriel : la génétique et les questions de race. Le Seuil, collection Sciences ouvertes, Paris, 2008
Autisme, le gène introuvable : Le Seuil, Paris, 2012
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages