Comment réagiriez vous si l’on vous « offrait » votre génotypage complet, ouvrant la possibilité de prédire d’éventuelles maladies, réactions à certains médicaments etc. ? Certes, dans le cas de l’Estonie (contrairement à ce que propose des sociétés privées telles 23andMe) il s’agit d’une démarche s’inscrivant en santé publique pour le meilleur du bien public et non pour le pire du bien privé… Néanmoins, l’ampleur de la cohorte humaine visée n’est pas sans poser des questions.
L’Estonie a lancé un programme visant à recruter et à génotyper 100.000 nouveaux participants à la biobanque (pour une population nationale totale de 1,316 million de citoyens estoniens !) dans le cadre de son programme national de médecine personnalisée. Cette biobanque comportait déjà 50.000 génotypes de citoyens estoniens. Le gouvernement veut développer un système de soins de santé en offrant à un maximum de ses résidents un génotypage scannant le génome qui sera traduit en rapports personnalisés. Ce rapport serait intégré à la pratique médicale quotidienne par l’entremise du portail national de cybersanté.
Cette biobanque a été initiée à trois fins (Leitsalu et al., International Journal of Epidemiology, 2015) :
- promouvoir le développement de la recherche génétique ;
- recueillir des informations sur l’état de santé de la population estonienne, ainsi que des informations génétiques
- utiliser les résultats de la recherche génétique pour améliorer la santé publique.

Au sein de l’article de Leitsalu et al., les auteurs abordent les forces et faiblesses de leur entreprise :
La principale force de la Biobanque estonienne consiste au fait qu’il s’agit d’une biobanque basée sur la population avec une base de données longitudinales et prospectives. Cela signifie qu’un large éventail de groupes d’âge et de phénotypes sont représentés. Alors que les populations urbaines ont généralement tendance à être surreprésentées, ce n’est pas le cas pour la cohorte de la Biobanque estonienne. La biobanque dispose d’ADN, de plasma et de globules blancs pour chaque donneur. Cela signifie qu’il est possible d’analyser les effets directs des variants de séquence sur le métabolisme. De plus, il est possible de transformer les cellules en lignées cellulaires ou en cellules souches pluripotentes induites (iPS) et de réaliser directement des expériences de biologie moléculaire ou de génétique. Une autre force est fournie par la HGRA (the Estonian Human Genes Research Act) ainsi que par le formulaire de consentement général qui permet de participer à un large éventail de projets de recherche sans avoir à communiquer de nouveau et à demander un nouveau consentement. La HGRA et le formulaire de consentement permettent également aux donneurs de demander la divulgation de leurs données génétiques, de leurs caractéristiques héréditaires et des risques génétiques obtenus à partir de la recherche génétique menée. Cela permettrait à terme de mener des projets sur les tests du génome personnel, la perception des risques et la gestion des risques en milieu industriel.
De plus, la HGRA permet à la Biobanque d’obtenir des renseignements supplémentaires en reliant les dossiers aux registres électroniques nationaux et aux principaux hôpitaux. Tous les registres sont reliés de façon centralisée par une infrastructure technique à l’échelle nationale qui permet l’échange sécurisé de données entre les bases de données. La HGRA a également imposé des restrictions sur les activités de l’EGCUT (Estonian Genome Center of the University of Tartu) et les données collectées dans la Biobanque estonienne. La participation devait être entièrement volontaire – seules les personnes intéressées pour rejoindre la Biobanque estonienne, après en avoir entendu parler soit lors d’événements promotionnels spéciaux, soit par les médias, soit par des amis, soit au cabinet du médecin de famille ou à l’hôpital, sont recrutées. L’EGCUT n’a pas été autorisé à envoyer les lettres d’invitation à leur adresse domiciliaire. Par conséquent, la biobanque ne représente pas un échantillon aléatoire classique et pourrait être sujette à un biais de recrutement. Une proportion considérable de la population recrutée pourrait toutefois compenser ce biais. Par conséquent, bien qu’elle ne soit pas aléatoire sur le plan classique, la cohorte peut quand même être considérée comme représentative de la population. Bien que le recrutement était ouvert à tous, il y a une disproportion d’Estoniens ethniques et de Russes ethniques dans la biobanque, les Estoniens étant surreprésentés (81% dans la biobanque contre 70% dans la population générale) et les Russes sous-représentés (16% dans la biobanque contre 25% dans la population générale). Une autre faiblesse est la profondeur limitée de certains sous-questionnaires. Par exemple, un questionnaire relativement bref sur la fréquence des aliments a été administré sans information détaillée sur l’apport en énergie ou en nutriments ; les mesures des traits glycémiques à jeun, comme le niveau d’insuline, ne sont disponibles que pour un nombre limité d’échantillons. La profondeur limitée des données recueillies peut parfois limiter le nombre de projets dans lesquels les données peuvent être utilisées. Toutefois, des questionnaires plus complets auraient exigé des durées d’entrevue encore plus longues et auraient coûté beaucoup plus cher, ce qui aurait pu entraîner une réduction de la taille de la cohorte.
Le pays dispose de nombreuses solutions numériques sécurisées incorporées dans les fonctions gouvernementales qui relient les diverses bases de données du pays par des voies cryptées de bout en bout. Un site Web a été créé dans le cadre du projet, afin que les Estoniens puissent se porter volontaires et donner leur consentement à être génotypés. La génération des données est assurée par l’institut de génomique de l’Université de Tartu (aujourd’hui les 50.000 premiers génotypages ont été réalisés et analysés, les 100.000 pousseront à une population estonienne à 10 % génotypée)
Les efforts internationaux ont permis d’identifier des milliers d’associations entre les variants génétiques et les maladies, ou traits génétiques, et de créer des cartes des variations uniques au sein des populations.
« Aujourd’hui, nous avons suffisamment de connaissances sur le risque génétique des maladies complexes et la variabilité interindividuelle des effets des médicaments pour commencer à utiliser systématiquement ces informations dans les soins de santé au quotidien « , a déclaré Jevgeni Ossinovski, ministre de la Santé et du Travail. « En coopération avec l’Institut national pour le développement de la santé et l’Université de Tartu, nous allons permettre à 100.000 autres personnes de rejoindre la biobanque estonienne, afin de stimuler le développement de la médecine personnalisée en Estonie et de contribuer ainsi à l’avancement des soins de santé préventifs. «
Le gouvernement estonien a alloué 5 millions d’euros au programme au cours de l’année 2018. Le projet sera coordonné par l’Institut national pour le développement de la santé, dont la tâche est d’élaborer et de mettre en œuvre des procédures et des principes pour la mise en œuvre efficace de la recherche scientifique dans la pratique médicale.
Andres Metspalu, directeur du Centre estonien du génome à l’Université de Tartu, se félicite de l’initiative du ministère des Affaires sociales d’augmenter le nombre de participants à la biobanque. « Nous sommes heureux qu’avec le soutien de ce projet, les résultats des travaux à long terme du centre de génomique seront transférés en médecine pratique et donneront un nouvel élan à nos recherches futures. L’Université contribuera également à la création d’un système de rétroaction pour les participants de la biobanque, et à la formation des professionnels de la santé pour qu’ils puissent donner aux patients une rétroaction fondée sur l’information génétique« .
Le projet sera mis en œuvre sur la base de la loi estonienne sur la recherche sur les gènes humains et du même formulaire de consentement général qui a été utilisé pour les 50.000 premiers participants. Le prélèvement officiel d’échantillons a débuté le 2 avril 2018. A voir si l’expérience relativement pionnière ,à cette échelle, menée en Estonie, fera école ou tâche d’huile dans d’autres pays de l’Union Européenne !
Vous qui cherchez une méthode permettant de mettre en évidence des variants rares au sein d’une population hétérogène de produits PCR, l’ice-COLD PCR est peut être faite pour vous.
En effet, identifier des variants rares noyés dans de l’ADN « sauvage » a été techniquement une demande forte de divers champs d’expertises médicales tels que la cancérologie, l’infectiologie et le diagnostic prénatal. Nous avons déjà parlé sur Biorigami, d’une approche assez largement répandue qui consiste à séquencer très profondément (à haut-débit donc) tout ou partie de génome en vue d’identifier ces variants rares potentiellement associés à des phénomènes de résistance à des antibiotiques (à titre d’exemple vous pouvez consulter le travail d’Eurekagenomics présenté sous forme de poster)
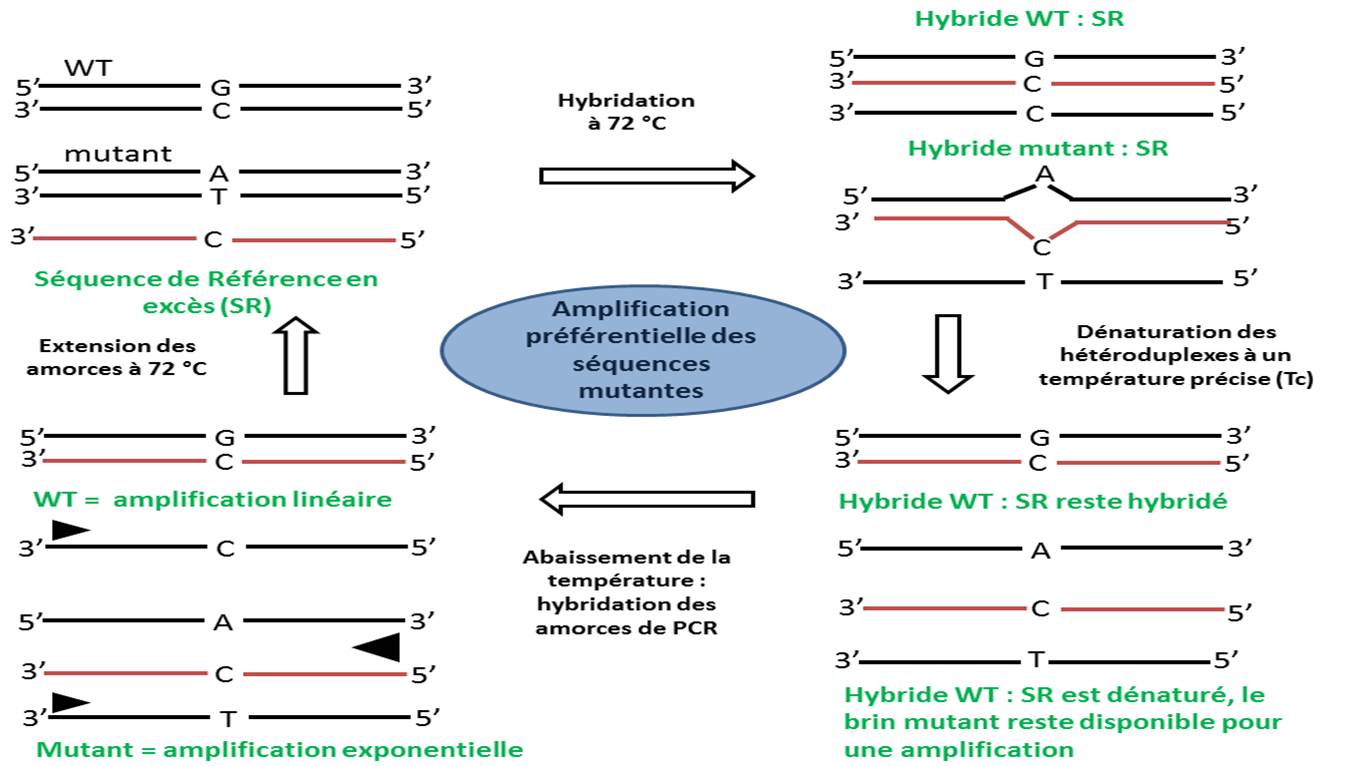
Derrière l’acronyme tautologique, ice COLD, se cache la terminologie : Improved & Complete Enrichment CO-amplification at Lower Denaturation temperature PCR, ce qui pourrait se traduire en français approximatif par : une amélioration de la COLD PCR qui est elle même : une co-amplification à sub-température de dénaturation-PCR. Qu’est ce qui se cache derrière cet énième acronyme ? Pour y voir plus plus clair, voici pour commencer, ce petit schéma:
En observant le schéma ci-dessus, commençons en haut à gauche : nous avons donc des séquences double brin sauvage en large quantité et potentiellement de l’ADN mutant. En premier lieu, il convient de designer des amorces permettant une amplification du locus d’intérêt (pour un amplicon de taille généralement autour de 100 pb). Il vous faudra ensuite une séquence (notée SR sur le schéma) 3′ phosphate pour prévenir des amplifications lors des phases de PCR. Cet oligonucléotide (employé autour d’une concentration de 25 nM finale) viendra s’hybrider parfaitement sur la cible ADN wt et constituera un hétéroduplexe avec toute cible contenant un polymorphisme. A l’aide de la connaissance du Tm (melting temperature ou température de fusion) de l’homoduplexe ADNwt//SR -(note : pour ce faire, il sera nécessaire de designer un autre couple d’amorces afin que la taille du brin SR amplifiée soit égale au brin SR qui lui même est plus court que l’amplicon généré par les premières amorces designées) acquise par la réalisation de courbe de fusion par PCR en temps réel, les hétéroduplexes seront dénaturés en chauffant à une température critique (Tc), généralement de 1 °C inférieure au Tm constaté de l’homoduplexe. Nous sommes, à l’issue de cette étape, en bas à droite de notre schéma, ensuite il restera à réaliser la PCR qui ciblera largement préférentiellement les ADNs disponibles, dénaturés… donc ceux portant des mutations.
Les avantages de cette technique sont multiples :
Cette technique peut être appliquée sur diverses types d’échantillons : ADN fœtal circulant (sérum, plasma), cellules tumorales circulantes, divers fluides corporels, FNA, FFPE, TMA. Elle nécessite de petites quantités de tissu ou d’ADN et peut permettre détecter toutes les mutations présentes au sein du locus amplifié.
En aval, cette méthode accepte une foultitude de techniques de détection qui permettent de réaliser le diagnostic à proprement parlé des mutations :
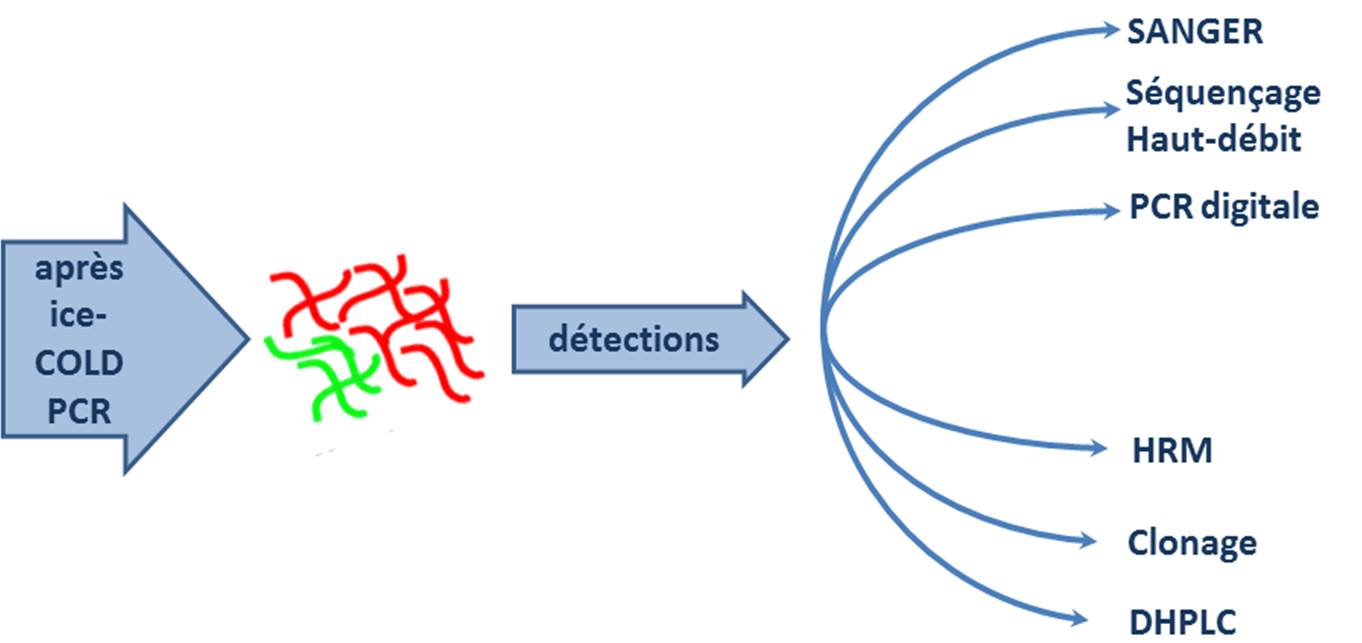
L’ice-COLD PCR est une technique qui est peu exigeante, elle nécessite de disposer d’un thermocycleur assez correct et d’un fournisseur d’oligonucléotides performant (rapide et pas cher) et évidemment de s’y connaitre quelque peu en PCR…
Si, par hasard, vous souhaiteriez plus de précisions, voici une vidéo des plus instructives, l’occasion pour nous de présenter l’excellent site LabTube.com dont une capture d’écran vous est proposée ci-dessus. Ce site internet héberge moult vidéos de conférences, diapositives illustrant une technique (dont l’ice COLD-PCR), bref une source d’informations techniques dont il ne faut pas se priver. Pour preuve au sujet de la recherche de mutations rares voici une présentation de Jorg Tost, directeur du laboratoire épigénétique et environnement (LEE), du Centre national de génotypage, Institut de Génomique/CEA.
La HRM est une technique accessible (en temps/finance) sous diffusée. Loin d’avoir l’ambition de réparer cette injustice (à mes yeux), cet article vise à aborder une technique analytique de biologie moléculaire survolée, méconnue et snobée à l’heure de la biologie haut-débit.
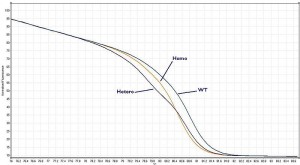
La HRM (High Resolution Melt, Courbe de Fusion à Haute-Résolution) permet de distinguer des amplicons (produits PCR) variant d’un minimum de 1 base… (avec plus ou moins de difficulté selon que la mutation concernée est une transversion A/T ou un INDEL par exemple). Cette technique est l’alliance de machine qPCR permettant un contrôle fin de la température (pour ce qui nous concerne nous sommes les heureux possesseurs d’un RotorGene 6000, de Corbett dorénavant vendu sous le nom de Rotor-Gene Q, Qiagen) et d‘agents intercallants de l’ADN saturants et moins « toxiques » pour la PCR comme peut l’être l’EvaGreen. Au rayon des molécules fluorescentes pour cette application vous pourrez trouver le LC Green et LC Green Plus , le ResoLight, Chromofy, SYTO 9 et le SybrGreen I bien sûr ! (de toute évidence sur la plateforme RotorGene, cette molécule permet de bons résultats). Ainsi que le montre les courbes ci-dessous, après amplification, les produits de PCR sont chauffés jusqu’à complète dénaturation par pas de température les plus fins possibles, plus ceux-ci sont petits, plus la technique est résolutive. Le point d’inflexion (l’endroit où la dérivée s’annule) reporté en abscisses est le Tm du produit PCR préalablement amplifié. Or le Tm d’un produit PCR est dépendant de sa concentration, de la concentration en sels du milieu dans lequel il est solubilisé mais surtout de sa composition en bases azotées.
L’hétéroduplexe (la forme hétérozygote) montrera une courbe avec 2 points d’inflexions (ainsi que vous pouvez l’observer sur la courbe ci-dessus). Les 3 populations : homozygotes AA et BB, ainsi que la forme hétérozygote AB se distinguent entre elles.
Au niveau des stratégies de design puis d’analyse, un document plutôt bien réalisé est disponible ici. Les plateformes permettant de réaliser de « vraies » courbes HRM possèdent un logiciel permettant une normalisation qui facilite grandement l’interprétation des résultats.
Les applications de cette techniques sont évidentes :
– le génotypage à bas débit et très bas coût : de simples amorces suffisent (nul besoin de sondes internes doublement marquées) un mastermix contenant un fluorochrome et le tour est joué (il sera judicieux d’avoir sous la main des échantillons références homozygote des deux formes, au minimum). Bien designé, ce diagnostic de génotypage permet une discrimination des substitutions A/T (SNP de classe 4) les plus difficiles à distinguer compte tenu de la faible différence de Tm (autour des 0,2 °C) entre les deux formes.
– le criblage (screening) de mutants : cette application permet de réaliser un premier filtre en vue d’un séquençage (par exemple) sur des zones courtes de l’ADN potentiellement impliquées dans un phénotype particulier. La reproductibilité et la sensibilité de la technique est correcte comme le montre l’article Tù Nguyen-Dumont et al., BMC 2011
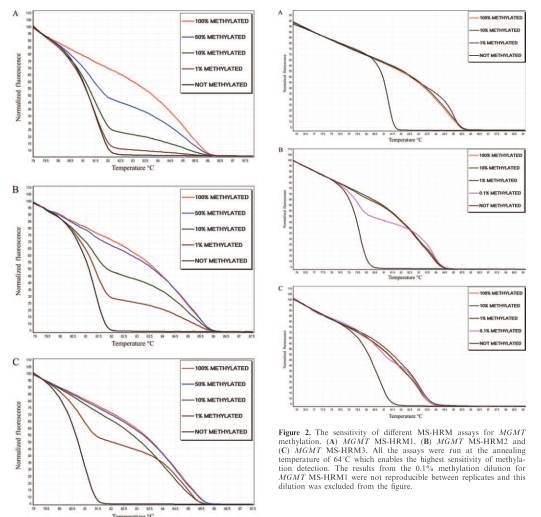
– méthylation de l’ADN : recherche de base 5-méthylcytosine (à l’aide d’un protocole bi-sulfite permettant de « remplacer » les cytosines non méthylées par des uraciles) à l’instar de ce que développe la publication dans NAR de Tomasz K. Wojdac et Alexander Dobrovic. Cette étude montre que l’intermédiaire de la MS-HRM (Methylation-Sensitive High Resolution Melting) il est possible de distinguer des zones méthylées à 0,1 % (ainsi que les courbes tirées de l’article sus-cité montrent pour des sensibilités descendant jusqu’à 1% d’amplicons méthylés, visibles ci-dessous).
En conclusion, la HRM est une technique largement méconnue et sous-employée, relativement simple à développer. Il est nécessaire de normaliser la concentration des échantillons (afin que ceux-ci aient des courbes d’amplification franchissant le seuil autour des 25 Ct). Dans l’idéal, la concentration en sels ne doit pas varier entre les échantillons… appuyé sur un bon design… et le tour est joué. De plus amples informations sont disponibles sur le site gene-quantification.info (la référence en matière de qPCR) ainsi que sur le site highresolutionmelt.co.uk.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages