La HRM est une technique accessible (en temps/finance) sous diffusée. Loin d’avoir l’ambition de réparer cette injustice (à mes yeux), cet article vise à aborder une technique analytique de biologie moléculaire survolée, méconnue et snobée à l’heure de la biologie haut-débit.
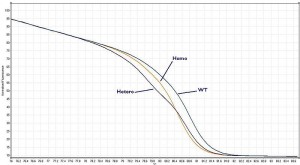
La HRM (High Resolution Melt, Courbe de Fusion à Haute-Résolution) permet de distinguer des amplicons (produits PCR) variant d’un minimum de 1 base… (avec plus ou moins de difficulté selon que la mutation concernée est une transversion A/T ou un INDEL par exemple). Cette technique est l’alliance de machine qPCR permettant un contrôle fin de la température (pour ce qui nous concerne nous sommes les heureux possesseurs d’un RotorGene 6000, de Corbett dorénavant vendu sous le nom de Rotor-Gene Q, Qiagen) et d‘agents intercallants de l’ADN saturants et moins « toxiques » pour la PCR comme peut l’être l’EvaGreen. Au rayon des molécules fluorescentes pour cette application vous pourrez trouver le LC Green et LC Green Plus , le ResoLight, Chromofy, SYTO 9 et le SybrGreen I bien sûr ! (de toute évidence sur la plateforme RotorGene, cette molécule permet de bons résultats). Ainsi que le montre les courbes ci-dessous, après amplification, les produits de PCR sont chauffés jusqu’à complète dénaturation par pas de température les plus fins possibles, plus ceux-ci sont petits, plus la technique est résolutive. Le point d’inflexion (l’endroit où la dérivée s’annule) reporté en abscisses est le Tm du produit PCR préalablement amplifié. Or le Tm d’un produit PCR est dépendant de sa concentration, de la concentration en sels du milieu dans lequel il est solubilisé mais surtout de sa composition en bases azotées.
L’hétéroduplexe (la forme hétérozygote) montrera une courbe avec 2 points d’inflexions (ainsi que vous pouvez l’observer sur la courbe ci-dessus). Les 3 populations : homozygotes AA et BB, ainsi que la forme hétérozygote AB se distinguent entre elles.
Au niveau des stratégies de design puis d’analyse, un document plutôt bien réalisé est disponible ici. Les plateformes permettant de réaliser de « vraies » courbes HRM possèdent un logiciel permettant une normalisation qui facilite grandement l’interprétation des résultats.
Les applications de cette techniques sont évidentes :
– le génotypage à bas débit et très bas coût : de simples amorces suffisent (nul besoin de sondes internes doublement marquées) un mastermix contenant un fluorochrome et le tour est joué (il sera judicieux d’avoir sous la main des échantillons références homozygote des deux formes, au minimum). Bien designé, ce diagnostic de génotypage permet une discrimination des substitutions A/T (SNP de classe 4) les plus difficiles à distinguer compte tenu de la faible différence de Tm (autour des 0,2 °C) entre les deux formes.
– le criblage (screening) de mutants : cette application permet de réaliser un premier filtre en vue d’un séquençage (par exemple) sur des zones courtes de l’ADN potentiellement impliquées dans un phénotype particulier. La reproductibilité et la sensibilité de la technique est correcte comme le montre l’article Tù Nguyen-Dumont et al., BMC 2011
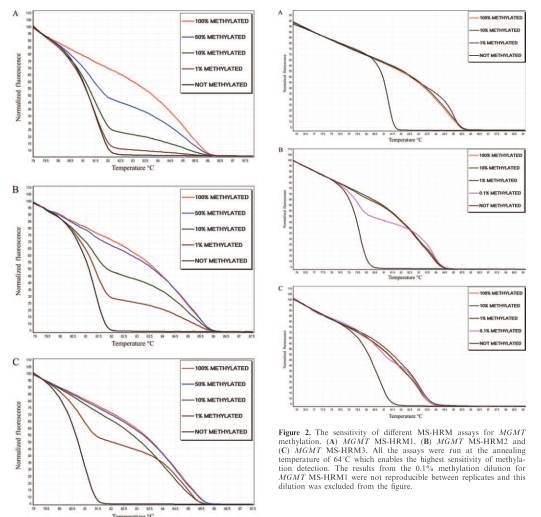
– méthylation de l’ADN : recherche de base 5-méthylcytosine (à l’aide d’un protocole bi-sulfite permettant de « remplacer » les cytosines non méthylées par des uraciles) à l’instar de ce que développe la publication dans NAR de Tomasz K. Wojdac et Alexander Dobrovic. Cette étude montre que l’intermédiaire de la MS-HRM (Methylation-Sensitive High Resolution Melting) il est possible de distinguer des zones méthylées à 0,1 % (ainsi que les courbes tirées de l’article sus-cité montrent pour des sensibilités descendant jusqu’à 1% d’amplicons méthylés, visibles ci-dessous).
En conclusion, la HRM est une technique largement méconnue et sous-employée, relativement simple à développer. Il est nécessaire de normaliser la concentration des échantillons (afin que ceux-ci aient des courbes d’amplification franchissant le seuil autour des 25 Ct). Dans l’idéal, la concentration en sels ne doit pas varier entre les échantillons… appuyé sur un bon design… et le tour est joué. De plus amples informations sont disponibles sur le site gene-quantification.info (la référence en matière de qPCR) ainsi que sur le site highresolutionmelt.co.uk.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages