Pour rien vous cacher, il s’agit ici d’un article avec un petit goût d’auto-promotion. En effet, les 3 membres de Biorigami participent aussi activement à une plateforme d’expertise nouvellement créée, cette entité a pour ambition la réalisation de quelques prestations, principalement en génomique haut-débit, mais aussi la participation à des projets nécessitant conseils, expertises et analyses… pour répondre au mieux aux questions biologiques posées. Enchaînons en citant les pages actualités du site internet de Gènes Diffusion : « cette alliance entre les deux structures (ndlr : Gènes Diffusion et l’Institut Pasteur de Lille) a pris le nom de PEGASE (Plateforme d’Expertises Génomiques Appliquées aux Sciences Expérimentales). Elle permet de mutualiser les moyens de la branche R&D génomique de Gènes Diffusion et du laboratoire TAG (Transcriptomic analysis and Applied Genomics) de l’Institut Pasteur de Lille.
La création de cette unité devrait permettre le développement de nouvelles activités, collaborations scientifiques et prestations, dès les prochains mois
Issue de la collaboration entre Gènes Diffusion et l’Institut Pasteur de Lille, PEGASE (Plateforme d’Expertises Génomiques Appliquées aux Sciences Expérimentales) vient de lancer son site web. Cette présence sur le web permettra à la nouvelle structure de faire l’interface avec les différents acteurs de la génomique.
Par ailleurs, le site permettra également de faire partager à la communauté scientifique les différentes expertises en biologie moléculaire et bio-informatique issues des travaux de recherche de l’unité. »
Vous pourrez obtenir plus de précision concernant cette nouvelle plateforme en suivant le lien suivant : www.pegase-biosciences.com.
Pour les plus joueurs d’entre vous, un concours est lancé pour fêter cette naissance, le projet exécutable sur puce 318 de Ion Torrent, sera réalisé par nos soins, gratuitement… pour plus de précisions, nous vous invitons à suivre le lien suivant : concours PEGASE.
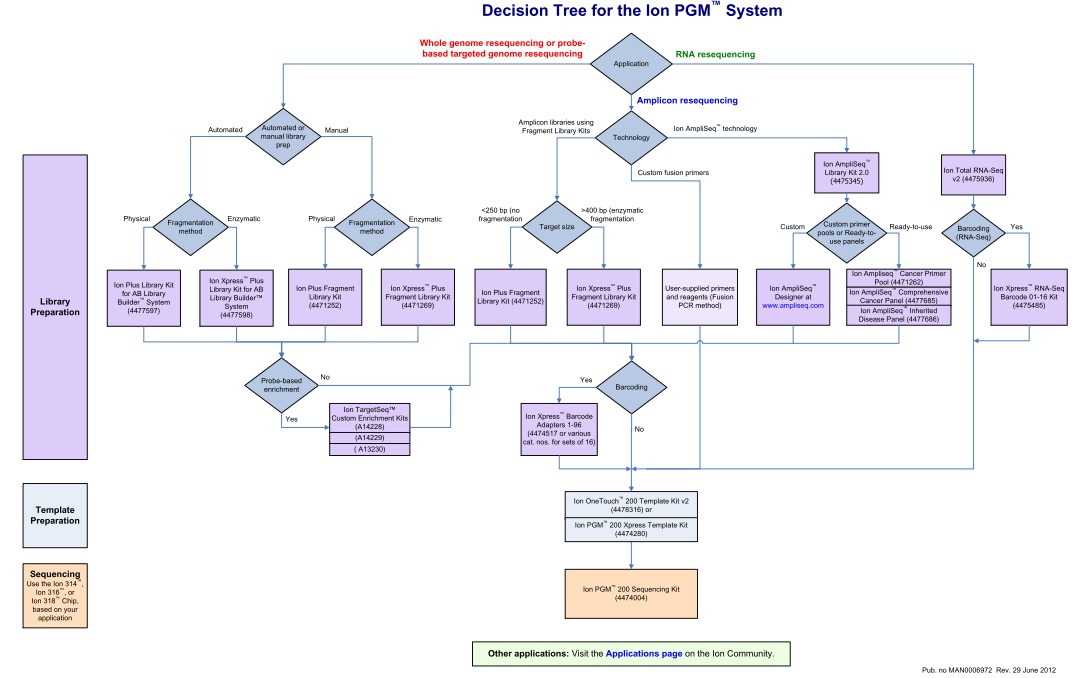
Un document qui n’est pas sans rappeler celui publié en novembre 2011, en intégrant les dernières mises à jour.
Cet arbre de décision est l’occasion de retrouver l’ensemble des modalités de séquençage qu’offre la technologie Ion Torrent via le PGM. En attendant l’arrivée du kit permettant de séquencer 400b, l’essentiel des modifications résident dans la préparation des échantillons avec notamment de nouvelles indexations (évolution du kit à 96 barcodes en DNA-seq et apparition du kit à 16 barcodes en RNA-seq), un élargissement du panel de primers dédiés à la recherche sur le cancer, etc…
Pour les privilégiées de la Ion Community, ce document est également l’occasion de retrouver aisément la liste des protocoles Ion Torrent selon l’ application d’intérêt en cliquant sur le lien en bas de page.
 Les outils d’assemblage, de mapping sur référence deviennent relativement pléthoriques… il est de ce fait relativement aisé de choisir l’outil permettant de gérer au mieux les défauts de votre séquenceur, vous n’avez que l’embarras du choix en somme. Même si cette étape n’est pas à négliger, il n’en reste pas moins que le rôle du biologiste restera toujours d’apporter du sens à une ou plusieurs millions de séquences, informations qui viendront tenter d’élucider une question biologique. Les outils permettant de faire parler un ensemble de reads, que vous pratiquiez le de novo RNA-seq ou séquençage génomique de novo sont plutôt restreints, peu diffusés, limités ou tout simplement absents… Ces grands absents constituent le point noir de l’analyse secondaire de vos reads. De nombreux biologistes recherchent un financement (séquencer massivement coûte encore un peu cher !), font séquencer l’objet de leur étude -hors humain, rat, souris- repartent quand tout va bien avec de beaux contigs, et se retrouvent face à un livre écrit dans une langue inconnue. Ce livre est censé renfermer la réponse à beaucoup de leurs questions… on comprend leur frustration d’autant qu’ils ne possèdent pas de pierre de Rosette.
Les outils d’assemblage, de mapping sur référence deviennent relativement pléthoriques… il est de ce fait relativement aisé de choisir l’outil permettant de gérer au mieux les défauts de votre séquenceur, vous n’avez que l’embarras du choix en somme. Même si cette étape n’est pas à négliger, il n’en reste pas moins que le rôle du biologiste restera toujours d’apporter du sens à une ou plusieurs millions de séquences, informations qui viendront tenter d’élucider une question biologique. Les outils permettant de faire parler un ensemble de reads, que vous pratiquiez le de novo RNA-seq ou séquençage génomique de novo sont plutôt restreints, peu diffusés, limités ou tout simplement absents… Ces grands absents constituent le point noir de l’analyse secondaire de vos reads. De nombreux biologistes recherchent un financement (séquencer massivement coûte encore un peu cher !), font séquencer l’objet de leur étude -hors humain, rat, souris- repartent quand tout va bien avec de beaux contigs, et se retrouvent face à un livre écrit dans une langue inconnue. Ce livre est censé renfermer la réponse à beaucoup de leurs questions… on comprend leur frustration d’autant qu’ils ne possèdent pas de pierre de Rosette.
Cet article part d’un constat, l’important est moins de savoir si votre séquenceur fournit les reads les plus propres, les plus longs, si votre assembleur est le plus adapté… car tous ces efforts resteront vains si vous écrivez un livre dont personne ne peut comprendre le sens. J’arrête là de filer la métaphore.

Blast2GO -ici dans sa version gratuite- est un outil qui trouvera vite (trop vite) ses limites mais qui permettra d’apporter du sens à des séquences générées à haut-débit. La publication de Conesa et al. (Bioinformatics – 2005), dévoile le pipeline d’analyse. Grossièrement, il s’agit de blaster des séquences (en 2005 – on envisageait par séquences des reads et non des reads assemblés) automatiquement sur le serveur du NCBI, les résultats de blast sont récupérés, les autres phases du pipeline sont réalisées en interrogeant les bases de données de Blast2GO, in fine des termes GO permettent d’annoter les séquences dont on souhaite décoder le sens. La principale qualité de Blast2GO réside dans son ergonomie, l’interface Java6 permet une prise en main directe (aucune ligne de code). Ses défauts sont multiples… tout d’abord Blast2GO plante… très régulièrement, ensuite pour une séquence d’entrée (dont la taille sera nécessairement inférieure à 7000 bases) vous ne disposerez que d’une annotation pour le meilleur des hits.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages