Comment réagiriez vous si l’on vous « offrait » votre génotypage complet, ouvrant la possibilité de prédire d’éventuelles maladies, réactions à certains médicaments etc. ? Certes, dans le cas de l’Estonie (contrairement à ce que propose des sociétés privées telles 23andMe) il s’agit d’une démarche s’inscrivant en santé publique pour le meilleur du bien public et non pour le pire du bien privé… Néanmoins, l’ampleur de la cohorte humaine visée n’est pas sans poser des questions.
L’Estonie a lancé un programme visant à recruter et à génotyper 100.000 nouveaux participants à la biobanque (pour une population nationale totale de 1,316 million de citoyens estoniens !) dans le cadre de son programme national de médecine personnalisée. Cette biobanque comportait déjà 50.000 génotypes de citoyens estoniens. Le gouvernement veut développer un système de soins de santé en offrant à un maximum de ses résidents un génotypage scannant le génome qui sera traduit en rapports personnalisés. Ce rapport serait intégré à la pratique médicale quotidienne par l’entremise du portail national de cybersanté.
Cette biobanque a été initiée à trois fins (Leitsalu et al., International Journal of Epidemiology, 2015) :
- promouvoir le développement de la recherche génétique ;
- recueillir des informations sur l’état de santé de la population estonienne, ainsi que des informations génétiques
- utiliser les résultats de la recherche génétique pour améliorer la santé publique.

Au sein de l’article de Leitsalu et al., les auteurs abordent les forces et faiblesses de leur entreprise :
La principale force de la Biobanque estonienne consiste au fait qu’il s’agit d’une biobanque basée sur la population avec une base de données longitudinales et prospectives. Cela signifie qu’un large éventail de groupes d’âge et de phénotypes sont représentés. Alors que les populations urbaines ont généralement tendance à être surreprésentées, ce n’est pas le cas pour la cohorte de la Biobanque estonienne. La biobanque dispose d’ADN, de plasma et de globules blancs pour chaque donneur. Cela signifie qu’il est possible d’analyser les effets directs des variants de séquence sur le métabolisme. De plus, il est possible de transformer les cellules en lignées cellulaires ou en cellules souches pluripotentes induites (iPS) et de réaliser directement des expériences de biologie moléculaire ou de génétique. Une autre force est fournie par la HGRA (the Estonian Human Genes Research Act) ainsi que par le formulaire de consentement général qui permet de participer à un large éventail de projets de recherche sans avoir à communiquer de nouveau et à demander un nouveau consentement. La HGRA et le formulaire de consentement permettent également aux donneurs de demander la divulgation de leurs données génétiques, de leurs caractéristiques héréditaires et des risques génétiques obtenus à partir de la recherche génétique menée. Cela permettrait à terme de mener des projets sur les tests du génome personnel, la perception des risques et la gestion des risques en milieu industriel.
De plus, la HGRA permet à la Biobanque d’obtenir des renseignements supplémentaires en reliant les dossiers aux registres électroniques nationaux et aux principaux hôpitaux. Tous les registres sont reliés de façon centralisée par une infrastructure technique à l’échelle nationale qui permet l’échange sécurisé de données entre les bases de données. La HGRA a également imposé des restrictions sur les activités de l’EGCUT (Estonian Genome Center of the University of Tartu) et les données collectées dans la Biobanque estonienne. La participation devait être entièrement volontaire – seules les personnes intéressées pour rejoindre la Biobanque estonienne, après en avoir entendu parler soit lors d’événements promotionnels spéciaux, soit par les médias, soit par des amis, soit au cabinet du médecin de famille ou à l’hôpital, sont recrutées. L’EGCUT n’a pas été autorisé à envoyer les lettres d’invitation à leur adresse domiciliaire. Par conséquent, la biobanque ne représente pas un échantillon aléatoire classique et pourrait être sujette à un biais de recrutement. Une proportion considérable de la population recrutée pourrait toutefois compenser ce biais. Par conséquent, bien qu’elle ne soit pas aléatoire sur le plan classique, la cohorte peut quand même être considérée comme représentative de la population. Bien que le recrutement était ouvert à tous, il y a une disproportion d’Estoniens ethniques et de Russes ethniques dans la biobanque, les Estoniens étant surreprésentés (81% dans la biobanque contre 70% dans la population générale) et les Russes sous-représentés (16% dans la biobanque contre 25% dans la population générale). Une autre faiblesse est la profondeur limitée de certains sous-questionnaires. Par exemple, un questionnaire relativement bref sur la fréquence des aliments a été administré sans information détaillée sur l’apport en énergie ou en nutriments ; les mesures des traits glycémiques à jeun, comme le niveau d’insuline, ne sont disponibles que pour un nombre limité d’échantillons. La profondeur limitée des données recueillies peut parfois limiter le nombre de projets dans lesquels les données peuvent être utilisées. Toutefois, des questionnaires plus complets auraient exigé des durées d’entrevue encore plus longues et auraient coûté beaucoup plus cher, ce qui aurait pu entraîner une réduction de la taille de la cohorte.
Le pays dispose de nombreuses solutions numériques sécurisées incorporées dans les fonctions gouvernementales qui relient les diverses bases de données du pays par des voies cryptées de bout en bout. Un site Web a été créé dans le cadre du projet, afin que les Estoniens puissent se porter volontaires et donner leur consentement à être génotypés. La génération des données est assurée par l’institut de génomique de l’Université de Tartu (aujourd’hui les 50.000 premiers génotypages ont été réalisés et analysés, les 100.000 pousseront à une population estonienne à 10 % génotypée)
Les efforts internationaux ont permis d’identifier des milliers d’associations entre les variants génétiques et les maladies, ou traits génétiques, et de créer des cartes des variations uniques au sein des populations.
« Aujourd’hui, nous avons suffisamment de connaissances sur le risque génétique des maladies complexes et la variabilité interindividuelle des effets des médicaments pour commencer à utiliser systématiquement ces informations dans les soins de santé au quotidien « , a déclaré Jevgeni Ossinovski, ministre de la Santé et du Travail. « En coopération avec l’Institut national pour le développement de la santé et l’Université de Tartu, nous allons permettre à 100.000 autres personnes de rejoindre la biobanque estonienne, afin de stimuler le développement de la médecine personnalisée en Estonie et de contribuer ainsi à l’avancement des soins de santé préventifs. «
Le gouvernement estonien a alloué 5 millions d’euros au programme au cours de l’année 2018. Le projet sera coordonné par l’Institut national pour le développement de la santé, dont la tâche est d’élaborer et de mettre en œuvre des procédures et des principes pour la mise en œuvre efficace de la recherche scientifique dans la pratique médicale.
Andres Metspalu, directeur du Centre estonien du génome à l’Université de Tartu, se félicite de l’initiative du ministère des Affaires sociales d’augmenter le nombre de participants à la biobanque. « Nous sommes heureux qu’avec le soutien de ce projet, les résultats des travaux à long terme du centre de génomique seront transférés en médecine pratique et donneront un nouvel élan à nos recherches futures. L’Université contribuera également à la création d’un système de rétroaction pour les participants de la biobanque, et à la formation des professionnels de la santé pour qu’ils puissent donner aux patients une rétroaction fondée sur l’information génétique« .
Le projet sera mis en œuvre sur la base de la loi estonienne sur la recherche sur les gènes humains et du même formulaire de consentement général qui a été utilisé pour les 50.000 premiers participants. Le prélèvement officiel d’échantillons a débuté le 2 avril 2018. A voir si l’expérience relativement pionnière ,à cette échelle, menée en Estonie, fera école ou tâche d’huile dans d’autres pays de l’Union Européenne !
 Si vous vous intéressez au séquençage haut-débit, que vous souhaitez avoir un panorama des diverses technologies à disposition et que vous êtes friands de schémas de principe: la publication de Sara Goodwin, John D. McPherson & W. Richard McCombie. Cet article publié dans la revue Nature de mai 2016 promet de faire un retour en arrière sur 10 ans d’évolution du séquençage haut-débit. Elle parvient à tenir ses promesses et livre effectivement des schémas (avec la charte graphique « Nature ») très bien faits, très pédagogiques ! En outre, le tableau (Table 1 | Summary of NGS platforms) permet à tous les pourvoyeurs de projets nécessitant le recours à du séquençage, d’avoir un pense bête sous la main pour associer la bonne technologie à la question biologique qui leur incombe… Et comme vous êtes pressés, vous pourrez retrouver l’intégralité de cet article en vous promenant et cliquant sur l’image ci-dessous.
Si vous vous intéressez au séquençage haut-débit, que vous souhaitez avoir un panorama des diverses technologies à disposition et que vous êtes friands de schémas de principe: la publication de Sara Goodwin, John D. McPherson & W. Richard McCombie. Cet article publié dans la revue Nature de mai 2016 promet de faire un retour en arrière sur 10 ans d’évolution du séquençage haut-débit. Elle parvient à tenir ses promesses et livre effectivement des schémas (avec la charte graphique « Nature ») très bien faits, très pédagogiques ! En outre, le tableau (Table 1 | Summary of NGS platforms) permet à tous les pourvoyeurs de projets nécessitant le recours à du séquençage, d’avoir un pense bête sous la main pour associer la bonne technologie à la question biologique qui leur incombe… Et comme vous êtes pressés, vous pourrez retrouver l’intégralité de cet article en vous promenant et cliquant sur l’image ci-dessous.
Plutôt bien réalisée, voici la campagne d’Illumina pour le lancement de son nouveau séquenceur le NextSeq500, un nouveau séquenceur de paillasse. Ce dernier né des séquenceurs SBS est capable de délivrer de 16 à 120 Gb, selon les modes de séquençage, avec des spécifications permettant d’envisager le séquençage par run de 16 exomes ou 20 transcriptomes ou encore 1 génome humain (à plus de 30 X de profondeur). Quoi qu’il en soit, ce nouveau venu et la campagne marketing qui en résulte, visent à populariser, démocratiser encore et toujours le séquençage pour mieux inonder le marché, c’est parti avec cette publicité…
En ce début d’année, cet article est l’occasion d’aborder rapidement les divers axes de développements, les différents acteurs du séquençage haut-débit de deuxième génération.
– Commençons par Life Technologies et sa gamme Ion Torrent. En fin d’année 2013, la Ion Community (forum où se retrouvent les utilisateurs de la technologie Ion Torrent) s’agite à l’annonce de 3 nouveautés majeures (early access program) :
(i) L’accès à une nouvelle chimie de séquençage, la Hi-Q ™, permettant d’accroître la fiabilité de séquençage. Les erreurs seraient réduites de 90 %, ceci même au niveau des homopolymères, et pour des reads de 400 bases, témoignage de Dag Harmsen à l’appui ! En clair, il semble que ce soit l’enzyme (what else ?) qui ait été remplacé.
(ii) La deuxième annonce concerne l’arrivée de la chimie Avalanche où plusieurs heures d’amplification clonale à l’aide d’un automate One-Touch peuvent être remplacées par l’emploi d’un tube, ce qui prend alors 2 heures pour obtenir une librairie de 500 pb, et ce, de façon isothermique. Un choc de simplification qui ravira les utilisateurs pour lesquels cette étape est limitante.
(iii) La troisième annonce concerne la mise à disposition de kits permettant de réaliser des analyses métagénomiques ciblées 16 S. Un système exploitant le PGM et sa capacité de produire des reads de 400 pb. L’inconnu ici réside dans la mise à disposition de la communauté d’un pipeline analytique performant.
– Qiagen, qui n’est pas connu pour être un acteur de poids sur la scène du séquençage haut-débit, arrive en force en cette année 2014 avec une solution intégrant tous les jalons nécessaires à la complétude d’une étude. Fort de son rachat d’une solution de séquençage (lire l’article : Qiagen investit… le séquençage haut-débit de 2ème génération), Qiagen propose un environnement logiciel des plus intéressants ! En effet, la société néerlandaise a racheté les sociétés CLC Bio et Ingenuity systems. Ces deux sociétés proposent l’une des toutes meilleures solutions d’analyse de séquences: une solution d’assemblage de novo réellement performante grâce à CLC genomics workbench, et Ingenuity systems proposant les pipelines d’analyses suivants: IPA, pour donner un sens biologique aux données omiques, Ireport pour l’analyse de données d’expression et Variant Analysis, un pipeline permettant d’optimiser la recherche de mutations causales.
Ainsi QIAGEN, à l’instar de ce que nous avons tâché de représenter par le schéma ci-dessous, possède actuellement tous les maillons (ou pas loin) d’une chaîne allant de l’échantillon à l’analyse finale traduisant des données de séquences en sens biologique.

– Illumina, quant à elle, semble avoir l’ambition de devenir une sorte de Apple de la « génomicosphère ». En effet, Illumina propose BaseSpace, un Itunes pour les biologistes. D’ailleurs, notons qu’Illumina propose sur Itunes une application : MyGenome, qui propose « d’explorer un véritable génome humain » et d’afficher des rapports sur les variations génétiques importantes. « L’application MyGenome fournit une interface simple, intuitive, et éducative pour vous lancer à la découverte du génome humain« . Revenons à BaseSpace, une interface entre vous et un cloud hébergeant des applications et des données permettant d’analyser les séquences en sortie de MiSeq ou HiSeq. Ce cloud permet aux utilisateurs de délocaliser le stockage de leurs données. L’idée : simplifier au maximum l’analyse par la mise à disposition d’outils et la mise en réseau des utilisateurs. Illumina s’est aperçu que si le goulot d’étranglement constitué par l’analyse de données de séquençage haut-débit volait en éclat, nécessairement les runs pourraient se multiplier avec leur chiffre d’affaire. Le schéma ci-dessous reprend quelques éléments de la solution analytique développée par Illumina.
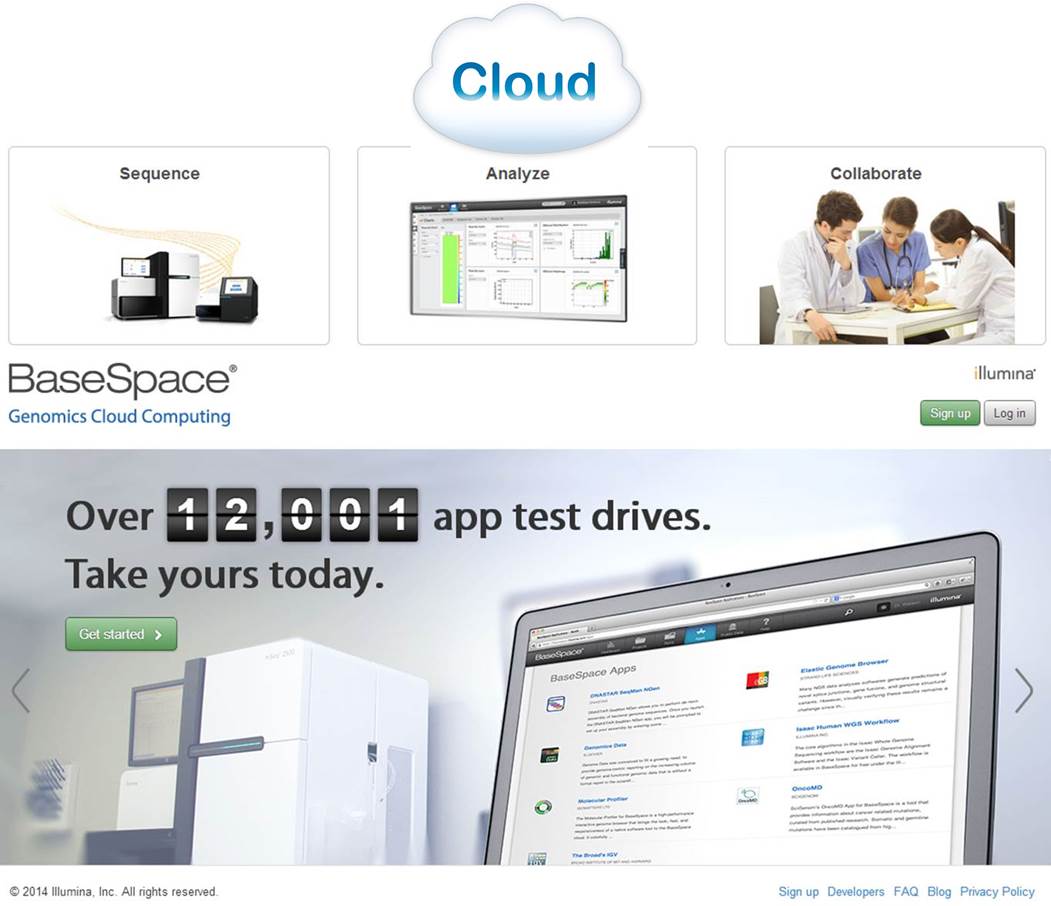
Une communauté de plus de 12000 utilisateurs, un espace permettant l’utilisation d’une vingtaine d’applications. L’objectif d’Illumina : créer un espace attractif, émulant et incitant les intervenants à mettre à disposition les applications développées en priorité sur cet espace. Anticipant une demande exponentielle d’analyses et d’espace de stockage lorsque le HiSeq a été intégré au BaseSpace, Illumina a décidé de mettre en place une politique de tarification qui limiterait la quantité d’espace libre pour stocker et traiter les données génomiques dans le cloud. En vertu de cette logique, les utilisateurs reçoivent un téraoctet gratuit d’espace pour le stockage et le traitement des données et seraient alors en mesure d’acheter du stockage supplémentaire par incréments de téraoctet ou 10 téraoctets – un téraoctet coûterait 250 $ par mois ou $ 2,000 d’avance pour une année complète , tandis que 10 téraoctets seraient à 1500 $ par mois ou une avance des frais annuels de $ 12 000 (données chiffrées début 2013).
En conclusion, si les années précédentes ont vu le lancement de nouveaux séquenceurs, avec depuis 2011 l’arrivée de séquenceurs de paillasse, les années 2013-2014 attendent la diffusion de séquenceurs de 3ème génération. Qiagen est un petit nouveau dans la course, ce nouvel acteur est capable, sans réel développement, de proposer une solution complète grâce à une stratégie de rachat pertinente. Illumina et Life Technologies, pendant ce temps, poursuivent leur développement en essayant d’émuler les utilisateurs avec, respectivement, leur BaseSpace et Ion Community. L’opérateur historique, Roche est le grand silencieux avec une stratégie peu lisible…

Le séquençage haut-débit voit cohabiter depuis quelques années deux générations de séquenceurs.
Au passage, une question Trivial Pursuit pour laquelle il faudra avoir un œil de caracal : quelqu’un sait quelle société a développé la première génération de séquenceurs haut-débit ? et quand ?
Les séquenceurs de deuxième génération se voient conditionnés sous forme de séquenceurs de paillasse (PGM de Ion Torrent, Miseq d’Illumina, GS-junior de Roche) permettant une démocratisation du séquençage, pendant que leurs grands frères pulvérisent la loi de Moore pour envisager un rendement (coût / Mb) toujours plus compétitif.
La large diffusion du séquençage de 3ème génération se laisse désirer laissant le champ libre à la génération précédente. Cet article vise à réaliser un court état des lieux du séquençage haut-débit de troisième génération : un futur plus ou moins lointain, de nouvelles applications potentielles.
La question : séquenceurs de 3ème génération, l’âge de raison, c’est pour quand ? est l’interrogation qui a hanté l’AGBT 2013 marqué par le silence d’Oxford Nanopore. Cette année 2013 fut marquée par le retrait d’Illumina du capital de la société britannique : « Oxford Nanopore Technologies Ltd a annoncé la vente d’une participation détenue par son concurrent américain Illumina Inc., une étape vers la fin d’une relation pleine de conflits dans la course au développement des séquenceurs haut-débit permettant de séquencer plus rapidement et pour moins cher. »
Avant de caractériser ce que sont, seront, pourront être les 3ème générations de séquenceurs, commençons par un rapide tour des caractéristiques générales de leurs prédécesseurs et principalement de ce qui constitue leurs points faibles :
– la phase d’amplification clonale (réalisée par PCR) est source de biais (doublons, erreurs de PCR)
– les problèmes liés au déphasage engendrant une chute de la qualité le long du read produit (ce qui bride la production de reads vraiment longs)
-des reads courts (de moins d’une centaine à environ 800 bases – vous l’aurez noté ce point est en partie une conséquence du précédent)
-des machines et des consommables onéreux
– des temps de run longs
Ainsi l’objectif principal des séquenceurs de 3ème génération est de palier les défauts de leurs aînés en produisant des reads plus longs, plus vite pour moins cher. Les séquenceurs de 2ème génération, quels que soient leurs modes de détection (mesure de fluorescence, mesure de pH) sont trop peu sensibles pour envisager la détection d’une simple molécule, d’un simple nucléotide : nécessairement la librairie doit être amplifiée, ce qui provoque des biais, des temps de préparation relativement longs et l’usage de consommables qui impacte le coût final de séquençage… assez rapidement la qualité chute plus vos reads s’allongent ce qui oblige à brider les tailles de reads que ces technologies sont capables de délivrer. En outre, travaillant sur une matrice qui est une copie de votre librairie initiale, l’information portée par les bases méthylées est perdue (ceci oblige à ajouter une phase de traitement au bisulfite qui peut être hasardeuse)
Actuellement l’une des seules technologies de 3ème génération réellement utilisée est celle de Pacific Biosciences (les hipsters disent « PacBio »). La firme, fondée en 2004, a lancé en 2010, son premier séquenceur de troisième génération le Pacbio RS basé sur une technique de séquençage SMRT (Single Molecule Real Time sequencing.) Aujourd’hui la société Roche qui n’a pu absorber Illumina lors de son OPA, a investi 75 millions de USD, le 25 septembre 2013, pour co-développer des kits diagnostiques in vitro exploitant la technologie de PacBio.
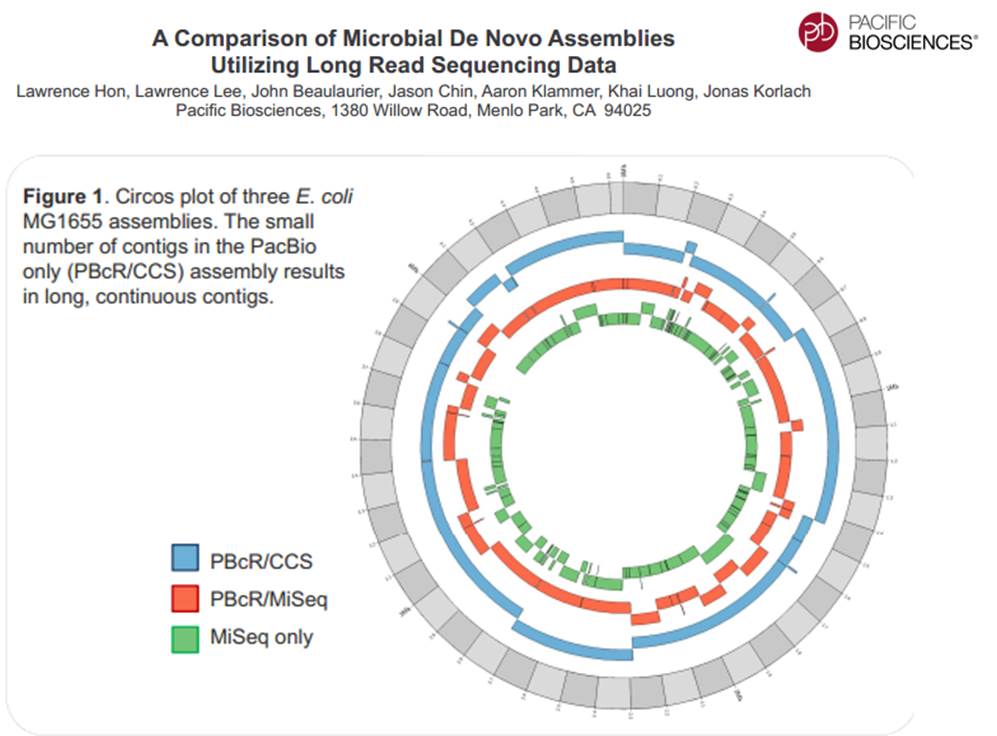
La technologie de PacBio est aujourd’hui exploitée pour réaliser du séquençage de novo de petits génomes :
Avec ces 200 à 300 Mb délivrés par SMRT-cell, séquencer des organismes eucaryotes supérieurs demande un investissement important, malgré tout, cette technologie délivrant des reads de plusieurs milliers de bases, permet d’envisager une diminution du nombre de contigs obtenus par les seules stratégies reads-courts / gros débit.
Face à la technologie proposée par PacBio, d’autres technologies essaient d’émerger pour arriver à occuper le marché du séquençage de 3ème génération :
– La combinaison détection optique et multipore est une voie envisagée pour le séquençage de 3ème génération avec le travail mené par NobleGen biosciences.
– L’imagerie directe de l’ADN
Le microscope électronique offre une résolution possible jusqu’à 100 pm, de sorte que les biomolécules et les structures microscopiques tels que des virus, des ribosomes, des protéines, des lipides, des petites molécules et des atomes même simples peuvent être observés. Bien que l’ADN est visible lorsqu’on l’observe avec un microscope électronique, la résolution de l’image obtenue n’est pas suffisamment élevée pour permettre le déchiffrement de la séquence, c’est à dire, le séquençage de l’ADN. Cependant, lors du marquage différentiel des bases de l’ADN avec des atomes lourds ou des métaux, un tel séquençage devient possible.

– Le séquençage à l’aide de transistor (Transistor-mediated DNA sequencing– une technologie développée par IBM)

Dans le système conceptualisé par IBM, l’ADN est contraint de passer par le pore à cause de la tension électrique subie, la vitesse de passage de la molécule à séquencer est maîtrisée à l’aide de contacts métalliques à l’intérieur du nanopore. La lecture des bases serait réalisée lors du passage de l’ADN simple brin au travers du pore (ça rappelle quelque chose…)
– Et Oxford Nanopore dans tout cela ? Si la société anglaise a annoncé la vente de la participation d’Illumina, elle a marqué l’année 2013 par son silence assourdissant. Passé l’oxymore, en cette fin d’année, coup de poker ou réel lancement, Oxford Nanopore propose un programme d’accès à sa technologie Minion où pour 1000 USD, il est possible de postuler à l’achat des clés USB de séquençage.
La stratégie d’Oxford Nanopore est basée, en partie, sur la possible démocratisation du séquençage de 3ème génération, elle s’oppose à celle de PacBio qui mise sur son arrivée précoce sur le secteur du séquençage haut-débit : décentralisation contre l’inverse. En clair, l’investissement d’un PacBio est tel que l’outil est réservé à des centres, des prestataires de services pouvant assumer cet investissement, ce qui oblige à centraliser les échantillons pour les séquencer, contre les produits (encore en développement) d’Oxford Nanopore dont la promesse est : le séquençage pour tous (ou presque).
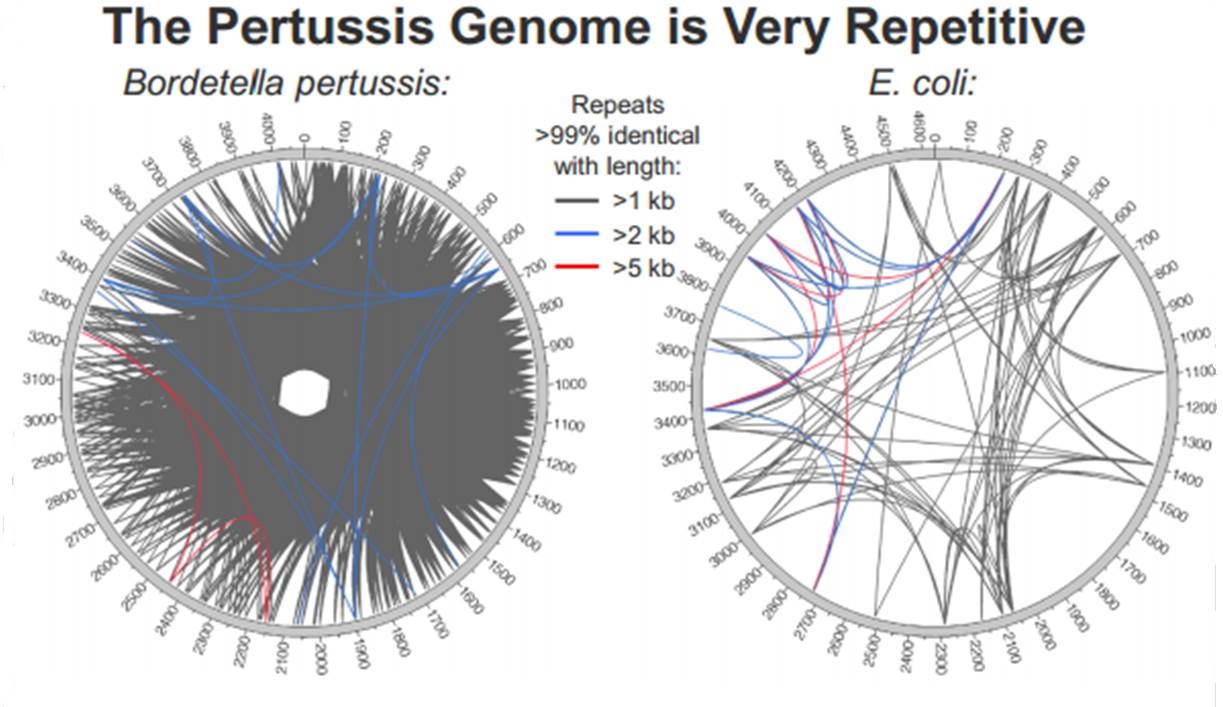
PacBio revendique sa participation à un projet qui consiste à doubler la quantité de génomes bactériens « terminés » (actuellement de 2384) en quelques mois.
En cliquant ci-dessus sur la représentation graphique qui illustre la différence de plasticité de génome entre le génome d’une Bordetella pertussis (l’agent pathogène responsable de la coqueluche) et celui d’Escherichia coli, un poster vous apparaîtra. Ce dernier reprend les caractéristiques de l’utilisation de la technologie de PacBio à des fins d’assemblage de novo de génomes bactériens par une stratégie non-hybride (seuls des reads de PacBio sont utilisés). Les résultats sont assez bluffants, la longueur des reads de PacBio permet un assemblage complet (au prix de plusieurs SMRT cells tout de même !), de génomes bactériens « difficiles » tel que celui de Bordetella pertussis connu pour posséder un GC % relativement élevé (environ 65 %) ainsi que de nombreux éléments transposables. Les génomes possédant de nombreux éléments répétés posent de grandes difficultés d’assemblage, c’est un des arguments qui permet à PacBio de positionner sa technologie actuellement… en quelques mois les stratégies hybrides (reads courts générés par des séquenceurs de 2ème génération) ont laissé place aux stratégies non-hybrides où le séquençage PacBio se suffit à lui-même.
 23andMe, comme Knome et Decode, est une société californienne de biotechnologie pionnière dans la génomique personnelle. La société a été lancée en 2006 par Anne Wojcicki, Linda Avey et Paul Cusenza sur la base d’un concept marketing simple et audacieux : mettre des tests génétiques à disposition du grand public en les vendant en direct sur une plateforme internet. Aujourd’hui la PME revendique 45 employés, 20 millions de USD et une omniprésence sur le web. Le modèle de développement de 23andMe passe par une exploitation judicieuse d’un site web, leur meilleur VRP. La photo du couple ci-dessus représente Anne Wojcicki, la co-fondatrice de 23andMe et Sergey Brin le co-fondateur avec Larry Page de Google, la société maîtresse du web. Google a d’ailleurs investi près de 4 millions de dollars dans la PME de Madame dès ses débuts en mai 2007. Notez qu’à l’époque le test génétique vendu par la toute jeune société de biotechnologie coûtait 999 USD (le prix aura donc été divisé par 10 en 5 ans). Pour la petite histoire, la mère de Sergey Brin est atteinte de la maladie de Parkinson, ce qui a incité le milliardaire à réaliser un test génétique en 2006, pour découvrir qu’il était porteur de la mutation du gène LRRK2. Ceci explique son intérêt pour les tests génomiques personnels.
23andMe, comme Knome et Decode, est une société californienne de biotechnologie pionnière dans la génomique personnelle. La société a été lancée en 2006 par Anne Wojcicki, Linda Avey et Paul Cusenza sur la base d’un concept marketing simple et audacieux : mettre des tests génétiques à disposition du grand public en les vendant en direct sur une plateforme internet. Aujourd’hui la PME revendique 45 employés, 20 millions de USD et une omniprésence sur le web. Le modèle de développement de 23andMe passe par une exploitation judicieuse d’un site web, leur meilleur VRP. La photo du couple ci-dessus représente Anne Wojcicki, la co-fondatrice de 23andMe et Sergey Brin le co-fondateur avec Larry Page de Google, la société maîtresse du web. Google a d’ailleurs investi près de 4 millions de dollars dans la PME de Madame dès ses débuts en mai 2007. Notez qu’à l’époque le test génétique vendu par la toute jeune société de biotechnologie coûtait 999 USD (le prix aura donc été divisé par 10 en 5 ans). Pour la petite histoire, la mère de Sergey Brin est atteinte de la maladie de Parkinson, ce qui a incité le milliardaire à réaliser un test génétique en 2006, pour découvrir qu’il était porteur de la mutation du gène LRRK2. Ceci explique son intérêt pour les tests génomiques personnels.
Début 2013, le produit coûte : 99 USD auxquels il faut ajouter 79,95 USD pour les frais de port (ces frais limitent l’accès au vieux continent à 23andMe), la note se monte donc pour un Français à 137,70 € (mi 2011, le prix était de 200 €). Il est facile dorénavant de comprendre que, les volumes des puces Illumina grimpant, la part du coût allouée aux consommables a baissé, mais que la distribution web a ses limites… la poste.
Tests génomiques ?
Le test génétique que propose 23andMe permet :
– d’estimer les facteurs de risques d’être, un jour, atteint d’une maladie à composante majoritairement génétique
– d’estimer vos aptitudes physiques et intellectuelles (en présupposant que la variabilité génétique de ces traits contribue fortement auxdits traits estimés)

– de déterminer son origine géographique, ainsi que, pour le folklore, de quel personnage célèbre dont 23andMe possède le génotype, vous vous rapprochez le plus (sur le ce point lire notre article : A la recherche du chromosome perdu : aux sources des hominidés)
– enfin, 23andMe accumule des génotypages venant alimenter une base de données associée à une population de référence et ainsi accroître l’information génomique disponible pour d’éventuelles recherches (à mener par d’autres…), tout cela financé par les clients de 23andMe… il fallait y penser et oser, c’est chose faite !
Principe ?
Contrairement à un amalgame souvent rencontré, la société californienne n’utilise que très peu le séquençage pour son produit phare. Le principe réside dans l’extraction de l’ADN contenu dans les cellules de la salive, ADN qui fera l’objet d’un génotypage haut-débit permettant de cartographier le génome du client. 23andMe ne fait que compiler les données de GWAS (Genome Wide Association Studies), les études d’association publiées et agglomérées plus ou moins subtilement. Ce type d’études consiste en l’association de profils génétiques à des phénotypes caractérisés. En outre, 23andMe exploite la connaissance des marqueurs en Déséquilibre de Liaison avec une maladie ou une aptitude, le tout en concaténant l’information scientifique publique.
Des témoignages de clients pour en convaincre de nouveaux
Depuis le début 2013, plusieurs témoignages ont été utilisés par la presse pour décrire ce que proposait 23andMe, une publicité à moindre frais. Celui de Slate relate l’histoire de Jill Uchiyama, une personne qui a été adoptée à la naissance et qui à l’âge de 41 ans par l’intermédiaire des services proposés par 23ansMe a retrouvé quelques cousins plus ou moins proches (300 cousins) et en a rencontré un… un manière de recréer de simili-liens de sang. Un autre présent sur le site Quora.com témoigne du cas d’un père découvrant qu’il avait des risques de développer une intolérance au gluten alors que des praticiens ont perdu un temps infini à déterminer l’étiologie de la maladie cœliaque dont souffrait sa fille. Autant de témoignages de nature à convaincre de futurs clients !
Un accueil scientifique mitigé
Un premier article de Jane Kaye « The regulation of direct to consumer genetic tests » (Human Molecular Genetics, 2008), s’inquiète de la perte de contrôle des professionnels de santé, de l’évolution de la législation et de la confidentialité des données transitant par ces nouvelles sociétés de services. L’auteure appelle de ses vœux une régulation de cette « génomique récréative » où l’on peut partager sur les réseaux sociaux son potentiel génétique, ses risques de développer une maladie ou encore rechercher des « cousins » potentiels.
Deux ans après ce premier article, celui de Barbara Prainsack et Howard Wolinsky (Future Medicine, 2010) « Direct-to-consumer genome testing: opportunities for pharmacogenomics research ? » pondère le propos. Dans cet article, les auteurs ne s’inquiètent plus vraiment de l’existence d’un système divulguant des informations génomiques directement au consommateur sans passer par la case du prescripteur médical, mais s’interrogent sur les bénéfices que la recherche scientifique pourrait tirer de ces génotypages payés par les consommateurs eux-mêmes. Entrerait-on dans l’ère du libéralisme génomique ? L’article de Future Medicine développe l’aspect crowdsourcing (en français collaborat ou externalisation ouverte) en s’inquiétant légèrement tout de même d’un problème majeur : le respect de la vie privée…
Une inquiétude majeure
A une époque où des services étatiques développent des projets tels que PRISM, l’outil de surveillance du web utilisé par la NSA ou d’autres que la DGSE utiliserait, le consommateur de la génomique personnalisée pourrait et devrait s’inquiéter de la confidentialité des données génomiques le concernant. En effet, le web est le tuyau par lequel transite toutes les informations génomiques émanant des analyses de ces sociétés de service. La confidentialité des données semble être le nouveau frein au développement de ce type de marché.
Le terme « Nouvelle génération de séquençage à haut-débit » ( ou « Next generation sequencing » ) regroupe l’ensemble des technologies ou plateformes de séquençage développées depuis 2005 par quelques sociétés de biotechnologies.
L’objectif de cet article est de proposer de manière synthétique, un tour d’horizon des différents principes et caractéristiques de ces nouveaux outils et ainsi fournir quelques orientations et solutions techniques en réponses à des questions biologiques.
La position actuelle dans laquelle nous nous trouvons, entre la commercialisation de certains séquenceurs et ceux en cours de développement, est caractéristique d’une période charnière dissociant les technologies à haut débit dites de 2ème génération qui requièrent une étape d’amplification des molécules d’ADN en amont du décodage, de celles dites de 3ème génération permettant le décryptage direct d’une seule molécule d’ADN. Cette dernière catégorie fera à elle seule l’objet d’un prochain article.
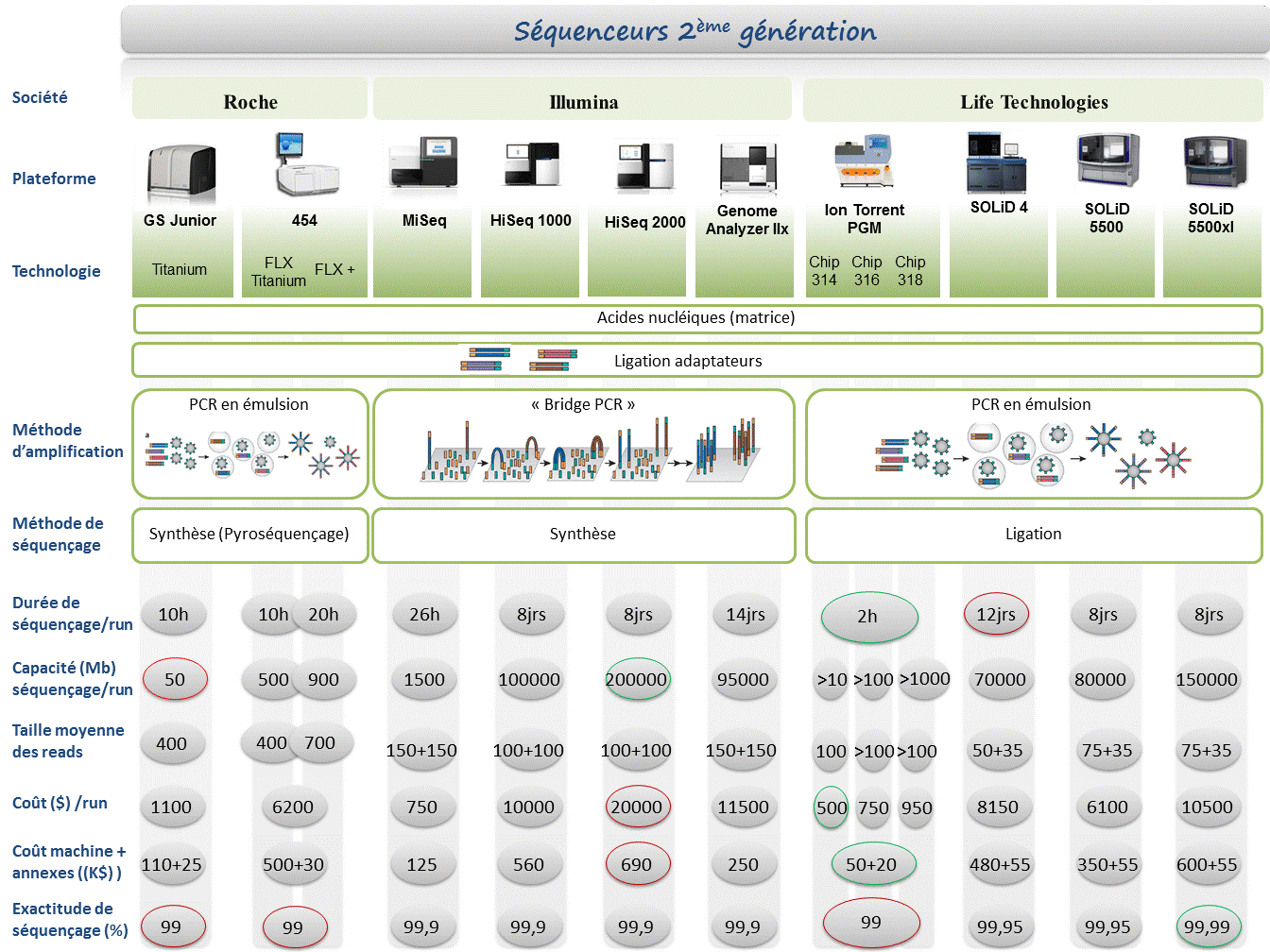
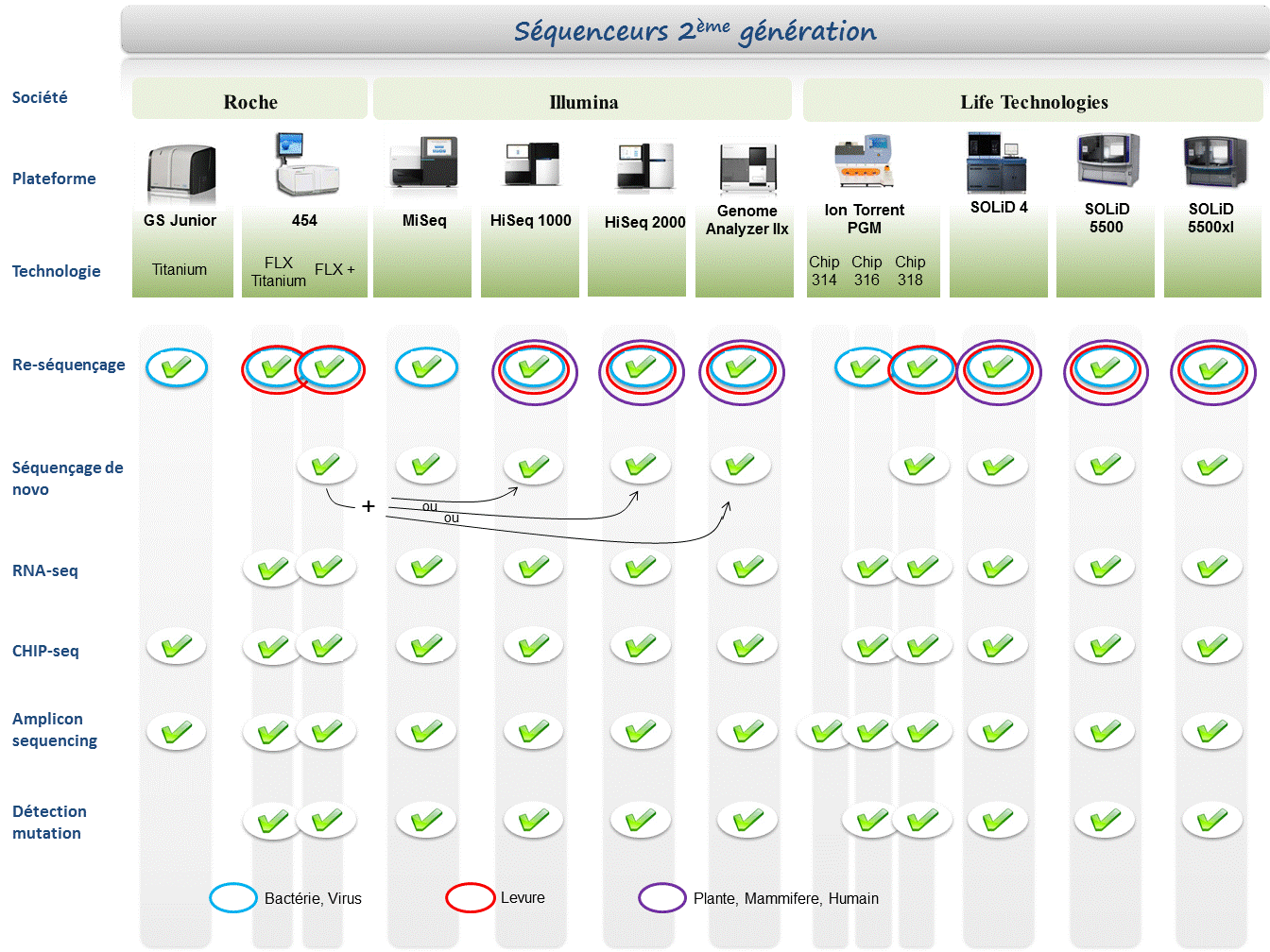
Le marché des séquenceurs de 2éme génération est couvert par 3 grand groupes que sont Roche, Illumina et Life Technologies ayant respectivement proposés de manière successive, leur première plateforme à savoir le 454, le Genome Analyser et enfin le SOLiD. Depuis, le marché s’est étoffé proposant un panel de technologies au principe et caractéristiques propres telles qu’elles sont mentionnées ci dessous. A noter que parmi ce panel, le PGM, Ion torrent est le seul a connaitre une évolution constante en terme de capacités de séquençage (10Mb – reads de 100b – Juin 2011 / 100Mb – reads de 100b – Sept 2011 / 100Mb – reads 200b – Nov 2011 / 1Gb – Jan 2012 )
Chaque plateforme possède ses avantages et inconvénients et nombreuses sont celles configurées pour répondre à de nombreuses approches « omics », dans certaines limites. Il s’agira de faire un choix technologique selon les champs d’applications souhaités.
De manière générale, le type d’organisme étudié prédéterminera la technologie à employer. La notion de profondeur est récurrente à chaque application et dans l’objectif d’un reséquençage, le choix de la plateforme peut être identifié, de manière simplifiée, sur la base d’un calcul rapide ( P = N / L où P: Profondeur, N: Nombre des nucléotides totaux des reads, L: Taille du génome étudié).
Concernant les séquenceurs de 2ème génération, le séquençage de novo est une application mentionnée chez de nombreux fournisseurs (cf le tableau ci-dessous). Toutefois, l’association de deux technologies générant à la fois des reads longs (type 454, Roche) et une profondeur conséquente (type GAIIx, Illumina) palliant aux problèmes liés aux homopolymères et erreurs de séquençage, est préconisée (Au cours de l’article à venir sur les séquenceurs de 3ème génération, nous aborderons les plateformes davantage configurées pour cette application).
Ce paramètre de profondeur sera également à prendre en considération pour les champs d’applications incluant la notion d’analyse quantitative (RNAseq, ChIPseq, …). Si la profondeur permet d’atténuer les erreurs de séquençage, il reste néanmoins préférable de s’orienter vers des technologies à Q30 minimum (1 erreur sur 1000) pour la détection de SNPs.
Selon les technologies évoquées ci-dessus, les caractéristiques et champs d’applications ont évolués. Aussi, je vous propose de retrouver l’ensemble de ces informations actualisées en cliquant sur ce lien.
L’ensemble des informations sont détaillées dans l’article mentionné ci-dessous:
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages