Le séquençage du génome humain pour 30$, c’est la promesse faite par David Weitz, co-fondateur de GnuBio au cours de l’année 2010. Trois ans plus tard, la start-up vient de lancer en béta-test son nouveau procédé de séquençage à haut débit. Il s’agit du premier système entièrement intégré (amplification des cibles, enrichissement, séquençage et analyse) qui propose pour le moment une application de target-sequencing destinée aux cliniciens et dédiée au diagnostic moléculaire.
Le séquençage du génome humain pour 30$, c’est la promesse faite par David Weitz, co-fondateur de GnuBio au cours de l’année 2010. Trois ans plus tard, la start-up vient de lancer en béta-test son nouveau procédé de séquençage à haut débit. Il s’agit du premier système entièrement intégré (amplification des cibles, enrichissement, séquençage et analyse) qui propose pour le moment une application de target-sequencing destinée aux cliniciens et dédiée au diagnostic moléculaire.
En 2010, David Weitz et son équipe de l’Université d’Harvard ambitionnent de développer une nouvelle technologie de séquençage à haut-débit, alliant les technologies de biologie moléculaire aux procédés de microfluidique développés quelques années plus tôt (2004) au sein de la société RainDance technologies.
Cette nouvelle approche repose sur la capacité à générer des gouttes de l’ordre du picolitre et pouvant être déplacée sur une puce microfluidique. Ces gouttes peuvent renfermer soit un couple d’amorces, des adaptateurs, ou tout autre type de réactifs nécessaires aux étapes de préparation de librairie et de séquençage (séquençage par hybridation-ligation, type SOLiD avec une fidélité de 99.99%). Dès lors, leurs quantités utilisées au sein de ces picogouttes sont considérablement revues à la baisse, ce qui constitue le point clé à une réduction des coûts de séquençage et donc la perspective d’un séquençage de génome humain à 30$, selon David Weitz.
Les projets de GnuBio sont désormais d’élargir le champs d’applications de leur séquenceur à l’analyse transcriptomique (RNA-seq), l’étude de la méthylation (ChiP-seq) ou encore le séquençage de génome entier. La société ambitionne une commercialisation de leur équipement au cours de l’année 2014.
A suivre…
 Noblegen Biosciences, start-up localisée dans le Massachusetts, ambitionne de commercialiser pour 2014 « optipore » (pour « optical detection » et « nanopore ») , un séquenceur de paillasse de troisième génération combinant nanotechnologies et un système de lecture optique permettant de réduire drastiquement les coûts de séquençage. L’objectif est de conquérir le marché des laboratoires cliniques dans la perspective de l’émergence d’une médecine personnalisée et de proposer ainsi un séquençage de génome humain complet à faible coût et dans un temps record. Dans cette course engagée, la société américaine se trouve en bonne position.
Noblegen Biosciences, start-up localisée dans le Massachusetts, ambitionne de commercialiser pour 2014 « optipore » (pour « optical detection » et « nanopore ») , un séquenceur de paillasse de troisième génération combinant nanotechnologies et un système de lecture optique permettant de réduire drastiquement les coûts de séquençage. L’objectif est de conquérir le marché des laboratoires cliniques dans la perspective de l’émergence d’une médecine personnalisée et de proposer ainsi un séquençage de génome humain complet à faible coût et dans un temps record. Dans cette course engagée, la société américaine se trouve en bonne position.
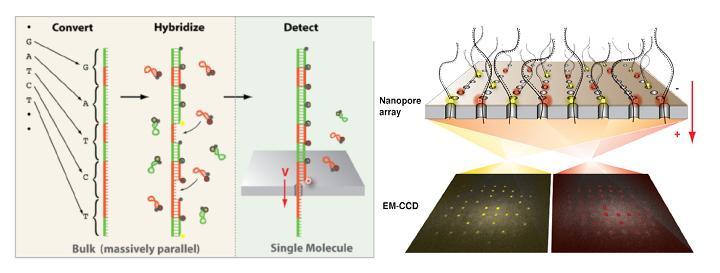
A l’instar du PGM Ion torrent, une puce en silicium constitue le coeur de la machine et renferme des centaines de nanopores.  La molécule d’ADN unique native destinée à être séquencée est tout d’abord convertie en une nouvelle molécule synthétique transformant chaque base par une séquence nucléotidique spécifique correspondante. A chacune des quatre signatures nucléotidiques correspond un « molecular beacon » complémentaire fluorescent initialement inactivé par un système de « quencher ». Leurs hybridations au brin néoformé aboutit à un ADN double brin. la molécule est dirigé vers les nanopores par des échanges ioniques et en raison de la taille des orifices, les « molecular beacon » sont contraints à se deshybrider libérant cette fois une fluorescence, lue par un capteur photographique de type CMOS, qui est traduite en séquence.
La molécule d’ADN unique native destinée à être séquencée est tout d’abord convertie en une nouvelle molécule synthétique transformant chaque base par une séquence nucléotidique spécifique correspondante. A chacune des quatre signatures nucléotidiques correspond un « molecular beacon » complémentaire fluorescent initialement inactivé par un système de « quencher ». Leurs hybridations au brin néoformé aboutit à un ADN double brin. la molécule est dirigé vers les nanopores par des échanges ioniques et en raison de la taille des orifices, les « molecular beacon » sont contraints à se deshybrider libérant cette fois une fluorescence, lue par un capteur photographique de type CMOS, qui est traduite en séquence.
L’ensemble des études et preuves de faisabilité sur lesquelles repose la technologie de séquençage « optipore » sont décrites et détaillées au travers de l’article libre d’accès ci dessous: A ce jour, peu de caractéristiques techniques liées à cette plateforme filtrent mais Frank Feist, cofondateur de la société, annonce une capacité de séquençage de 500 Gb/heure. De plus, en raison de l’étape de conversion initiale, la taille des fragments serait limitée à 200 bases.
A ce jour, peu de caractéristiques techniques liées à cette plateforme filtrent mais Frank Feist, cofondateur de la société, annonce une capacité de séquençage de 500 Gb/heure. De plus, en raison de l’étape de conversion initiale, la taille des fragments serait limitée à 200 bases.
Un soutien de 4,2 millions de dollars en septembre dernier de la part du National Human Genome Research Institute devrait permettre à la société américaine de conforter cette avancée prometteuse.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages