Une jeune start-up est née en mai 2012 : cette société issue du CNRS est porteuse d’une innovation dans le secteur du séquençage haut-débit. Enfin, une alternative française aux anglosaxons qui sont présents sur le marché depuis une dizaine d’année ! Maintenant… espérons que ce nouveau né n’arrive pas trop tard sur un marché animé par des fournisseurs de séquenceurs de 2ème génération (un marché mature) et d’autres fournissant des solutions de 3ème génération, riches de promesses.
Une jeune start-up est née en mai 2012 : cette société issue du CNRS est porteuse d’une innovation dans le secteur du séquençage haut-débit. Enfin, une alternative française aux anglosaxons qui sont présents sur le marché depuis une dizaine d’année ! Maintenant… espérons que ce nouveau né n’arrive pas trop tard sur un marché animé par des fournisseurs de séquenceurs de 2ème génération (un marché mature) et d’autres fournissant des solutions de 3ème génération, riches de promesses.
PicoSeq derrière ce nom emprunt d’humilité se cache une technologie de séquençage des plus ingénieuses : en effet, SIMDEQ™ (SIngle-molecule Magnetic DEtection and Quantification) la technologie de PicoSeq utilise une approche biophysique pour extraire des informations à partir de la séquence d’ADN ou d’ARN.
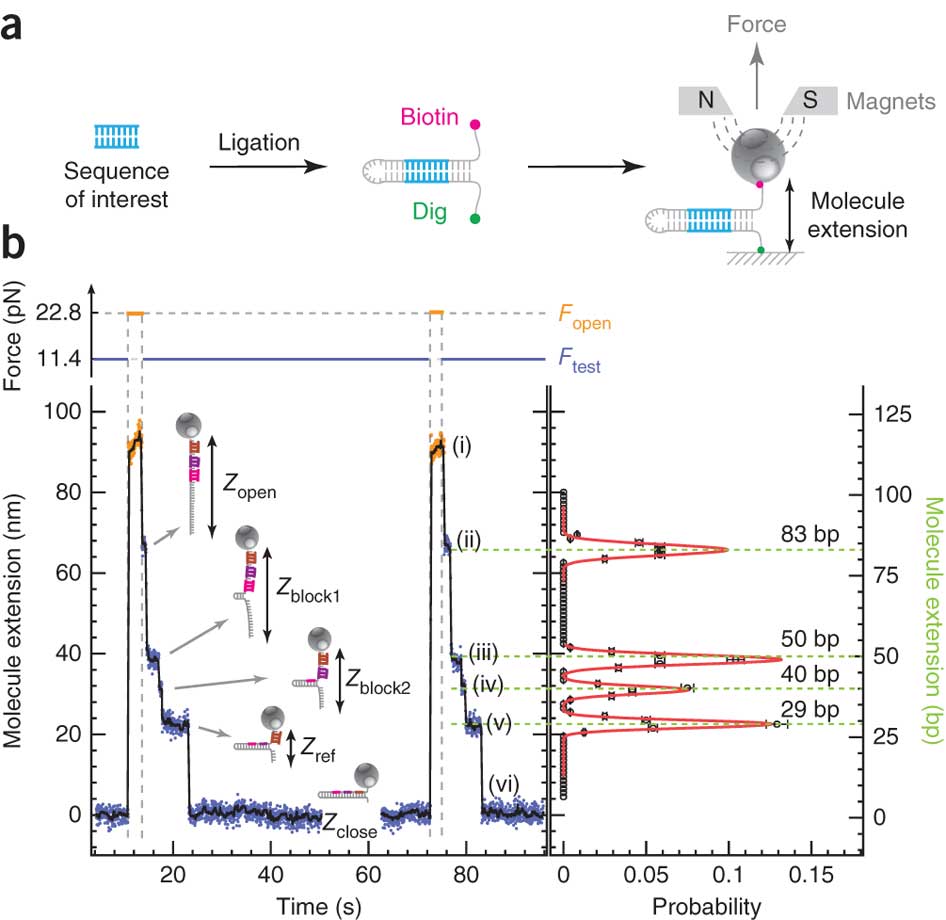
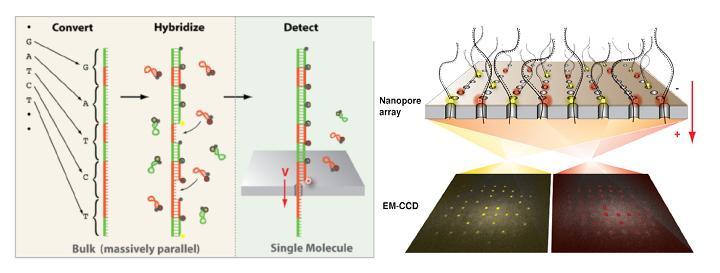
En s’appuyant sur cette représentation schématique tirée de Ding et al. (Nature Methods, 2012), on y voit un peu plus clair. Des fragments d’ADN ou d’ARN que l’on souhaite analyser servent de matrice pour la réalisation d’une librairie en «épingle à cheveux». Pour chaque épingle à cheveux, un côté d’un brin d’acide nucléique est attaché sur une surface solide plane et l’autre à une bille magnétique. En plaçant les billes dans un champ magnétique, modulant celui-ci de manière cyclique, les épingles à cheveux peuvent être auto-hybridées ou non (zip ou unzip). Ce processus peut être effectué des milliers de fois sans endommager les molécules constitutives de la librairie. La position de chaque bille est suivie à très haute précision permettant de voir ce processus d’ouverture et de fermeture en temps réel: nous avons donc là un signal brut permettant, en fonction de la force appliquée pour ouvrir totalement l’épingle à cheveu et des séquences d’oligonucléotides séquentiellement introduites dans le système de modifier la distance bille-support et de jouer sur le temps nécessaire où la force s’applique pour ouvrir l’épingle à cheveu…
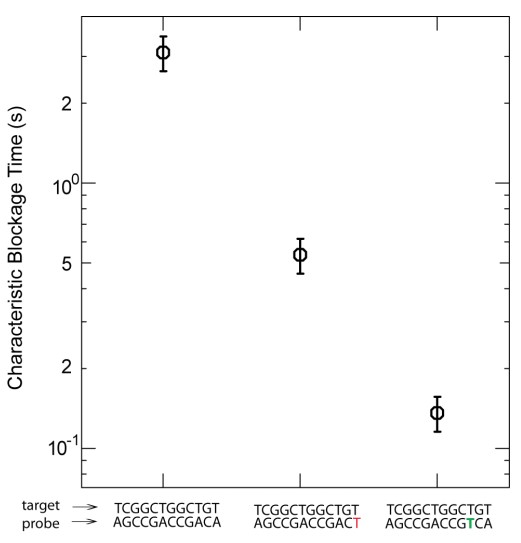
La figure ci-dessus (présente dans les données supplémentaires de l’article sus-cité) permet d’appréhender le potentiel de discrimination de la méthode… où le temps de blocage est fonction du nombre de mésappariements et de la position de ces mésappariements…
Finalement si l’aspect technique est intéressant puisqu’en rupture avec les méthodes proposées par PacBio certainement un peu moins avec le système proposé par Oxford Nanopore Technologies, si la perspective annoncée par PicoSeq est réellement séduisante: l’accès modifications épigénétiques de l’ADN, la question centrale est de savoir si le pas de la commercialisation (dans des conditions propices au succès) d’un tel outil, sera franchi.
Un article d’ Atlantico de septembre 2015, titré : les trois raisons pour lesquelles la France est incapable de rivaliser avec les géants américains de l’analyse ADN, est assez éclairant pour imaginer comment la concrétisation d’une preuve de concept peut être un chemin ubuescokafkaïen. Pour illustrer cela les propos de Gordon Hamilton, le directeur de la startup PicoSeq qui s’inquiète sur les entraves « typically french » peuvent faire office de témoignage. Ce dernier s’inquiète : « La qualité de la recherche scientifique (en France) est incroyable, l’une des meilleures » « le seul souci, c’est que l’on a beaucoup de difficultés ici à transformer ces recherches en vrai business » pour finir par citer en exemple les lenteurs administratives spécificités latines : « nous avons mis presque deux ans pour négocier les licences nécessaires aux brevets de Picoseq. Le même processus en Californie prend entre deux semaines et deux mois. Sur un marché aussi rapide que celui-ci, deux ans c’est très long. Tout change vite, c’est donc impossible pour nous d’être de sérieux concurrents de ces sociétés américaines qui ont toujours un temps d’avance ».
 Noblegen Biosciences, start-up localisée dans le Massachusetts, ambitionne de commercialiser pour 2014 « optipore » (pour « optical detection » et « nanopore ») , un séquenceur de paillasse de troisième génération combinant nanotechnologies et un système de lecture optique permettant de réduire drastiquement les coûts de séquençage. L’objectif est de conquérir le marché des laboratoires cliniques dans la perspective de l’émergence d’une médecine personnalisée et de proposer ainsi un séquençage de génome humain complet à faible coût et dans un temps record. Dans cette course engagée, la société américaine se trouve en bonne position.
Noblegen Biosciences, start-up localisée dans le Massachusetts, ambitionne de commercialiser pour 2014 « optipore » (pour « optical detection » et « nanopore ») , un séquenceur de paillasse de troisième génération combinant nanotechnologies et un système de lecture optique permettant de réduire drastiquement les coûts de séquençage. L’objectif est de conquérir le marché des laboratoires cliniques dans la perspective de l’émergence d’une médecine personnalisée et de proposer ainsi un séquençage de génome humain complet à faible coût et dans un temps record. Dans cette course engagée, la société américaine se trouve en bonne position.
A l’instar du PGM Ion torrent, une puce en silicium constitue le coeur de la machine et renferme des centaines de nanopores. 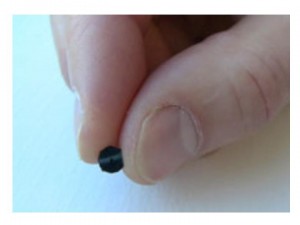 La molécule d’ADN unique native destinée à être séquencée est tout d’abord convertie en une nouvelle molécule synthétique transformant chaque base par une séquence nucléotidique spécifique correspondante. A chacune des quatre signatures nucléotidiques correspond un « molecular beacon » complémentaire fluorescent initialement inactivé par un système de « quencher ». Leurs hybridations au brin néoformé aboutit à un ADN double brin. la molécule est dirigé vers les nanopores par des échanges ioniques et en raison de la taille des orifices, les « molecular beacon » sont contraints à se deshybrider libérant cette fois une fluorescence, lue par un capteur photographique de type CMOS, qui est traduite en séquence.
La molécule d’ADN unique native destinée à être séquencée est tout d’abord convertie en une nouvelle molécule synthétique transformant chaque base par une séquence nucléotidique spécifique correspondante. A chacune des quatre signatures nucléotidiques correspond un « molecular beacon » complémentaire fluorescent initialement inactivé par un système de « quencher ». Leurs hybridations au brin néoformé aboutit à un ADN double brin. la molécule est dirigé vers les nanopores par des échanges ioniques et en raison de la taille des orifices, les « molecular beacon » sont contraints à se deshybrider libérant cette fois une fluorescence, lue par un capteur photographique de type CMOS, qui est traduite en séquence.
L’ensemble des études et preuves de faisabilité sur lesquelles repose la technologie de séquençage « optipore » sont décrites et détaillées au travers de l’article libre d’accès ci dessous: A ce jour, peu de caractéristiques techniques liées à cette plateforme filtrent mais Frank Feist, cofondateur de la société, annonce une capacité de séquençage de 500 Gb/heure. De plus, en raison de l’étape de conversion initiale, la taille des fragments serait limitée à 200 bases.
A ce jour, peu de caractéristiques techniques liées à cette plateforme filtrent mais Frank Feist, cofondateur de la société, annonce une capacité de séquençage de 500 Gb/heure. De plus, en raison de l’étape de conversion initiale, la taille des fragments serait limitée à 200 bases.
Un soutien de 4,2 millions de dollars en septembre dernier de la part du National Human Genome Research Institute devrait permettre à la société américaine de conforter cette avancée prometteuse.
 L’AGBT qui a eu lieu du 15 au 18 février, à Marco Island, a fait la part belle à la technologie d’Oxford Nanopore, ainsi qu’il avait été prévu. La société dont il est question a profité du rassemblement pour lever le voile sur 2 produits : le MinION et le GridION, il est à noter que le MinION, mini-système de séquençage de la taille d’une grosse clé USB (photo ci-contre) a une dénomination commerciale tout particulièrement adaptée au marché français.
L’AGBT qui a eu lieu du 15 au 18 février, à Marco Island, a fait la part belle à la technologie d’Oxford Nanopore, ainsi qu’il avait été prévu. La société dont il est question a profité du rassemblement pour lever le voile sur 2 produits : le MinION et le GridION, il est à noter que le MinION, mini-système de séquençage de la taille d’une grosse clé USB (photo ci-contre) a une dénomination commerciale tout particulièrement adaptée au marché français.
La technologie d‘Oxford Nanopore a été évoquée dans plusieurs de nos articles. Elle permet le séquençage et l’analyse à haut-débit de reads de taille ultra longue (plusieurs kb) en temps réel pour pas très cher : la promesse d’un séquençage de 3ème génération démocratisé. Clive G. Brown (directeur de la technologie chez Oxford Nanopore) a présenté ses deux nouveaux jouets :
– Le MinION (dont le prix serait inférieur à 900 $) est un consommable et séquenceur (les deux à la fois) jetable qui devrait permettre de générer 1 Gb de données
– Le GridION (que vous pouvez empiler à foison, voire photo ci-dessous) permet quant à lui de générer, par module, plusieurs dizaines de Gb / jour (on pencherait pour un minimum de 25 Gb) sachant que selon nos informations un module aurait un coût voisin de 30 k$. Oxford Nanopore insiste sur le fait qu’à la Gb générée ils seront concurrentiels en terme de coût des consommables. En outre, le volume de données générées s’adapte à la problématique de l’utilisateur puisqu’en effet tant que l’appareil séquence -d’où leur slogan « Run Until« – il génère des données (le débit journalier associé à une technologie prend tout son sens ici). La cartouche –consommable de séquençage– associée à la technologie GridION possède actuellement 2000 pores individuelles -en 2013, il est prévu de passer à un consommable en comportant 8000- avec cette évolution il sera donc possible avec 20 modules GridION (environ l’investissement équivalent à un HiSeq2000) de séquencer un génome humain en 15 minutes ! Une autre façon de voir les chose est la suivante, dans sa version « actuelle » à 2000 pores disponibles : pour un prix équivalent à celui d’une configuration de type Ion Proton, 5 modules GridION seront capables de séquencer un génome humain à 30 X (cela leur prendrait une demie journée).
D’autres éléments ont filtré lors de l’AGBT. En effet, il semblerait que la technologie d’Oxford Nanopore subisse un taux d’erreurs sur séquences brutes encore assez élevé de 4 % (comparé aux plus de 10 % pour la technologie de Pacific Bioscience). Clive G. Brown aurait laissé entendre que ce taux d’erreurs serait uniforme et le fruit d’une majorité d’erreurs systématiques (ce qui est plutôt bon signe, en vue d’une rapide amélioration du système). Au niveau du système de détection, une puce GridION comporte 2K capteurs (un par pore). Chaque capteur permet de distinguer 64 signaux différents, ceci a permis d’analyser le passage de triplets de base afin de pouvoir discriminer 4x4x4 profils différents (j’avoue que j’attendais plus : quid de la prise en compte d’un signal différentiel en cas de présence d’une 5-méthylcytosine ?).
Au niveau préparation des échantillons, un séquenceur de 3ème génération ne nécessite pas de phase d’amplification. Hormis une phase préalable de légère fragmentation de l’échantillon rien ne semble envisagé. Pour palier leur problème de fiabilité, on imagine aisément qu’à l’instar de Pacific Bioscience, une circularisation de l’ADN de l’échantillon permettra d’engendrer en séquençage un nombre suffisant de répétitions venant atténuer ce point négatif.
Un élément important -mais pas surprenant- réside dans la politique commerciale affichée : une distribution directe des machines ainsi qu’une adaptation tarifaire (en usant de forfaits) offrant la possibilité d’acheter la machine à prix réduit avec un report sur le coût des consommables devrait permettre à Oxford Nanopore de conquérir quelques marchés n’en doutons pas !
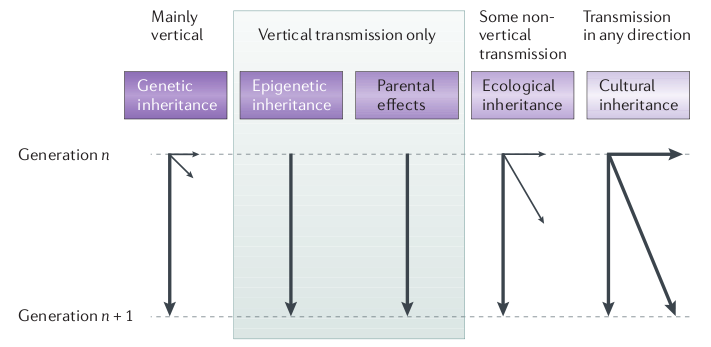
Pierre Barthélémy, journaliste scientifique au monde, a publié hier sur son blog (passeurdesciences.blog.lemonde.fr) une interview de Etienne Danchin (directeur de recherche CNRS et directeur de l’EDB) qui nous parle de la nécessité de s’extraire du dogme du « tout-génétique » pour s’intéresser plus précisément à d’autres mécanismes qui régissent l’hérédité, tel que l’épigénétique, l’hérédité culturelle et environnementale.
Vous pouvez retrouver l’interview dans son intégralité ici :
Cette interview fait suite à la publication d’une synthèse sur l’hérédité non génétique et son rôle dans l’évolution parue dans Nature Reviews Genetics en Juillet 2011 que vous pouvez consulter en intégralité ici.
Etienne Danchin n’est pas le seul à s’interroger sur la part réelle de la génétique dans les mécanismes de l’hérédité, nous vous conseillons une nouvelle fois l’excellente émission de Jean Claude Ameisen (à écouter dans ce post) qui aborde ces aspects de la transmission génétique et non génétique, entre générations.
L’hérédité culturelle est une composante en général peu discutée (notamment dans le domaine animal): quelle est la part de variation phénotypique qui peut être expliquée par une transmission sociale (ou transmission par apprentissage)? A noter que le rôle de la composante culturelle dans l’hérédité a été formalisé dans les années 1970, ça ne date donc pas d’hier, mais comme toutes les composantes non génétiques impliquées dans l’hérédité elle s’est retrouvée minimisée, voire ignorée, au profit de la composante génétique.
Quoi qu’il en soit, à l’heure de l’arrivée de la 3eme génération de séquenceur haut-débit et de la génération toujours plus astronomique de données génomiques, il est toujours bon de rappeler que le seul transfert de gènes n’explique qu’une proportion des mécanismes étudiés.
Pierre Barthélémy, journaliste scientifique à publié hier sur son blog (passeurdesciences.blog.lemonde.fr) une interview de Etienne Danchin (directeur de recherche CNRS) qui nous parle de la nécéssité de sortir du dogme du « tout-génétique » pour s’intérésser plus précisement à d’autre mécanisme qui régisse l’hériédité, l’épigénétique, l’hérédité génétique et environementale.
Vous pouvez retrouver l’interview dans son intégralité ici :
Cet interview est le résultats d’une synthèse sur l’hérédité non génétique et son rôle dans l’évolution publié dans Nature Reviews Genetics en Juillet 2011 que vous pouvez consulter en intégralité ici.
Etienne Danchin n’est pas le seul à s’intéroger sur la place réelle de la génétique dans les mécanisme de l’hérédité, nous vous conseillons une nouvelle fois l’excellente émission de Jean Claude Ameisen (à écouter dans ce post).
A Davos en Suisse (dans la patrie de Roche) au forum économique mondial, Jonathan Rothberg présente son séquenceur Ion Proton, une évolution de son PGM de Life Technologies. La dépêche AFP (mise en forme par 20minutes est disponible en cliquant sur l’image ci-dessous) est dithyrambique replaçant la machine de Life Technologies dans le contexte et le champ d’application de la médecine personnalisée… jouant au passage du violon de l’affect. Le Ion Proton, fort de la démonstration menée par son petit frère le PGM depuis un an (200 machines PGM en Europe !) s’appuie sur l’efficience du séquençage à l’aide de semi-conducteurs.
Il parait évident que la médecine personnalisée aura un futur au sein des pays développés… mais il ne faut pas oublier qu’il s’agit d’un futur que l’on augurait aux puces à ADN, il y a plus de 10 ans, sans réelle démocratisation au sein des cliniques. Avant toute chose, permettre de séquencer l’équivalent d’un génome humain ou de 2 exomes humains pour la somme soutenable de 1000 $ (en 2 heures) laisse entrevoir une accélération des recherches biomédicales mais aussi de toutes les autres ! Cet outil qui torpille un MiSeq (Illumina) sorti trop tard, diffusera dans les laboratoires… il remporte, malgré ses défauts d’ores et déjà connus, la bataille du séquençage 2ème génération. Le prochain pari (plus aléatoire) porterait plutôt sur le séquençage 3ème génération dont la promesse est une lecture d’une molécule d’ADN ou d’ARN (non amplifiée artificiellement) dans son « état » originel (méthylé ou non par exemple). La 3ème génération unifie génomique et épigénétique.
Le Ion Proton devrait être livrable à partir d’octobre 2012, quelques chanceux pourront malgré tout réaliser des runs dès le milieu de l’année (il faut en acheter un minimum de 4 pour être un parmi ces privilégiés).
Un Ion Proton devrait avoir un coût proche de 140 k€ auxquels il vous faudra ajouter 65 k€ de serveur informatique.
 Depuis la publication de 2006, d’Astier and al., J. Am. Chem. Soc., la communauté scientifique attendait la révolution du séquençage 3ème génération que constituait la géniale idée d’Oxford Nanopore : à savoir l’alliance des nanotechnologies, du génie génétique le tout à des fins de séquençage (et plus si affinité). Ainsi, un pore d’alpha-hémolysine (d’environ 1 nanomètre) ou la porine A de Mycobacterium smegmatis ou bien encore du graphène ont été investigués pour devenir le pore idéal dans lequel, après clivage par une exonucléase des liaisons phosphodiester, les bases azotées dont l’enchaînement constitue le séquençage passe afin d’être analysées (par analysées on entend lues, la lecture étant basée sur la différence de conductance électrique liée à la nature chimique des bases azotées). Le pari, pour Oxford Nanopore réside dans le fait de trouver un mode de détection suffisamment sensible pour analyser puis par puis un signal généré par le passage d’une base au travers de pores (autrement appelés trous). La résolution de la technologie constituait un défit, sa faiblesse provenait (espérons que ce verbe se conjugue aujourd’hui au passé) de la rapidité de passage des bases à travers le pore (1 à 5 µs / base)…
Depuis la publication de 2006, d’Astier and al., J. Am. Chem. Soc., la communauté scientifique attendait la révolution du séquençage 3ème génération que constituait la géniale idée d’Oxford Nanopore : à savoir l’alliance des nanotechnologies, du génie génétique le tout à des fins de séquençage (et plus si affinité). Ainsi, un pore d’alpha-hémolysine (d’environ 1 nanomètre) ou la porine A de Mycobacterium smegmatis ou bien encore du graphène ont été investigués pour devenir le pore idéal dans lequel, après clivage par une exonucléase des liaisons phosphodiester, les bases azotées dont l’enchaînement constitue le séquençage passe afin d’être analysées (par analysées on entend lues, la lecture étant basée sur la différence de conductance électrique liée à la nature chimique des bases azotées). Le pari, pour Oxford Nanopore réside dans le fait de trouver un mode de détection suffisamment sensible pour analyser puis par puis un signal généré par le passage d’une base au travers de pores (autrement appelés trous). La résolution de la technologie constituait un défit, sa faiblesse provenait (espérons que ce verbe se conjugue aujourd’hui au passé) de la rapidité de passage des bases à travers le pore (1 à 5 µs / base)…
Une attente à émouvoir un James Dewey Watson, plusieurs publications en guise de démonstration de la faisabilité d’un tel séquençage, des appels de fonds ayant abouti à l’intégration d’Illumina au niveau du capital d’Oxford Nanopore (investissement en deux fois d’abord de 28 millions $ puis de 41 millions) ont maintenu en haleine les scientifiques avides de séquençage 3ème génération.
Oxford Nanopore est l’un des seuls acteurs européens, dans le monde du séquençage haut-débit où l’hégémonie californienne est écrasante… Alors que les Californiens (PacBio, Life, Illumina) communiquent à grand renfort d’annonces choc, Oxford Nanopore garde une part de mystère (aucune spécification réellement disponible à ce jour), nourrissant notre curiosité avec quelques publications scientifiques et quelques animations dont certaines vous sont présentées ci-dessous.
http://vimeo.com/18630569
Cette première animation reprend les différentes modalités de séquençage
http://vimeo.com/20289048
Avec la vidéo ci-dessus et celle ci-après, Oxford Nanopore réinvente le magnétoscope VHS à visée séquençage haut-débit…
http://vimeo.com/19288315
Finalement, le message à peine subliminal de ce court article : Oxford Nanopore est une société à surveiller de près… de plus en plus près.
Note : si les liens vers les vidéos ne fonctionnent pas essayer toujours en suivant celui-ci…
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages