 L’AGBT qui a eu lieu du 15 au 18 février, à Marco Island, a fait la part belle à la technologie d’Oxford Nanopore, ainsi qu’il avait été prévu. La société dont il est question a profité du rassemblement pour lever le voile sur 2 produits : le MinION et le GridION, il est à noter que le MinION, mini-système de séquençage de la taille d’une grosse clé USB (photo ci-contre) a une dénomination commerciale tout particulièrement adaptée au marché français.
L’AGBT qui a eu lieu du 15 au 18 février, à Marco Island, a fait la part belle à la technologie d’Oxford Nanopore, ainsi qu’il avait été prévu. La société dont il est question a profité du rassemblement pour lever le voile sur 2 produits : le MinION et le GridION, il est à noter que le MinION, mini-système de séquençage de la taille d’une grosse clé USB (photo ci-contre) a une dénomination commerciale tout particulièrement adaptée au marché français.
La technologie d‘Oxford Nanopore a été évoquée dans plusieurs de nos articles. Elle permet le séquençage et l’analyse à haut-débit de reads de taille ultra longue (plusieurs kb) en temps réel pour pas très cher : la promesse d’un séquençage de 3ème génération démocratisé. Clive G. Brown (directeur de la technologie chez Oxford Nanopore) a présenté ses deux nouveaux jouets :
– Le MinION (dont le prix serait inférieur à 900 $) est un consommable et séquenceur (les deux à la fois) jetable qui devrait permettre de générer 1 Gb de données
– Le GridION (que vous pouvez empiler à foison, voire photo ci-dessous) permet quant à lui de générer, par module, plusieurs dizaines de Gb / jour (on pencherait pour un minimum de 25 Gb) sachant que selon nos informations un module aurait un coût voisin de 30 k$. Oxford Nanopore insiste sur le fait qu’à la Gb générée ils seront concurrentiels en terme de coût des consommables. En outre, le volume de données générées s’adapte à la problématique de l’utilisateur puisqu’en effet tant que l’appareil séquence -d’où leur slogan « Run Until« – il génère des données (le débit journalier associé à une technologie prend tout son sens ici). La cartouche –consommable de séquençage– associée à la technologie GridION possède actuellement 2000 pores individuelles -en 2013, il est prévu de passer à un consommable en comportant 8000- avec cette évolution il sera donc possible avec 20 modules GridION (environ l’investissement équivalent à un HiSeq2000) de séquencer un génome humain en 15 minutes ! Une autre façon de voir les chose est la suivante, dans sa version « actuelle » à 2000 pores disponibles : pour un prix équivalent à celui d’une configuration de type Ion Proton, 5 modules GridION seront capables de séquencer un génome humain à 30 X (cela leur prendrait une demie journée).
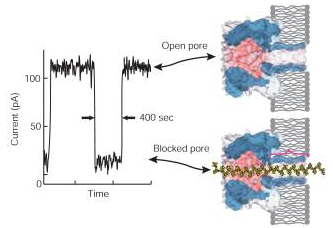
D’autres éléments ont filtré lors de l’AGBT. En effet, il semblerait que la technologie d’Oxford Nanopore subisse un taux d’erreurs sur séquences brutes encore assez élevé de 4 % (comparé aux plus de 10 % pour la technologie de Pacific Bioscience). Clive G. Brown aurait laissé entendre que ce taux d’erreurs serait uniforme et le fruit d’une majorité d’erreurs systématiques (ce qui est plutôt bon signe, en vue d’une rapide amélioration du système). Au niveau du système de détection, une puce GridION comporte 2K capteurs (un par pore). Chaque capteur permet de distinguer 64 signaux différents, ceci a permis d’analyser le passage de triplets de base afin de pouvoir discriminer 4x4x4 profils différents (j’avoue que j’attendais plus : quid de la prise en compte d’un signal différentiel en cas de présence d’une 5-méthylcytosine ?).
Au niveau préparation des échantillons, un séquenceur de 3ème génération ne nécessite pas de phase d’amplification. Hormis une phase préalable de légère fragmentation de l’échantillon rien ne semble envisagé. Pour palier leur problème de fiabilité, on imagine aisément qu’à l’instar de Pacific Bioscience, une circularisation de l’ADN de l’échantillon permettra d’engendrer en séquençage un nombre suffisant de répétitions venant atténuer ce point négatif.
Un élément important -mais pas surprenant- réside dans la politique commerciale affichée : une distribution directe des machines ainsi qu’une adaptation tarifaire (en usant de forfaits) offrant la possibilité d’acheter la machine à prix réduit avec un report sur le coût des consommables devrait permettre à Oxford Nanopore de conquérir quelques marchés n’en doutons pas !
 Le représentant européen des séquenceurs haut-débit de troisième génération, Oxford Nanopore va communiquer à l’AGBT… dans quelques jours. La société en profitera pour annoncer la commercialisation de ses premières machines permettant de séquencer en 24 heures un génome humain pour moins de 1000 $ (soit le même prix qu’un génome humain séquencé par un Ion Proton en 2 heures sur une plateforme de deuxième génération).
Le représentant européen des séquenceurs haut-débit de troisième génération, Oxford Nanopore va communiquer à l’AGBT… dans quelques jours. La société en profitera pour annoncer la commercialisation de ses premières machines permettant de séquencer en 24 heures un génome humain pour moins de 1000 $ (soit le même prix qu’un génome humain séquencé par un Ion Proton en 2 heures sur une plateforme de deuxième génération).
Oxford Nanopore souhaite commercialiser sa machine (en direct) avant la fin 2012. Un réelle course a lieu, puisqu’à cette échéance sera commercialisé le Ion Proton. Nous assisterons donc avant fin 2012 a un chevauchement technologique où 2ème et 3ème générations de séquenceurs risquent de se télescoper… les scientifiques en cours d’élaboration de dossiers de demandes de financements devront (vite) choisir leur camp… : partir sur une technologie qui a d’ores-et-déjà fait ses preuves (Ion Proton) donc de 2ème génération ou une technologie de 3ème génération totalement innovante… mais dont on ne possède pas de retour indépendant sur la qualité des données générées. La « guerre » technologique est lancée entre le transfuge de Solexa (technologie des séquenceurs Illumina), John Milton, le responsable scientifique d’Oxford Nanopore et la biotech’ rock-star Jonathan Rothberg, responsable scientifique de Ion Torrent chez Life Technologies.
 Chaque année depuis 13 ans, l’AGBT (Advances in Genome Biology and Technology) rassemble les fournisseurs de technologies de pointe, quelques « maîtres » en la matière venant dispenser des cours du soir (sorte de « master-class ») et beaucoup de scientifiques essentiellement Nord-Américains. En effet, cette grande messe de la biologie haut-débit a lieu à Marco Island, île de Floride, lieu de villégiature pour retraités liftés percevant leurs fonds de pension sous forme de rente viagère.
Chaque année depuis 13 ans, l’AGBT (Advances in Genome Biology and Technology) rassemble les fournisseurs de technologies de pointe, quelques « maîtres » en la matière venant dispenser des cours du soir (sorte de « master-class ») et beaucoup de scientifiques essentiellement Nord-Américains. En effet, cette grande messe de la biologie haut-débit a lieu à Marco Island, île de Floride, lieu de villégiature pour retraités liftés percevant leurs fonds de pension sous forme de rente viagère.
La Côte Est américaine accueille les grandes entreprises de biotechnologies de la côte opposée pour démontrer que la Californie avec des sociétés telles que Illumina, Life technologies, Roche, Pacific Biosciences, Agilent… reste le meilleur fournisseur de technologies de pointes pour les gros centres de recherches situés sur la Côté Est (NIH, MIT-Broad Institute, Yale…). La Côte Est héberge les deux tiers des séquenceurs haut débit nord-américains. Depuis quatre ans, il faut bien le dire l’AGBT consacre le séquençage haut-débit, technologie qui vampirise la plupart des présentations (n’oublions pas que la jeune société PacBio sponsor principal, vend des séquenceurs…) et sessions « workshop »… au détriment de technologies plus « anciennes » (les puces à ADN par exemple). On le sait, ce type de rassemblements adoube la nouveauté et consacre ce que, parfois, hâtivement, on nomme : innovation.
Bien que ces rassemblements endogames sont critiquables, ils constituent une tribune pour les principaux axes de développements des technologies appliquées au vivant. L’AGBT est un tremplin dont se servent un grand nombre de firmes californiennes pour annoncer des lancements de machines à générer de la séquence. Cet évènement très américanocentré n’a pas de réel pendant européen, malgré les différentes formes prises par des rassemblements tels que le « carrefour des biotechnologies » remplacé par l’Eurobio-event. L’Eurobio-event sorte de biennale biotech’ où l’on trouve rassemblés beaucoup de vendeurs de robotiques, de pipettes… beaucoup de conférences traitant du business, de l’investissement dans les biotechnologies plus que des biotechnologies en tant que technologies. Assez peu de science dans Eurobio-event, beaucoup de business.
L’AGBT oscille quant à elle, entre un monde de scientifiques avide de nouvelles méthodes visant à l’exploitation rationnelle des flots de données issues de la biologie haut-débit (ce que représente l’illustration ci-dessus) et un monde de vendeurs de solutions appliquées à la recherche en biologie – quand les uns partagent un même « problème » et réfléchissent à une solution optimale pour sortir du sens biologique d’un amas de données, les autres vendent les générateurs de « problèmes » en misant sur la communauté scientifique mondiale pour apporter la réponse aux nouveaux problèmes générés. Il s’agit de trier l’information émanant de ce type de rassemblements en n’oubliant pas que cette vitrine – telle que peut l’être l’AGBT ou tout autre évènement de cette ampleur – peut parfois être un mirage scientifique plus qu’un virage technologique…
L’AGBT cette année 2012 aura lieu du 15 au 18 février sur l’île Marco Island : http://agbt.org/about.html, on y parlera de la technologie de Pacific Biosciences (des retours sur expériences sont prévus, avec leur 15 % de taux d’erreur de séquençage, il s’agit de LA déception technologique de l’année, mais PacBio est aussi LE sponsor principal de la réunion, quel suspens !), on y parlera aussi du futur Ion Proton…
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages



