Le London Calling a été l’occasion pour Oxford Nanopore Technologies (ONT) de frimer un peu avec des annonces et une gamme de séquenceurs ciblant des marchés très différents. Cette technologie de rupture risque d’être un tsunami technologique pour finir par déferler dans nos vies, car avec un séquenceur qui tient dans la poche et se connecte à un smartphone, ce qui était hier science fiction devient réalité.
Le London Calling a été l’occasion pour Oxford Nanopore Technologies (ONT) de frimer un peu avec des annonces et une gamme de séquenceurs ciblant des marchés très différents. Cette technologie de rupture risque d’être un tsunami technologique pour finir par déferler dans nos vies, car avec un séquenceur qui tient dans la poche et se connecte à un smartphone, ce qui était hier science fiction devient réalité.
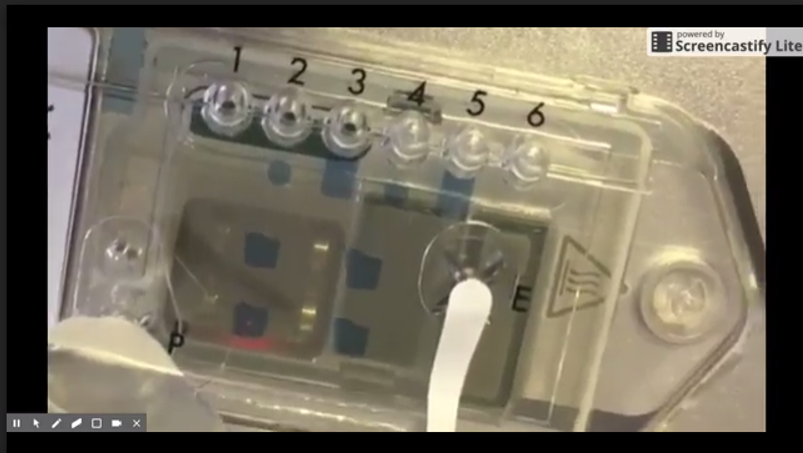
ONT a fourni, en ce début de mois de mai 2017, informations concernant les développements technologiques et les perspectives de commercialisation. L’une d’elles, concerne une nouvelle « flow cell » pour MinIon, appelée Flongle (Flow Cell Dongle) pour les applications de diagnostics cliniques. Le développement du Flongle aidera également le travail de l’entreprise sur SmidgION, un séquenceur miniature avec de petites flow cells alimentées par un téléphone mobile (voir la photo ci-dessous).
Clive Brown, responsable technologique d’ONT, a donc présenté diverses mises à jour lors du « London Calling 2017 », le grand barnum des utilisateurs de la technologie, en ce début de mois de mai. En ce qui concerne le séquenceur MinION, Brown a déclaré que la technologie est d’ores et déjà capable de délivrer plus de 20 Gb de données par run de 48 h. (Actuellement, les utilisateurs sont plus autour des 15 Gb, ce rendement plus limité serait partiellement lié à la préparation de la bibliothèque, en particulier la quantification appropriée de la taille et de la quantité d’ADN de départ, selon Brown).

En mars, la société avait parlé d’une nouvelle méthode de séquençage, appelée séquençage 1D2, où les deux brins d’une librairie bicaténaire sont poussés par nanopore séquentiellement sans être physiquement connectés. La méthode contourne un brevet détenu par Pacific Biosciences et aboutit à des lectures plus précises que la seule lecture 1D, où seul un brin de la librairie constituée est séquencé. ONT prévoit de publier le kit de séquençage 1D2 courant mois de mai 2017, ainsi qu’une nouvelle chimie appelée R9.5. Les kits 2D ne sont plus disponibles et l’entreprise vient juste d’interrompre les anciennes cellules R9.4.
Dans l’ensemble, la précision de lecture brute est maintenant supérieure à 90 % pour la chimie R9.4 et supérieure à 95 % pour la R9.5 (en mode 1D2), ces deux chimies tournant à 450 bases / min.
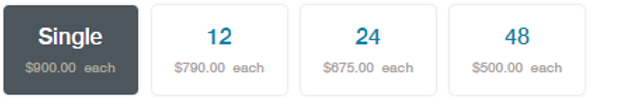
Ci-dessus, les prix actuels de la chimie 9.5. Là où l’on constate que le prix décroit drastiquement avec la quantité commandée… je pense qu’une mutualisation ou la création d’une centrale d’achats s’imposent ! Avec cette nouvelle chimie,ONT a diffusé une nouvelle version, la 1.6, de son logiciel MinKnow ainsi que de son basecaller Albacore (maintenant en 1.1). En outre, ONT a récemment commercialisé des kits de séquençage direct d’ARN et diffusé un pipeline pour réaliser le profil de résistance aux antibiotiques appelé ARMA (an analysis workflow for identification of antibiotic-resistant microorganisms in real time). À partir d’août, la société prévoit d’envoyer des flow cells à la température ambiante, des développements sont en cours pour allonger leur date limite d’exploitation (aujourd’hui une flow cell reçue doit être utilisée sous 8 semaines). Toujours visant le marché de l’utra-portabilité, en visant la décentralisation de l’acte de séquençage, ONT développe également un petit module de calcul pour le basecalling qui ferait environ la moitié de la taille du séquenceur MinION et pouvant y être directement connecté. Sur ce volet de la portabilité, le dispositif de préparation d’échantillons, VolTrax (voir vidéo ci-dessous), d’Oxford Nanopore est maintenant entre les mains de plus de 50 utilisateurs qui ont récemment reçu leurs premiers kits. La société développe actuellement un kit à base de transposase rapide et un kit d’indexation 4-plex rapide.
Les chercheurs de l’entreprise travaillent déjà sur une nouvelle version, VolTrax V2, qui devrait être disponible à la fin de 2017. Cette version permettra la PCR, la quantification des échantillons et le contrôle de la qualité des échantillons, tout en utilisant les mêmes aimants et appareils de chauffage que la version actuelle. Il sera également capable de gérer plus d’échantillons et d’exécuter des protocoles de préparation d’échantillons plus complexes. Il sera livré avec un chargeur de réactif avec des réactifs lyophilisés (lyophilisés, c’est mieux, parce que le séquenceur qui tient dans la main c’est bien, mais s’il vous faut un congélateur pour réaliser, sur le terrain, la moindre réaction…)
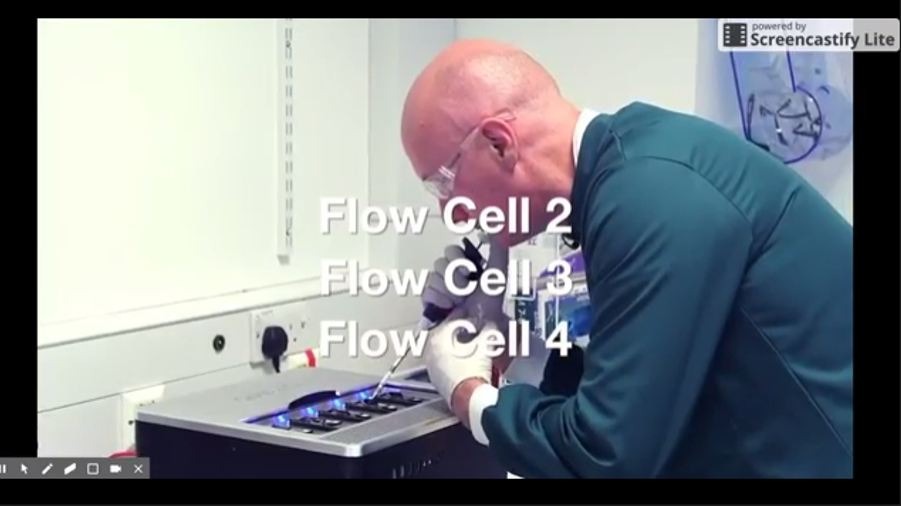
En mars, Oxford Nanopore a annoncé le lancement du GridIon X5, un séquenceur nanopore de bureau (et non plus de poche comme le MinION) pouvant permettre de travailler jusqu’à 5 flow cells à la fois et disposera d’un débit de 100 Gb par run de 48 heures. La plate-forme est livrée avec un cluster local de calcul haute performance qui permet le basecalling en temps réel ainsi que l’analyse des données. La première unité a été expédiée en début de semaine (en atteste la photo ci-dessous)

La société a récemment utilisé le GridIon pour séquencer un génome humain à 20 X, en utilisant 5 flow cells et la chimie R9.4 (en mode 1D).
Le GridION X5 permet d’exécuter simultanément ou individuellement jusqu’à cinq expériences; Les utilisateurs peuvent choisir d’utiliser tout ou partie de cette ressource à tout moment. La version actuelle de la chimie et du logiciel permet de générer jusqu’à 100 Gb de données pendant une exécution GridION X5 et le module de calcul peut analyser ces données en temps réel.
En utilisant la même technologie de base que le MinION et PromethION, le GridION X5 offre la possibilité de séquencer ADN et ARN en temps réel (dans la vidéo ci-dessous Clive Brown se transforme en Pipetman pour la promotion du GridION) :
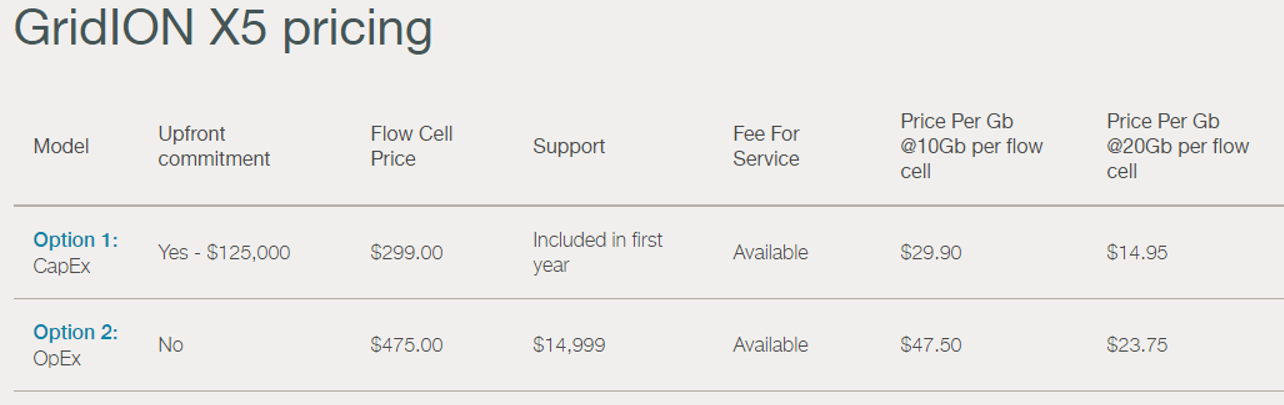
Oxford Nanopore devait envoyer il y a peu les premiers consommables pour PromethION permettant de délivrer 50 Gb et pourraient en générer jusqu’à 120 Gb /jour dans un futur proche. En vitesse de croisière cette configuration sera capable de générer plus de données que le NovaSeq d’Illumina. Du lourd, du gros et du très petit : de quoi satisfaire les plateformes de séquençages et bientôt certainement des utilisateurs non touchés actuellement par la technologie aujourd’hui, puisque la version ci-dessous permet de préparer une librairie et de la séquencer avec quelques breloques qui tiennent dans le creux d’une main !

 Plusieurs de nos articles évoquent la technologie d’Oxford Nanopore, entre impatience et curiosité.
Plusieurs de nos articles évoquent la technologie d’Oxford Nanopore, entre impatience et curiosité.
Le principe de cette technologie : séquencer sans pré-amplification (il s’agit d’un séquenceur de 3ème génération) une molécule d’ADN en en hydrolysant les nucléotides élémentaires, qui passeront dans des pores. Ces pores seront couplés à des capteurs saisissant la signature singulière de chaque base constitutive de la molécule à séquencer. Jusqu’à présent le problème majeur sur lequel ont buté les ingénieurs d’Oxford Nanopore : accélérer la célérité d’acquisition des capteurs ou ralentir le passage des nucléotides au travers des pores -nucléotides qui, au départ, s’engouffraient à plus de 1000 nucléotides par seconde ce qui provoquait des problèmes d’acquisitions des signaux et donc des erreurs, sachant que l’objectif étant de ne pas dépasser les 400 bases / seconde, seuil au-delà duquel le capture devient fou…
Le 3 mai 2012, Oxford Nanopore annonce une collecte de fonds de 31,4 millions de livres (environ 50,8 millions de dollars). Encore plus de fond pour un séquenceur qui deviendrait pour un peu, un gouffre financier… « Ce tour de table a permis une levée de fonds dont la quasi-totalité provient d’investisseurs existants, ces fonds permettront un développement de l’entreprise, comprenant le développement de notre infrastructure, un renforcement de nos forces de vente, une optimisation de la fabrication et la poursuite de la R & D » a déclaré le Dr Gordon Sanghera, chef de la direction d’Oxford Nanopore. Oxford Nanopore se préparerait donc (enfin) à vendre ses produits qui sont attendus pour la fin d’année 2012. Phénomène plutôt rassurant pour la société anglaise, elle bénéficie de la confiance renouvelée de ces investisseurs, plus informés que nous sur ce que renferment (vraiment) ses tiroirs.
Eviter l’écueil d’un PacBio, trop cher, commettant trop d’erreurs, arrivé pour occuper le marché balbutiant du séquençage troisième génération, semble être la marque d’Oxford Nanopore -une société qui prend son temps. Après la possibilité de séquencer des molécules d’ADN à l’état natif, de taille très longue, en un temps record, il semble qu’Oxford Nanopore, avec ce nouvel appel de fonds, ficelle sa stratégie commerciale pour un lancement en grande pompe de leurs 2 références : le GridION & le MinION, la clé USB qui séquence dont vous pouvez voir la photographie ci-contre
 L’AGBT qui a eu lieu du 15 au 18 février, à Marco Island, a fait la part belle à la technologie d’Oxford Nanopore, ainsi qu’il avait été prévu. La société dont il est question a profité du rassemblement pour lever le voile sur 2 produits : le MinION et le GridION, il est à noter que le MinION, mini-système de séquençage de la taille d’une grosse clé USB (photo ci-contre) a une dénomination commerciale tout particulièrement adaptée au marché français.
L’AGBT qui a eu lieu du 15 au 18 février, à Marco Island, a fait la part belle à la technologie d’Oxford Nanopore, ainsi qu’il avait été prévu. La société dont il est question a profité du rassemblement pour lever le voile sur 2 produits : le MinION et le GridION, il est à noter que le MinION, mini-système de séquençage de la taille d’une grosse clé USB (photo ci-contre) a une dénomination commerciale tout particulièrement adaptée au marché français.
La technologie d‘Oxford Nanopore a été évoquée dans plusieurs de nos articles. Elle permet le séquençage et l’analyse à haut-débit de reads de taille ultra longue (plusieurs kb) en temps réel pour pas très cher : la promesse d’un séquençage de 3ème génération démocratisé. Clive G. Brown (directeur de la technologie chez Oxford Nanopore) a présenté ses deux nouveaux jouets :
– Le MinION (dont le prix serait inférieur à 900 $) est un consommable et séquenceur (les deux à la fois) jetable qui devrait permettre de générer 1 Gb de données
– Le GridION (que vous pouvez empiler à foison, voire photo ci-dessous) permet quant à lui de générer, par module, plusieurs dizaines de Gb / jour (on pencherait pour un minimum de 25 Gb) sachant que selon nos informations un module aurait un coût voisin de 30 k$. Oxford Nanopore insiste sur le fait qu’à la Gb générée ils seront concurrentiels en terme de coût des consommables. En outre, le volume de données générées s’adapte à la problématique de l’utilisateur puisqu’en effet tant que l’appareil séquence -d’où leur slogan « Run Until« – il génère des données (le débit journalier associé à une technologie prend tout son sens ici). La cartouche –consommable de séquençage– associée à la technologie GridION possède actuellement 2000 pores individuelles -en 2013, il est prévu de passer à un consommable en comportant 8000- avec cette évolution il sera donc possible avec 20 modules GridION (environ l’investissement équivalent à un HiSeq2000) de séquencer un génome humain en 15 minutes ! Une autre façon de voir les chose est la suivante, dans sa version « actuelle » à 2000 pores disponibles : pour un prix équivalent à celui d’une configuration de type Ion Proton, 5 modules GridION seront capables de séquencer un génome humain à 30 X (cela leur prendrait une demie journée).
D’autres éléments ont filtré lors de l’AGBT. En effet, il semblerait que la technologie d’Oxford Nanopore subisse un taux d’erreurs sur séquences brutes encore assez élevé de 4 % (comparé aux plus de 10 % pour la technologie de Pacific Bioscience). Clive G. Brown aurait laissé entendre que ce taux d’erreurs serait uniforme et le fruit d’une majorité d’erreurs systématiques (ce qui est plutôt bon signe, en vue d’une rapide amélioration du système). Au niveau du système de détection, une puce GridION comporte 2K capteurs (un par pore). Chaque capteur permet de distinguer 64 signaux différents, ceci a permis d’analyser le passage de triplets de base afin de pouvoir discriminer 4x4x4 profils différents (j’avoue que j’attendais plus : quid de la prise en compte d’un signal différentiel en cas de présence d’une 5-méthylcytosine ?).
Au niveau préparation des échantillons, un séquenceur de 3ème génération ne nécessite pas de phase d’amplification. Hormis une phase préalable de légère fragmentation de l’échantillon rien ne semble envisagé. Pour palier leur problème de fiabilité, on imagine aisément qu’à l’instar de Pacific Bioscience, une circularisation de l’ADN de l’échantillon permettra d’engendrer en séquençage un nombre suffisant de répétitions venant atténuer ce point négatif.
Un élément important -mais pas surprenant- réside dans la politique commerciale affichée : une distribution directe des machines ainsi qu’une adaptation tarifaire (en usant de forfaits) offrant la possibilité d’acheter la machine à prix réduit avec un report sur le coût des consommables devrait permettre à Oxford Nanopore de conquérir quelques marchés n’en doutons pas !
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages