Simulateurs de Reads: contrôler pour mieux évaluer
 Le fait de simuler des données de séquençage est une approche de plus en plus populaire pour qui aime à jouer avec les solutions analytiques de séquençage haut-débit. Il va sans trop de développement nécessaire que l’une des caractéristiques de ces jeux de données synthétiques est leur totale maîtrise (organisme(s) à l’origine de la séquence, taux d’erreurs, d’insertion, de délétion, % de séquences contaminantes etc...). Le tout permettant relativement aisément d’exploiter des métriques telles que la F-measure qui peut se définir comme, un métrique qui combine la moyenne harmonique du rappel (sensibilité) et de la précision (spécificité), ceci donnant
Le fait de simuler des données de séquençage est une approche de plus en plus populaire pour qui aime à jouer avec les solutions analytiques de séquençage haut-débit. Il va sans trop de développement nécessaire que l’une des caractéristiques de ces jeux de données synthétiques est leur totale maîtrise (organisme(s) à l’origine de la séquence, taux d’erreurs, d’insertion, de délétion, % de séquences contaminantes etc...). Le tout permettant relativement aisément d’exploiter des métriques telles que la F-measure qui peut se définir comme, un métrique qui combine la moyenne harmonique du rappel (sensibilité) et de la précision (spécificité), ceci donnant
A des fins de comparaisons de différentes méthodes: plus une F-measure est élevée et proche de 1, plus votre méthode de mapping de reads, par exemple, sera jugée performante (encore faut il que le temps d’exécution soit jugé acceptable). Plus trivialement, ces reads synthétiques permettent de prendre en main les ressources, les logiciels et autres contingences nécessaires à une analyse post-séquençage liée à une technologie que vous souhaiteriez maîtriser. Des technologies pour lesquelles, trouver des données contrôlées, conformes à vos attentes, est plutôt difficile à exhumer. Certes la banque SRA du NCBI héberge une grande quantité de données produites sur un large spectre de technologies mais principalement dans un contexte de recherche donc difficilement contrôlable. Seules les séquences relatives à des run test, à partir de l’ADN d’organismes pris comme calibrateurs, telle que la coli DH10B permettent d’appréhender ces données en réalisant l’hypothèse que l’organisme séquencé correspond parfaitement à la séquence de référence disponible (est ce systématiquement le cas ? nous pouvons largement en douter…).
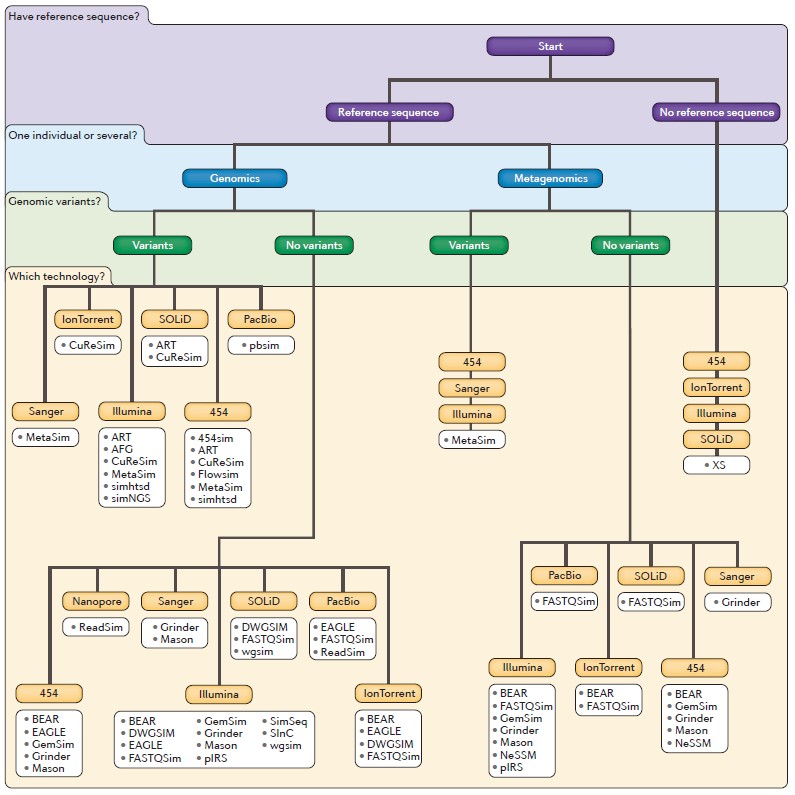
Quoi qu’il en soit un nombre croissant d’outils est disponible. Ces outils plus ou moins paramétrables permettent de simuler des données d’à peu près n’importe quel séquenceur… La publication de Merly Escalona et al. dans le Nature Reviews (Genetics) de juin 2016 vous est disponible en cliquant sur l’image « A comparison of tools for the simulation of genomic next-generation sequencing data » en tête de cette article. Cette publication est, à ce jour, le plus complet tour d’horizon de cette problématique liée aux simulateurs de données de séquençage… problématique qui n’est pas le seul apanage des bio-informaticiens ou bio-analystes…
Ce schéma reprend les caractéristiques de la vingtaine de simulateurs abordés dans la publication Escalona et al.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages