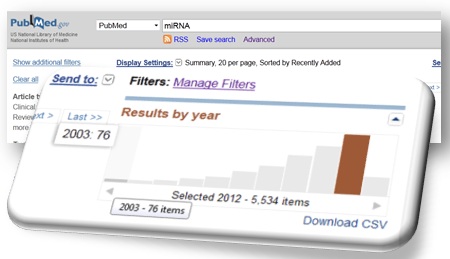
 L’accélération du débit bibliographique faisant référence aux « miRNA » atteste aisément de leur caractérisation récente (Lee RC et al., Cell (1993)) et de l’intérêt lié à leurs potentielles fonctions .
L’accélération du débit bibliographique faisant référence aux « miRNA » atteste aisément de leur caractérisation récente (Lee RC et al., Cell (1993)) et de l’intérêt lié à leurs potentielles fonctions .
Il aura fallu près de dix années supplémentaires pour mettre en évidence leur implication en tant que régulateurs biologiques (notamment au niveau de la régulation de l’expression des gènes) et leurs impacts dans de certains cancers… Aussi, le développement des nouvelles technologies de séquençage à haut débit contribue forcément à cette émergence.
Ce poste est l’occasion de présenter « miRNAtools » qui comme son nom l’indique, regroupe un grand nombre de liens renvoyant vers différents outils dédiés aux miRNA.
– Analyse de données NGS appliquées aux miRNAs (étude des profils d’expression). La liste des 7 softwares présentés n’est pas exhaustive et en voici quelques uns supplémentaires à tester: « mireap », « miRTRAP », « DSAP », « mirena », « miRNAkey », « SeqBuster », « E-mir », … . Une comparaison de l’efficacité de certains de ces outils fera l’objet d’un prochain poste.
– Prédiction de cibles selon les miRNA étudiés.
– Analyse de pathways impliquant les miR d’intérêt. Pour cette dernière application, le soft DIANA LAB – Mirpath proposé, bien que facile d’utilisation et gratuit, a le défaut de ne s’appliquer qu’aux organismes « humain » et « souris ». Dans ce registre et moyennant quelques milliers d ‘euros, « Ingenuity Pathway Analysis » (« IPA ») reste de loin l’outil idéal. En effet, en plus d’identifier les voies métaboliques au sein desquels sont impliqués les miR modulés comme proposé par Mirpath, « IPA » permet également d’intégrer les résultats de modulation de miR et d’expression de gènes pour une même condition d’étude…
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages


