Un article du Sydney Morning Herald du 20 novembre 2016 réalise un assez bon plan de communication pour la société Microba.
L’objet : les tribulations gastro-dentaires d’un testeur du service que souhaite bientôt proposer la société investissant le marché de la métagénomique personnelle… Dans la roue des sociétés proposant des services de génomique personnelle voire de génomique récréative, voici venir celles proposant d’appliquer le séquençage haut-débit couplé à de l’analyse automatisée de séquences pour révéler une part de ce qui constitue votre microflore corporelle. Presque triviales, ces analyses métagénomiques sont aujourd’hui à la portée de qui peut les payer. Vous qui ne savez qu’offrir à Noël… peut être une piste de cadeau ! La question que l’on peut légitimement se poser est : à quelles fins analyser votre flore intestinale…? revenons sur l’article du Sydney Morning Herald pour tenter d’y trouver un intérêt ou deux.

L’article commence sur un mode enthousiaste, la technologie permet de révéler une part de notre microbiote… à quoi bon s’en priver ? Le gonzo-journaliste rédacteur de l’article se propose comme cobaye et reçoit des « cotons tiges » de prélèvements qui lui permettent de procéder à l’envoi d’échantillons censés être des prélèvements de sa microflore corporelle -buccale et intestinale. Pas simple l’échantillonnage de la flore intestinale avec un coton tige ! Ainsi que le dit Dr Alena Rinke, porte-parole de Microba : « la recherche dans ce domaine vient d’exploser au cours des cinq ou dix dernières années. Nous constatons qu’il existe un nombre incroyable de liens entre les microbes de notre corps et des états pathologiques« . Ces « liens » peuvent être très divers : diabète, cancer du côlon, maladie inflammatoire de l’intestin, mélanome, polyarthrite rhumatoïde. En guise de commentaire, disons que tout comme corrélation n’est pas raison, liaison n’est pas causalité non plus. L’argument santé finit de motiver le jeune investigateur de sa flore intérieure. Donc le journaliste expédie ses cotons tiges à la société australienne qui réalise une métagénomique ciblée (séquençage haut-débit d’une portion du gène de l’ARN 16S).
Six semaines plus tard, ses résultats lui parviennent. Bilan : beaucoup de Ruminococcus, Blautia, Prevotella copri. Le petit bestiaire personnel qu’il héberge le fascine. Le test de Microba tente de mettre une couche d’interprétation de ces données de séquences mais le rendu semble un peu insatisfaisant pour le jeune intrépide. Il demande au professeur Andrew Holmes (plutôt sceptique sur l’intérêt du service) d’examiner rapidement ses résultats. La première métrique clé qui lui saute aux yeux est le nombre de Proteobacteria présente, dit – il. » En général, une personne en bonne santé a une faible proportion de bactéries dites « pro-inflammatoires ». Il faudrait pour Proteobacteria être inférieur à 5 %, et idéalement moins de 2 %. »
Le journaliste se voit rassuré avec une proportion de Proteobacteria, inférieure à 1 %.
Son microbiote est légèrement plus diversifié que la moyenne – c’est un bon point. Cela signifie qu’il est en bonne santé avec/grâce à un régime alimentaire équilibré et lui-même diversifié. Dans l’ensemble, le test de Microba conclut que sa flore intestinale suggère qu’il est en «excellente» santé avec un IMC de 20,7. Basé sur l’information de son microbiote, le test prédit qu’il a 28 ans (plutôt pas mal puisqu’il en accuse 26, avec un IMC de 21,9).
L’enthousiasme retombe un peu quand le cobaye-journaliste promoteur du service de Microba apprend qu’il a des proportions de Dialister en quantités supérieures à la moyenne. Dialister est un bon prédicteur de la gingivite. Il va donc se programmer une visite chez le dentiste ce qui fait toujours plaisir !
Comme l’a fait remarquer le professeur Holmes, les résultats reçu par le journaliste comporte beaucoup de données intéressantes – mais pas beaucoup d’idées utiles. A l’instar de ce que l’on a pu constater pour d’autres fournisseurs de « génomique personnelle », chaque échantillon reçu par Microba permet d’améliorer sa méthode de prédiction d’éléments interprétés du microbiote. Le service sera lancé au public au début d’année 2017.
Moralité : à moins d’avoir un Holmes sous la main, ces sociétés devront fournir à leurs clients des résultats automatiquement pré-interprétés pour que ces derniers puissent avoir un outil influençant leur mode de vie ou leur gestion de planning-dentiste. En effet, l’inventaire à la Prévert de ce que l’on a dans le ventre n’a que peu de sens et nécessiterait d’être remis en perspective. Cette liste, ces métriques devraient être rattachées à la singularité du client du service pour réellement pouvoir lui être utiles. Un modèle prédictif basé sur une méthode d’apprentissage automatique ne peut suffire ! On comprend que plus Microba va séquencer, plus précis sera son modèle mais son développement futur passera par une meilleure intégration d’un résultat interprétable par le commun des mortels.
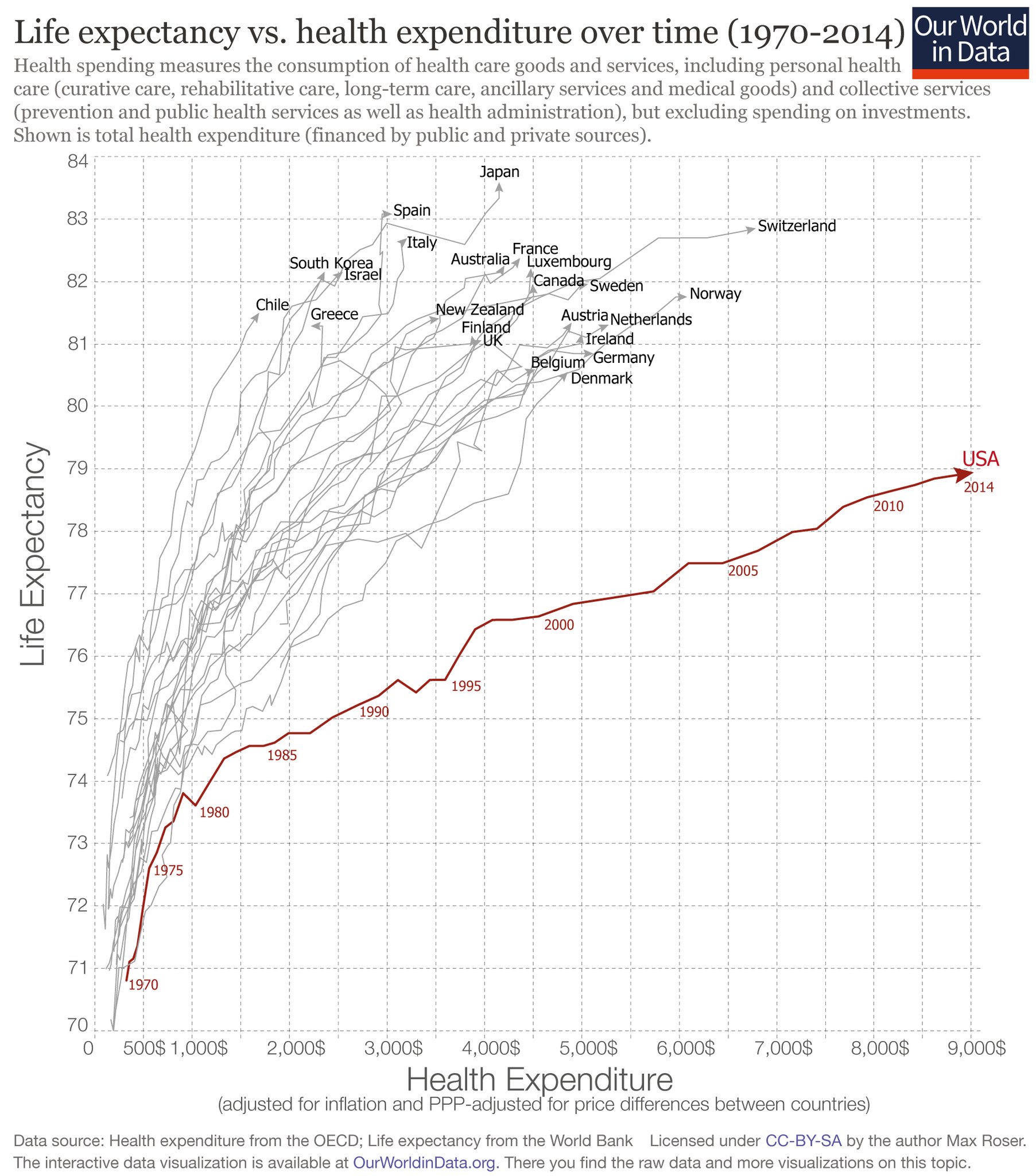
 Le développement des réseaux sociaux sur internet nous a permis d’appréhender la notion du « quand c’est gratuit, c’est toi le produit » en faisant référence, à des Facebook parmi d’autres, qui monnaient les informations personnelles des adhérents à leur service à des fins de profilage marketing. L’avènement des technologies à haut-débit en génomique a vu, il y a une dizaine d’années, émerger le concept de médecine personnalisée. Depuis, ce concept a évolué vers celui de médecine de précision (certainement plus consensuel et moins égotique). A partir de 2005, des sociétés privées se sont appropriées ce concept pour le faire déborder de la sphère de la recherche ou de la clinique pour proposer l’exploitation de profils génomiques à des fins de génomique récréative (23andMe, 2006). Un retour à l’égotisme en quelque sorte.
Le développement des réseaux sociaux sur internet nous a permis d’appréhender la notion du « quand c’est gratuit, c’est toi le produit » en faisant référence, à des Facebook parmi d’autres, qui monnaient les informations personnelles des adhérents à leur service à des fins de profilage marketing. L’avènement des technologies à haut-débit en génomique a vu, il y a une dizaine d’années, émerger le concept de médecine personnalisée. Depuis, ce concept a évolué vers celui de médecine de précision (certainement plus consensuel et moins égotique). A partir de 2005, des sociétés privées se sont appropriées ce concept pour le faire déborder de la sphère de la recherche ou de la clinique pour proposer l’exploitation de profils génomiques à des fins de génomique récréative (23andMe, 2006). Un retour à l’égotisme en quelque sorte.
23andMe a d’ailleurs un business model assez osé, consistant à vendre à des clients, leurs profils génomiques (données de puce à ADN) en échange de quelques indications concernant leur « pedigree génomique » (lire: 23 chromosomes, la NSA et moi) et quelques informations hautement critiquables. Ces dernières consistaient en des probabilités de susceptibilité à certaines afflictions (ce point là a suscité l’émoi de la FDA et ce service est aujourd’hui interrompu). En clair, 23andMe subventionne sa population de référence, ses bases de données génomiques par ses propres clients. En substance ceci constitue une évolution par rapport à Facebook, si l’on était taquin on pourrait abréger ceci en un : « vous payez pour devenir un produit ». Aujourd’hui, 23andMe habille le tout d’un « On average, a customer who consents to research contributes to over 230 studies« , se faire génotyper pour le bien de la recherche… pas loin de la servitude volontaire nappée de bon sentiment. Il est temps ici de rappeler ce que disait il y a peu un des plus grands promoteurs de la génomique personnelle : « Si je devais conseiller un jeune Craig Venter, je dirais, réfléchissez bien avant que vous veniez déverser votre génome sur Internet« . De l’aveu même de Craig Venter donc, il ne faudrait pas s’emballer avant d’envisager hypothéquer 50 % du patrimoine de ses propres enfants nés ou à naître sans qu’eux-mêmes n’aient eu mots à dire.
Aujourd’hui, arrive sur ce marché en pleine croissance, un nouvel acteur, Genos Research (page d’accueil ci-dessus). Ce dernier propose un nouveau business model : vous financez à raison de 399 USD votre propre séquençage d’exome (délivrant en moyenne 50 x fois plus de mutations que celles génotypées par l’intermédiaire des puces Illumina, telles que l’utilise 23andMe). Vous devenez propriétaire de vos données, à l’inverse des solutions concurrentes pour lesquelles, en général, vous en déléguez la propriété et l’utilisation future. Vous devenez donc le promoteur de votre propre information génomique que vous pouvez monnayer sur le marché de la recherche -priez pour avoir une maladie rare, ceci devrait accroître votre la cote de cette information. En outre, Genos permet aux utilisateurs de partager leurs données à travers leur plate-forme, ou d’exporter leurs données séquencées afin qu’ils puissent les transmettre à d’autres systèmes analytiques ou des conseillers génétiques –un business entier est d’ailleurs à bâtir : analyste de données personnelles génomiques. Genos est une société qui a bourgeonné à partir de Complete Genomics, une société de service de séquençage haut-débit, et a complété un financement privé soutenu par des entreprises en mai 2016. La société a  été co-fondée par Mark Blumling (l’ancien fondateur d’Hyperion Therapeutics, rachetée en mai 2015 par Horizon Pharma plc) et Clifford Reid (l’ancien fondateur et PDG de Complete Genomics, devenu une société de l’incroyable BGI’s company).
été co-fondée par Mark Blumling (l’ancien fondateur d’Hyperion Therapeutics, rachetée en mai 2015 par Horizon Pharma plc) et Clifford Reid (l’ancien fondateur et PDG de Complete Genomics, devenu une société de l’incroyable BGI’s company).
Pour le moins sensibles, que deviennent vos données génomiques au gré des acquisitions, fusions de sociétés de biotechnologie ? Ces changements de propriétaire sont monnaie courante et peuvent vous faire perdre de vue vos données qui pourraient finir par être le nouveau pétrole de l’ère post Donald Trump.
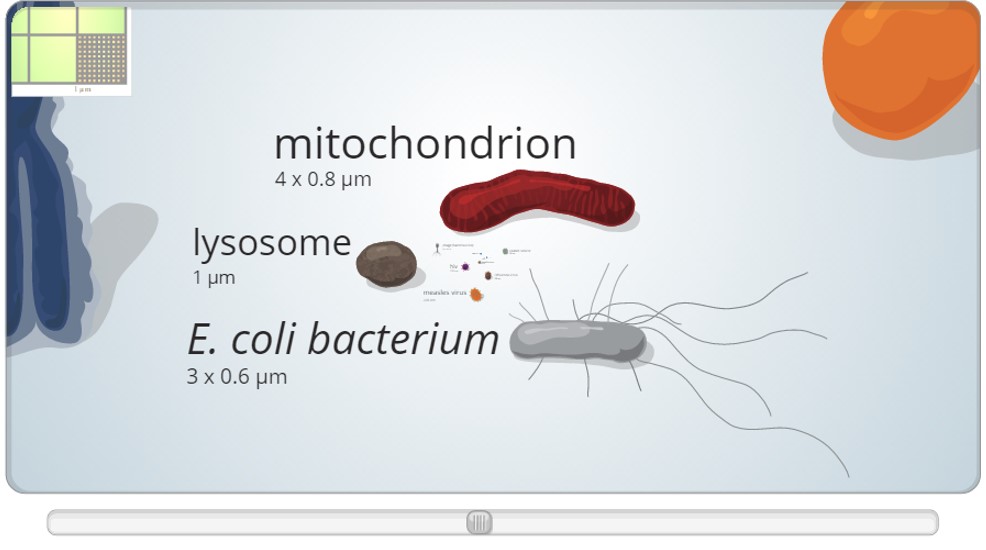
En cliquant sur l’image ci-dessus, vous pourrez vous apercevoir qu’un ribosome fait la même taille qu’un rhinovirus… une infographie très simple mais efficace pour relativiser les « tailles du vivant »
Il y a peu, le système MiSeqDx a été la première plate-forme de séquençage haut-débit approuvée par la FDA (US Food and Drug Administration) pour le diagnostic in vitro (IVD). Ceci élargit encore les applications de ces couteaux suisses de la génomique pour aller flirter avec les promesses d’une médecine de précision. C’était, en partie, le souhait des promoteurs des séquenceurs de paillasse (benchtop sequencer) du MiSeq en passant par le Ion Torrent pour aller jusqu’au prometteur séquenceur-clé USB, d’Oxford Nanopore. Ce dernier permet une analyse en temps réel des données générées par le séquenceur. Ce mode opératoire, le temps réel, trouve tout son sens dans le cadre d’applications cliniques où le temps est l’ennemi du clinicien.
Alors que les mappeurs permettant de confronter des reads générés à une référence génomique, sont optimisés pour être de plus en plus rapides, il est très étonnant voire absurde de constater que cette étape ne pouvait être réalisée qu’une fois le run de séquençage, terminé. Aujourd’hui, cet affront fait au bon entendement est en passe d’être réparé dans cette publication, d’octobre 2016, dans Bioinformatics où l’équipe de bioinformatique du Robert Koch Institute propose une première approche dans le sens d’une analyse en temps réel (à base d’extension de k-mers). Une affaire à suivre et un code source disponible : https://gitlab.com/SimonHTausch/HiLive
Source de l’article : HiLive – Real-Time Mapping of Illumina Reads while Sequencing, Bioinformatics. 2016 Oct 29
« Science et alimentation ne sont pas nécessairement fâchées ! » Regards croisés entre un chercheur et un philosophe
Source : La science au service de l’alimentation ? Regards croisés entre un chercheur et un philosophe
Le premier MOOC, en français, ambitieux, pour tous les botaniques en herbe… du néophyte au plus confirmé. Ce MOOC s’est ouvert le lundi 05 septembre 2016, dans cette première session (il y en aura d’autres), pour une durée de 7 semaines. Des semaines durant lesquelles vous pourrez collectionner des badges synonymes de votre épanouissement culturelle au sujet des plantes. Vous pouvez accéder directement au MOOC de Tela Botanica en cliquant sur la capture d’écran ci-dessous.
 Le fait de simuler des données de séquençage est une approche de plus en plus populaire pour qui aime à jouer avec les solutions analytiques de séquençage haut-débit. Il va sans trop de développement nécessaire que l’une des caractéristiques de ces jeux de données synthétiques est leur totale maîtrise (organisme(s) à l’origine de la séquence, taux d’erreurs, d’insertion, de délétion, % de séquences contaminantes etc...). Le tout permettant relativement aisément d’exploiter des métriques telles que la F-measure qui peut se définir comme, un métrique qui combine la moyenne harmonique du rappel (sensibilité) et de la précision (spécificité), ceci donnant
Le fait de simuler des données de séquençage est une approche de plus en plus populaire pour qui aime à jouer avec les solutions analytiques de séquençage haut-débit. Il va sans trop de développement nécessaire que l’une des caractéristiques de ces jeux de données synthétiques est leur totale maîtrise (organisme(s) à l’origine de la séquence, taux d’erreurs, d’insertion, de délétion, % de séquences contaminantes etc...). Le tout permettant relativement aisément d’exploiter des métriques telles que la F-measure qui peut se définir comme, un métrique qui combine la moyenne harmonique du rappel (sensibilité) et de la précision (spécificité), ceci donnant
A des fins de comparaisons de différentes méthodes: plus une F-measure est élevée et proche de 1, plus votre méthode de mapping de reads, par exemple, sera jugée performante (encore faut il que le temps d’exécution soit jugé acceptable). Plus trivialement, ces reads synthétiques permettent de prendre en main les ressources, les logiciels et autres contingences nécessaires à une analyse post-séquençage liée à une technologie que vous souhaiteriez maîtriser. Des technologies pour lesquelles, trouver des données contrôlées, conformes à vos attentes, est plutôt difficile à exhumer. Certes la banque SRA du NCBI héberge une grande quantité de données produites sur un large spectre de technologies mais principalement dans un contexte de recherche donc difficilement contrôlable. Seules les séquences relatives à des run test, à partir de l’ADN d’organismes pris comme calibrateurs, telle que la coli DH10B permettent d’appréhender ces données en réalisant l’hypothèse que l’organisme séquencé correspond parfaitement à la séquence de référence disponible (est ce systématiquement le cas ? nous pouvons largement en douter…).
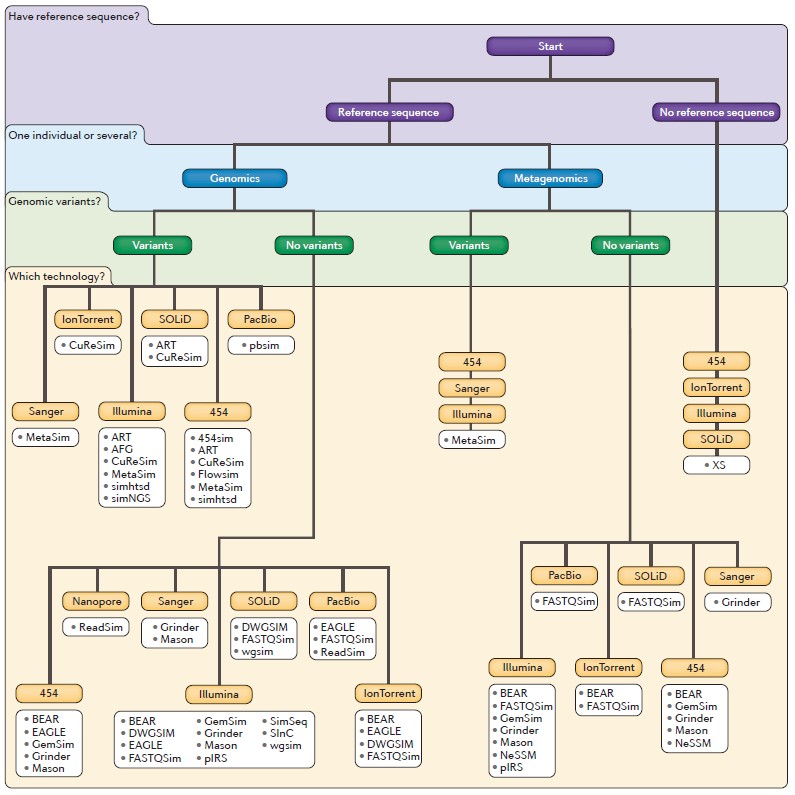
Quoi qu’il en soit un nombre croissant d’outils est disponible. Ces outils plus ou moins paramétrables permettent de simuler des données d’à peu près n’importe quel séquenceur… La publication de Merly Escalona et al. dans le Nature Reviews (Genetics) de juin 2016 vous est disponible en cliquant sur l’image « A comparison of tools for the simulation of genomic next-generation sequencing data » en tête de cette article. Cette publication est, à ce jour, le plus complet tour d’horizon de cette problématique liée aux simulateurs de données de séquençage… problématique qui n’est pas le seul apanage des bio-informaticiens ou bio-analystes…
Ce schéma reprend les caractéristiques de la vingtaine de simulateurs abordés dans la publication Escalona et al.
 A moins de sortir d’une longue phase d’hibernation, tout un chacun aura eu les oreilles rebattues par les applications liées à la technologie CRIPSR/Cas9, la révolution de l’édition de gènes (comprenez modification). Plus facile à mettre en oeuvre que les technologies TALEN et autres nucléases doigt de zinc (voir la table ci-dessous), cette technologie est tout à la fois moins onéreuse et nettement plus efficace. Cette technologie est souvent comparée, du fait de sa précision d’utilisation, à une méthode de micro-chirurgie. Un nombre exponentiel d’articles scientifiques et non scientifiques font le panégyrique de cette révolution qui est la conséquences des travaux de Jennifer Doudna et Emmanuelle Charpentier. Ces dernières ont permis de configurer cette technologie à partir de l’élucidation d’un mécanisme de vaccination primitif bactérien (nous reviendrons sur ce point fondamental dans un futur article).
A moins de sortir d’une longue phase d’hibernation, tout un chacun aura eu les oreilles rebattues par les applications liées à la technologie CRIPSR/Cas9, la révolution de l’édition de gènes (comprenez modification). Plus facile à mettre en oeuvre que les technologies TALEN et autres nucléases doigt de zinc (voir la table ci-dessous), cette technologie est tout à la fois moins onéreuse et nettement plus efficace. Cette technologie est souvent comparée, du fait de sa précision d’utilisation, à une méthode de micro-chirurgie. Un nombre exponentiel d’articles scientifiques et non scientifiques font le panégyrique de cette révolution qui est la conséquences des travaux de Jennifer Doudna et Emmanuelle Charpentier. Ces dernières ont permis de configurer cette technologie à partir de l’élucidation d’un mécanisme de vaccination primitif bactérien (nous reviendrons sur ce point fondamental dans un futur article).
| Technologie | Première utilisation | Première application sur animaux vivants | Echelle de temps pour la mise en œuvre |
| Zinc finger nucleases | 1996 | 2002 | mois / année |
| TALENs | 2010-2011 | 2011 | semaines |
| CRISPR/Cas9 | 2012 | 2012-2013 | jours |
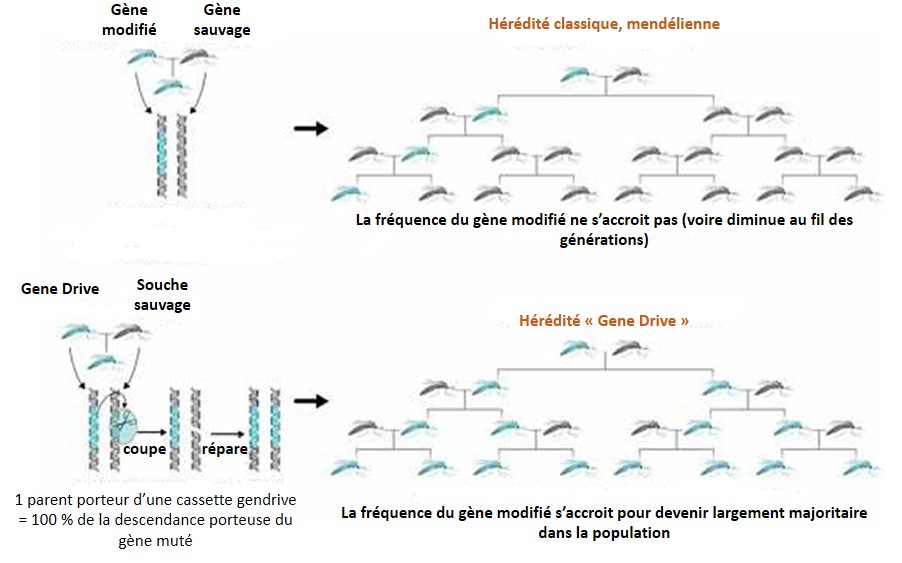
Donc non ! il ne s’agit pas ici uniquement de contribuer à l’enthousiasme, certes communicatif, de cette révolution dans le domaine des biotechnologies qui est lui-même friand de ces révolutions qui se succèdent les unes aux autres… Il s’agit dans ce premier article abordant CRISPR/Cas9 de proposer un début de décryptage de ce qui se cache derrière la terminologie de Gene Drive, application de CRISPR/Cas9. Cette terminologie est encore une fois peu aisée à traduire à mi-chemin entre le pilotage et le forçage génétique. Quoi qu’il en soit derrière l’appellation Gene Drive, se cache une technologie qui mérite que l’on s’arrête pour pousser plus avant la réflexion, tant l’enfer est pavé de bonnes intentions… Pour faire monter la pression, il faut savoir que James Clapper, le directeur du renseignement national des États-Unis, a placé la technologie CRISPR mais surtout sa formulation « Gene Drive » au niveau des armes de destruction massive… rien que ça.
Gene Drive, technologiquement concentre dans une cassette, l’outil permettant d’accélérer sa propagation (les ciseaux permettant la microchirurgie sont embarqués et encodés au sein même du génome ciblé) ainsi que le gène que l’on souhaite voir introduire au niveau d’une espèce donnée, évidemment cette technologie s’intéresse principalement aux populations sauvages. Ainsi cette cassette possède sa propre capacité de propagation, échappant aux lois de Mendel. Correctement encodée au sein d’une partie de population « augmentée ou diminuée », une dizaine de générations d’individus se reproduisant de façon sexuée permet de faire en sorte que la cassette a contaminé l’intégralité ou l’extrême majorité de la population sauvage (voir le schéma ci-dessous). L’exemple souvent développé, sponsorisé par la Fondation Bill-et-Melinda-Gates, consiste à proposer aux dirigeants africains des anophèles modifiés par cette technologie pour être résistants au parasite, Plasmodium falciparum, l’agent du paludisme. Rappelons que 650 000 personnes meurent chaque année de la malaria (rapport OMS de 2013), principalement sur le continent africain. Toujours dans les bons coups, Bill, pour soigner son karma! Ce dernier oublie néanmoins que le moustique génétiquement modifié agent mutagène puisque porteur de la cassette Gene Drive ne s’arrête pas aux frontières… mais qu’importe le moyen technologique existe donc que ne serions nous pas de fâcheux obscurantistes à réfléchir aux conséquences de l’utilisation de cette technologie avant de l’employer la fleur au fusil. Les « obscures rabat-joie » souvent du côté des écologues s’opposent aux biologistes moléculaires, rapides prescripteurs d’une technologie prometteuse pour éradiquer un nombre substantiel de fléaux, quitte à modifier durablement la nature pour l’intérêt supérieur humain ou parfois même pour l’intérêt supérieur de quelques uns…
La technologie Gene Drive peut être utilisée pour :
• Éradiquer les maladies telles que le paludisme, la dengue, la fièvre jaune, virus du Nil occidental, la maladie du sommeil ainsi que beaucoup d’autres en modifiant les espèces d’insectes vecteurs des parasites, virus, bactéries causes de ces maladies
• Éradiquer les espèces envahissantes. Les dix premières espèces envahissantes aux États-Unis causent environ 42 milliards USD de dommages chaque année
• Certains évoquent avec beaucoup d’aplomb, l’utilisation de cette technologie pour une agriculture plus « durable » en inversant la résistance aux pesticides et aux herbicides. Cette fois c’est la plante résistante au glyphosate (la molécule du RoundUp) qui est ciblée.
Que de beaux et bons sentiments… en guise de promesses de cette technologie !
Les limites de la technologie :
• De « nombreuses » générations sont nécessaires pour répandre la mutation dans la population. La durée totale dépend du cycle de reproduction de l’organisme, du nombre d’individus porteurs de la cassette Gene Drive introduit initialement dans la population, de l’efficacité de la cassette et du flux génétique (transfert d’allèles d’une population à une autre). Par exemple, cela pourrait prendre quelques années pour modifier une population d’insectes. Si 10 individus d’une population étaient porteurs d’une cassette Gene Drive parmi une population constante de 100.000 organismes, il faudrait environ 16 générations – environ un an- pour se propager à 99% de la population sous de peu réalistes, conditions optimales.
• Cette technologie est inopérante sur des organismes ne reproduisant pas de façon sexuée comme les bactéries et les virus et aura des problèmes avec les espèces qui peuvent se reproduire de façon sexuée ou non, comme cela peut être le cas pour beaucoup de plantes.
• Certains types d’altérations devraient être réintroduites sans cesse. Par exemple, une cassette Gene Drive engendrant un trait qui est quelque peu nuisible à l’organisme finira par se « briser ». De même, une cassette engendrant une résistance aux herbicides inversée dans une mauvaise herbe aurait à lutter contre la sélection naturelle dans les zones où les herbicides ont été appliqués.
La technologie ouvre donc la boîte de Pandore de la modification génétique de population sauvage. Plus l’intervalle générationnel de la population cible sera court, plus l’objectif d’une population « panmutée » sera facile à atteindre. Ci-dessous vous pourrez avoir un excellent point concernant la technologie à l’aide du travail de « the National Academies of Sciences Engineering and Medicine ».
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages