Le système alimentaire est confronté à la défiance des consommateurs tiraillés entre hantise et factualité. Les crises alimentaires, appréciées des médias, modifient notre perception d’un risque associé au besoin vital de se nourrir. À quels dangers réels le consommateur est-il confronté ? À quels défis notre système alimentaire devra-t-il faire face pour garantir notre sécurité alimentaire ?
Source : Crises alimentaires : entre risques authentiques et peurs médiagéniques

A l’occasion de son stage d’observation, la jeune Marieke nous fait partager ses remarques suite à son passage sur la plateforme de génomique Pegase-biosciences. Ce point de vue peut permettre à d’autres jeunes lycéens ayant une inclination pour le monde de la recherche en sciences de la vie de confirmer ou d’infirmer leur motivation pour débuter un parcours scientifique. Comme quoi, parfois, ces stages d’observation peuvent être un peu plus qu’une figure imposée.
Laissons la parole à Marieke !
Dans le cadre de mon année de 1ère S, j’ai effectué un stage d’observation d’une semaine, à l’Institut Pasteur de Lille. Ayant un attrait pour les sciences, je voulais découvrir le milieu de la recherche. J’avais quelques a priori que je voulais confirmer ou non, savoir si ce domaine me convient. Pendant ce stage, j’ai échangé avec 18 scientifiques (10 femmes, 8 hommes) travaillant à l’Institut Pasteur de Lille en recherche biologique. Je me suis intéressée au groupe BDPEE (axé parasitologie), et au groupe Pégase (axé génomique). En amont du stage, je me posais plusieurs questions : quels sont les métiers relatifs à la recherche, pour quel profil particulier, quelles formations pour y accéder ?
Commençons par la conclusion ! De tous ces entretiens, il en est ressorti un constat global : quelle que soit la profession exercée, le secteur de la recherche regroupe des personnes passionnées et curieuses, pour qui la science semble être une vocation. Le domaine de la recherche demande également de la rigueur, de l’organisation et un esprit de synthèse. Ces qualités sont fondamentales pour la quasi-totalité des chercheurs.
Age moyen : 30 ans
Répartition : 10 femmes (55%), 8 hommes (45%)
Nombre moyen d’années post bac : environ 5
Emploi actuel est le premier : 80%
En réalisant les interviews, j’ai constaté qu’il existait des professions différentes au sein de la recherche. Pour faciliter leur description, je les ai rassemblées par domaine.
A- Biologie « in vitro [1] »
(i) Enseignement – chercheur : 50 % recherche, 50 % enseignement
(maître de conférences, professeur)
Les 2 fonctions sont très complémentaires et se renforcent mutuellement. Les maîtres de conférences actualisent leur cours grâce aux publications et leurs travaux de recherche permettent de garder un pied dans la communauté scientifique. La transmission, le partage du savoir apportent une vraie satisfaction, complétée par le projet de recherche. Avec la HDR (Habilitation à Diriger les Recherches), les maîtres de conférences et professeurs encadrent les doctorants. Enfin, malgré peu de postes disponibles, le statut de fonctionnaire est synonyme de sécurité d’emploi.
La volonté de vouloir poursuivre des recherches, la préparation des cours, la constitution de la bibliographie (c’est-à-dire la mise à jour en continu des connaissances liées à la thématique d’étude), la recherche de moyens pour financer le projet de recherche, nécessite beaucoup de temps. Cependant cet investissement parait secondaire si l’avidité de connaissances et la curiosité sont au rendez-vous.
(ii) Gestion du laboratoire et des projets de recherche : 50 % recherche, 50 % gestionnaire
(chargé de recherche, chargé d’études)
La gestion du laboratoire implique la mise en place et l’encadrement de projets de recherche. Le chargé de recherche développe et teste des idées par la conceptualisation de protocoles, ce qui demande une capacité de concentration importante. Il encadre l’équipe technique en gérant les relations avec le reste de l’entreprise ou de l’équipe de recherche.
(iii) Equipe Technique et Ingénieur : 50 % recherche, 50 % production
(technicien, ingénieur d’étude, ingénieur de recherche)
Ces personnes collaborent aux découvertes : autant sur la mise en place des protocoles que sur les manipulations. Elles prennent part aux discussions pour enrichir et interpréter les hypothèses de travail. Le renouvellement des technologies et des techniques enrichit les connaissances, et ne laisse pas de place à la monotonie. L’autonomie et les responsabilités sont également un aspect plaisant de cette fonction. Cette profession permet une évolution de carrière.
B- Biologie « in silico [2] », Bio-informatique
(bio-informaticien, ingénieur de recherche, analyste en génétique quantitative)
Les travaux en génomique [3] génèrent un nombre très important de données qu’il faut traiter et analyser afin que les biologistes « in vitro » puissent dégager du sens, l’équipe informatique interprète les données générées, ou crée des algorithmes permettant leur interprétation. Par l’informatique, elle répond à des questions d’ordre biologique, ce qui confère un aspect pluridisciplinaire à ce domaine. L’analyste met en évidence l’essence des résultats à l’aide d’études statistiques. Le renouvellement des problématiques très diverses confère un aspect non répétitif très apprécié.
Cette science s’adresse à des personnes rigoureuses, entreprenantes, ayant un attrait pour les mathématiques (logique, analyse, statistiques).
C’est un métier de plus en plus porteur, car, grâce aux nouvelles technologies (génotypage ou séquençage à haut-débit), les données générées sont de plus en plus importantes et conséquentes.
Lorsque je me suis intéressée à la formation, je me suis rendu compte qu’elles étaient très variées. En effet, il n’y a pas une formation type, classique pour accéder au monde de la recherche. La plupart des interviewés ont un parcours atypique et on constate que les passerelles sont multiples.
Cependant, parmi les différentes orientations, certaines voies sont plus souvent empruntées :
BTS – formation concrète délivrant des bases solides notamment pour les manipulations.
Fac (Licence et Master) – enseignement général, pluridisciplinaire. Recommandé pour les étudiants qui hésitent sur leur orientation. Cependant, cette formation nécessite une motivation non négligeable.
Doctorat – 3 ans consacrés à la réalisation d’une thèse. Il peut éventuellement être « surdiplômant ».
Je trouve que c’est un milieu propice à la réflexion scientifique, passionnant, idéal si on aime s’investir dans des projets. Certains enseignants – chercheurs sont capables de passer 8 ans sur un seul parasite, le Cryptosporidium par exemple.
J’ai été particulièrement impressionnée par la quantité d’anglais. La maîtrise de l’anglais scientifique est une aptitude vitale en recherche pour communiquer lors des congrès, pour lire et rédiger des protocoles et des articles.
Je ne savais pas du tout que la publication avait une place si importante dans le quotidien des chercheurs. A la fois pour se tenir informé des avancées, pour communiquer ses découvertes, pour faciliter l’obtention de moyens financiers, pour critiquer les travaux de recherches, pour se faire reconnaître, les scientifiques publient des articles, souvent en anglais, dans la presse scientifique. La publication est nécessaire pour trouver des financements. Ainsi, une sorte de cercle vicieux s’installe progressivement : il faut des financements pour publier et pour obtenir ces financements, il faut publier. Or plus le journal à une audience importante, plus il a un facteur d’impact élevé et plus il est difficile de publier dans ce journal. Cette pratique, fondamentalement utile au sein de la communauté scientifique, est cependant contestée. Par exemple, au-delà des 192 heures de cours obligatoires, il est demandé aux maîtres de conférences de produire un article par an. Cela peut éventuellement déboucher sur une pratique de la publication déraisonnée et excessive.
[1] In vitro : locution latine signifiant au laboratoire, par extension ici cela désigne les expériences menées au laboratoire
[2] In silico : locution latine signifiant par ordinateur
[3] La génomique est une discipline de la biologie moderne. Elle étudie le fonctionnement d’un organisme, d’un organe, d’un cancer, etc. à l’échelle du génome, et non plus limitée à celle d’un seul gène
L’un des pionniers des séquences nucléique et protéique est décédé, il y a peu, le 19 novembre 2013, à l’âge de 95 ans. Il avait commencé sa thèse en octobre 1940 avec pour projet de rechercher une protéine consommable purifiée d’herbe (relativement abondante dans le royaume de Georges VI). C’est, au final, sous la direction d’Albert Neuberger (qui avait fuit l’Allemagne avec l’arrivée d’Hitler au pouvoir) que Frederick Sanger devient docteur en 1943.
Les deux hommes se respectaient, à propos de « son maître » Albert, Frédérick Sanger avait déclaré : « c’est Neuberger, qui le premier, m’a appris à faire de la recherche, à la fois techniquement et comme un mode de vie, et je lui dois beaucoup. »
L’homme qui a donné son nom à une technique de séquençage des nucléotides ouvrant la voie à un pan entier d’une science, à un institut de recherche (le Wellcome Trust Sanger Institute) employant plus de 900 personnes est une fierté (jamais anoblie) de l’empire britannique : une des quatre personnes à avoir reçu deux prix Nobel (prix Nobel de chimie). Son premier prix Nobel de chimie pour le séquençage de l’insuline (1958) sanctionne un travail de 10 ans. Rejoignant Crick (le penseur), au laboratoire de biologie moléculaire du Conseil médical de la Recherche à Cambridge, Sanger (le fonceur) se met à développer une technique de séquençage de l’ADN. Ce travail lui apportera son second prix Nobel en 1980.
« Quand j’étais jeune, mon père me disait que les deux activités les plus intéressantes dans la vie sont la recherche de la vérité et de la beauté, et je crois qu’Alfred Nobel a dû ressentir la même chose quand il a donné ces prix pour la littérature et les sciences.«

Un document reprenant le nécessaire à savoir sur l’oeuvre de Frederick Sanger (réalisé par Richard Daniellou, Enseignant-chercheur, ENSCR) est disponible au format pdf.
L’image en tête de cet article est issue d’un portrait de Frederick Sanger réalisé par Paula A. MacArthur (huile sur toile : 1991, collection: National Portrait Gallery de Londres).
 Tout commence ici par un film de piètre qualité (je ne crois pas qu’il en existe une version française), Who the #$&% Is Jackson Pollock ? Ce film retrace l’histoire d’une femme, Terri Horton, de 73 ans, ancienne conductrice de camion long courrier qui a acheté une toile dans un magasin d’aubaines pour 5 USD. Il s’avère que cette toile a été attribuée à Jackson Pollock et en demande 50 millions de USD. Le film dépeint les tentatives de Horton pour authentifier et vendre la peinture comme une œuvre originale de Pollock. Son authenticité est douteuse, car la peinture a été achetée dans un magasin d’aubaines, n’est pas signé, et est sans provenance. Le principal problème avec la peinture, c’est qu’elle « n’a pas l’âme d’un Pollock », selon les collectionneurs. En outre, Pollock avait de nombreux imitateurs de son vivant. C’est là qu’intervient un spécialiste en techniques légales d’authentification, iconoclaste et donc décrié, Peter Paul Biro. Un document pdf (disponible ici) reprend toute la palette mise à disposition de ce dernier pour permettre une authentification légale. Tout part d’une observation « microscopique » dont l’objectif est de rechercher des traces éventuelles de l’artiste (poils, cheveux, empreintes digitales etc.). Les pages 13 et 14 abordent la technique mise en œuvre.
Tout commence ici par un film de piètre qualité (je ne crois pas qu’il en existe une version française), Who the #$&% Is Jackson Pollock ? Ce film retrace l’histoire d’une femme, Terri Horton, de 73 ans, ancienne conductrice de camion long courrier qui a acheté une toile dans un magasin d’aubaines pour 5 USD. Il s’avère que cette toile a été attribuée à Jackson Pollock et en demande 50 millions de USD. Le film dépeint les tentatives de Horton pour authentifier et vendre la peinture comme une œuvre originale de Pollock. Son authenticité est douteuse, car la peinture a été achetée dans un magasin d’aubaines, n’est pas signé, et est sans provenance. Le principal problème avec la peinture, c’est qu’elle « n’a pas l’âme d’un Pollock », selon les collectionneurs. En outre, Pollock avait de nombreux imitateurs de son vivant. C’est là qu’intervient un spécialiste en techniques légales d’authentification, iconoclaste et donc décrié, Peter Paul Biro. Un document pdf (disponible ici) reprend toute la palette mise à disposition de ce dernier pour permettre une authentification légale. Tout part d’une observation « microscopique » dont l’objectif est de rechercher des traces éventuelles de l’artiste (poils, cheveux, empreintes digitales etc.). Les pages 13 et 14 abordent la technique mise en œuvre.
Même si l’attribution d’un tableau à son auteur par l’intermédiaire de la caractérisation singulière de l’ADN de ce dernier restera anecdotique, le besoin d’identification et d’authentification peuvent être un marché potentiel pour des sociétés de biotechnologie.
Ce sujet avait été abordé dans notre article « l’ADN, nouveau support de l’information numérique » qui montrait l’utilisation faite de cette molécule en guise de code barre pour traquer les pétroliers dégazant le reliquat de leur cargaison (je serais curieux de connaître le nombre de capitaines mazouteurs confondus par cette méthode).

Ainsi, l’une de ces sociétés de biotechnologie, la société Applied DNA Sciences, Inc, propose des produits d’aide à l’identification et l’authentification. L’ADN devient un code barre censé être inviolable, un élément visant à protéger de la contrefaçon des processeurs, des vêtements, des spiritueux. Une autre application vise à sécuriser les transferts de fonds (un nouveau moyen pour marquer et tenter de suivre les billets marqués).
Cette société surfant sur le besoin de sécurité et de dissuasion développe un spray fluorescent chargé en molécules d’ADN. Mise en situation… Un voleur braque un bijoutier à l’aide d’une arme de poing (cf. schéma ci-dessous).
Le bijoutier a deux choix :
. user de violence et de mesures coercitives pouvant flirter avec le délit
. oublier qu’un revolver est pointé sur lui, appuyer sur un bouton rouge qui asperge l’agresseur d’un spray fluorescent génétiquement marqué… prier pour que cela suffise à mettre en fuite le braqueur.
On l’aura compris, il ne s’agit pas là de l’idée du siècle. L’ADN est mis à toutes les sauces, de l’authentification à l’arme absolue de dissuasion pour tout braqueur effrayé par l’association d' »idées » : ADN + experts police + haute technologie = preuve = prison.

L’ADN comme moyen d’identifier l’auteur d’un tableau -comme quelque chose dans la continuité des empreintes digitales de Vinci laissées lors de son brillant sfumato– au molecular fingerprinting des peintres contemporains en passant par l’absurde spray anti-braqueur qui laisse esquisser un sourire moqueur, la molécule support de l’information génétique est d’ores et déjà la base d’un business associé au besoin d’authentification sans cesse plus important…
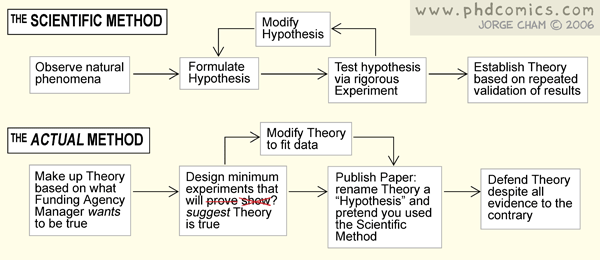
 Un bon nombre de thésards abordent fébrilement la période de la fin d’été. Un été passé à bronzer à l’ombre des néons clignotants des laboratoires et arpentant des bureaux devenus déserts, on imagine le thésard comme une ombre fantomatique à la recherche d’une machine à café… une vision, un peu cliché ? Quoi qu’il en soit, un site internet vous permettra de travailler légèrement votre anglais et de faire preuve d’un peu d’autodérision : il s’agit du très connu et parfois même brillant : PhD comics (www.phdcomics.com). Il s’agit de brasser les névroses des aspirants chercheurs pour porter leur point de vue sur ce futur métier pollué par tant d’images d’Epinal.
Un bon nombre de thésards abordent fébrilement la période de la fin d’été. Un été passé à bronzer à l’ombre des néons clignotants des laboratoires et arpentant des bureaux devenus déserts, on imagine le thésard comme une ombre fantomatique à la recherche d’une machine à café… une vision, un peu cliché ? Quoi qu’il en soit, un site internet vous permettra de travailler légèrement votre anglais et de faire preuve d’un peu d’autodérision : il s’agit du très connu et parfois même brillant : PhD comics (www.phdcomics.com). Il s’agit de brasser les névroses des aspirants chercheurs pour porter leur point de vue sur ce futur métier pollué par tant d’images d’Epinal.




Ainsi, le PhD -dans le contexte de PhD Comics l’abréviation est mise pour Piled Higher and Deeper en lieu et place de Philosophiæ doctor (l’intitulé du diplôme de docteur dans le système universitaire anglo-saxon) – est un parcours semé d’embûches entre désillusions, travail de força et stress d’un avenir pour lequel le diplôme de docteur ne semble plus un passeport pour un travail universitaire intellectuellement enrichissant et financièrement roboratif.

Jorge Cham, l’auteur des textes et dessins, connait le sujet ! Il a, en effet, débuté cette bande dessinée alors qu’il était étudiant à Standford où il visait un doctorat en génie mécanique. Comme le monsieur est dessinateur de comics, universitaire et américain, il s’est engagé dans un vaste tour des universités (plus d’un centaine) pour présenter son « the Power of Procrastination » (le pouvoir de la procrastination). Avec cette thèse (à prendre ici dans les deux sens), Jorge Cham, contrecarre les discours tenus par les coachs certainement plus cupides que psychologues, adeptes du développement personnel à l’anglo-saxonne. En effet, lors de sa présentation, l’auteur de PhD Comics démontre que le fait de remettre à plus tard est en général quelque chose de bénéfique pour l’élaboration d’un concept (ce en quoi il mérite l’appellation primitive de PhD !).
Ceci étant, PhD Comics met le doigt sur une réalité tangible de notre côté de l’Atlantique : une espèce de « paupérisation » du diplôme et avant cela du statut des étudiants se destinant à l’accomplissement d’une thèse… des éléments faisant tellement partie du panorama qu’on ne les voit plus (sur le sujet lire : Thésard, une vie de loser – article de Libération du 20 mars 2007)
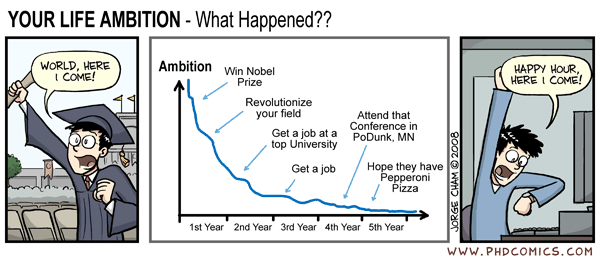
Encore que pour ce qui nous intéresse ici, les thésards des Sciences du vivant peuvent être enviés par ceux des Sciences humaines qui sont entrés avant eux dans cette spirale de la lose. Donc la caricature proposée par PhD Comics nous amène à nous poser des questions quant au statut et au devenir d’étudiants parfois désabusés, mais aussi à la perception de la science comme objet d’accomplissement professionnel (plus que comme discipline intellectuelle). Ainsi, il en va du morceau choisi ci-dessous.
 Quand l’histoire d’un homme entre en collision avec l’Histoire évolutive des hominidés et de ce que Carl Linnaeus en 1758, nomma Homo sapiens, cela produit un article au titre provoquant : « L’homme qui ne descendait pas d’Adam » (Pierre Barthélémy dans son excellent blog, Passeur de Sciences, 10 mars 2013).
Quand l’histoire d’un homme entre en collision avec l’Histoire évolutive des hominidés et de ce que Carl Linnaeus en 1758, nomma Homo sapiens, cela produit un article au titre provoquant : « L’homme qui ne descendait pas d’Adam » (Pierre Barthélémy dans son excellent blog, Passeur de Sciences, 10 mars 2013).
Tout d’abord, il semble utile de rappeler le concept d’Adam et Eve phylogénétique. Théologues rangez vos saintes écritures, il s’agit ici d’un concept scientifique et technique d’anthropogénétique. Les variations génétiques liées au chromosome Y, ainsi que celles présentes sur la mitochondrie sont utilisées pour étudier l’histoire de ces seuls fragments chromosomiques. L’Adam du chromosome Y et l’Ève mitochondriale ne sont donc que des vues de l’esprit.
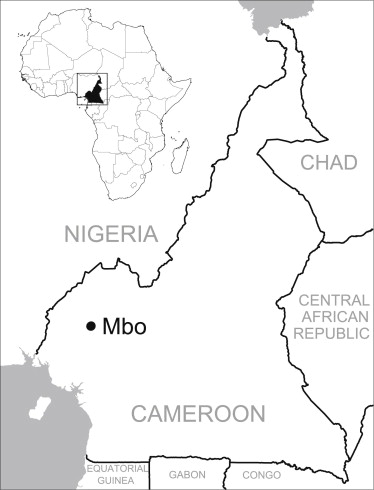
Parlons donc du cas d’Albert Perry dont le chromosome Y ne ressemblait à rien de connu. Actuellement, l’Adam du chromosome Y a vécu théoriquement entre 60.000 et 140.000 ans avant maintenant (la Eve mitochondriale quant à elle, aurait théoriquement vécu entre 140.000 et 240.000 ans avant maintenant). Surprise, après étude du chromosome Y d’Albert Perry, on s’étonne d’être passé à côté de cette lignée humaine. En exhumant des bases de données dont visiblement l’analyse n’avait pas été complète, Mendez et al. ont trouvé 11 séquences issues du chromosome Y de 11 membres de la tribu des Mbo (An African American Paternal Lineage Adds an Extremely Ancient Root to the Human Y Chromosome Phylogenetic Tree, AJHG, 2013). Cette tribu est plus ou moins localisée au Nord Ouest du Cameroun, une région d’où a été déporté un grand nombre d’esclaves. Le fait d’ajouter cette lignée humaine « oubliée », l’âge de notre Adam théorique doublerait (en accord avec la théorie de la coalescence) pour être comprise entre 237-581 000 ans.
- Carte localisant approximativement les personnes parlant le dialecte des Mbo – tirée de AJHG, Mendez et al., 2013
Le cas Perry est un exemple intéressant à plusieurs titres. En tout premier lieu, il permet de voir comment un cas particulier étudié peut avoir une incidence sur la compréhension de l’histoire humaine. En ce sens, ceci est à porter au crédit de la génomique personnelle : avec le développement des sociétés telles que 23andMe, on peut présumer que dans un avenir proche, les nouvelles données que le particulier payera afin de connaitre sa propre histoire, serviront peut être à l’ensemble de la communauté scientifique… tel que l’exemple d’Albert Perry (passé à la postérité) le suggère. Ensuite, cet exemple montre à quel point la connaissance est relative. Notre développement technologique, notre puissance de génération de données avec pour limite actuelle le temps accordé à leur analyse, laissent présager que de plus en plus vite notre connaissance sera volatile, les références changeront de plus en plus vite…
Ce petit article est aussi l’occasion de vous proposer une visite sur le très beau, très documenté blog francophone : The fossil and Mr Darwin et particulièrement de prendre le temps de lire l’article dont il est question ici : mais où est passé Adam ?
Bonne lecture !
Les médias se font l’écho des conséquences dues à la révolution technologique engendrée par l’évolution du séquençage haut-débit.
Qu’est ce que la biotechnologie peut faire pour vous ? Comment peut-elle repousser la date de péremption de vos artères ?
En effet, Arte (pour TF1, il va falloir attendre un épisode de Dr House traitant le sujet) diffuse un documentaire américain de moins d’une heure dont l’objectif est de présenter les évolutions technologiques dans le champ de la médecine personnalisée. Il s’agit tout d’abord de développer le concept pour le grand public (en s’appuyant sur des exemples où le mélodrame l’emporte un peu sur le débat bioéthique).
Ce document présente les acteurs majeurs de cette révolution biotechnologique qui sont tous américains (alors que, rappelons le, la plus grande plateforme de séquençage au monde est chinoise). Malgré quelques défauts, il s’agit d’un documentaire faisant tout d’abord oeuvre de pédagogie et développant des exemples plutôt précis comme autant d’arguments poussant à la large utilisation du séquençage haut-débit. Le documentaire vaut surtout pour l’accent mis sur la manière d’interpréter les résultats, sur la pertinence de ceux-ci mais tait les notions économiques liées à un futur marché qui s’annonce éléphantesque.
N’est ce pas un problème que votre génome soit, dans un avenir proche, séquencé, « interprété » et stocké par une firme privée ?
Le documentaire dont il est question est disponible ici-même (vous trouverez le synopsis dans les quelques lignes ci-dessous)
« LE DÉCRYPTAGE DU GÉNOME HUMAIN
D’ici peu, en échange de quelques centaines d’euros, chacun d’entre nous pourra obtenir un séquençage complet de son ADN. Des informations qu’il sera possible de lire, de stocker et de soumettre à l’analyse. Cette révolution est déjà en marche, ainsi que le montrent deux exemples spectaculaires : un patient cancéreux qui semble défier la mort et un malade de la mucoviscidose à présent capable de respirer normalement. Dans ces deux cas, les scientifiques sont parvenus à détecter et à neutraliser les anomalies génétiques à l’origine des maladies. Demain, grâce au décryptage du génome, il sera possible de faire un diagnostic personnalisé pour chaque individu et de mettre en place des traitements ultra personnalisés. Mais quelles sont les conséquences d’une telle révolution ? Connaître les maladies dont nous aurons à souffrir dans l’avenir, est-ce une bénédiction ou un fardeau ? Que se passerait-il si de telles informations tombaient entre les mains de compagnies d’assurance, d’employeurs ou de futurs conjoints ? Une chose est sûre : cette nouvelle ère, marquée par une médecine personnalisée s’appuyant sur nos gènes, est une réalité qui nous concerne tous. »
(Etats-Unis, 2012, 53mn)
ARTE F
Date de première diffusion : Jeu., 25 avr. 2013, 22h50
Date(s) de rediffusion : Dimanche, 5 mai 2013, 10h50
Pendant ce temps, de l’autre côté de l’Atlantique, Laurent Alexandre intervient dans l’émission grand public : ce soir (ou jamais !). Le débat avait pour thème l’obsolescence programmée. Ainsi au bout de 1h20 d’émission Frédéric Taddeï lance le débat sur l’obsolescence du corps humain. Après un extrait de l’assez mauvais film Repo Men de Miguel Sapochnik (réalisateur de certains épisodes de Dr House… c’est dire s’il doit s’y connaître en médecine personnalisée…), Laurent Alexandre, l’auteur du livre « la mort de la mort » et président de la société DNAVision, intervient ensuite. Son discours est clair et d’une grande pédagogie, il est peut être un peu dommage que l’exemple mis en avant fut le diagnostic prénatal non invasif de la trisomie 21 par séquençage haut-débit.
Evidemment très rapidement l’argument eugéniste se fait entendre. Finalement, l’intervention de Laurent Alexandre fait écho au documentaire américain.
Si les questionnements liés à la bioéthique sont au cœur du débat, rarement sont abordées les questions économiques et de confidentialité des données, associées à l’exploitation de nos données les plus intimes : la connaissance quasi-exhaustive de notre génome (et donc d’une partie de celui de nos descendants qui n’auraient rien souscrit).
Car depuis la publication de Gymrek et al., dans Science de janvier 2013 : Identifying personal genomes by surname inference, il semble difficile de soutenir l’anonymisation des données présentes dans les banques publiques. Il semble difficile d’imaginer que déposer son génome dans les banques d’une firme privée ne constitue pas un problème dont il faut d’ores et déjà prendre la mesure.
Car si tout le monde s’accorde sur les bienfaits potentiels de la médecine qui exploite la connaissance approfondie des bases nucléiques d’un patient ou d’un bien portant-futur mourant, actuellement le pouvoir est aux mains des firmes privées (Knome, 23andMe), qui se constituent des bases de données colossales. Ces bases de données hébergeant des quantités exponentielles de génomes humains (dans l’immédiat il s’agit plutôt de génotypes) permettront à leurs détenteurs de dominer un marché (voire plus !) qui s’annonce colossal…
 A l’heure où Jonathan Rohtberg et Craig Venter cherchent de l’ADN sur Mars (lire : Searching for Alien Genomes), l’ADN sur Terre ne serait-il pas à la veille de changer de statut ? En effet, si l’ADN est « le support de l’information génétique »… est-il possible que cette macromolécule devienne le support de l’information tout court ?
A l’heure où Jonathan Rohtberg et Craig Venter cherchent de l’ADN sur Mars (lire : Searching for Alien Genomes), l’ADN sur Terre ne serait-il pas à la veille de changer de statut ? En effet, si l’ADN est « le support de l’information génétique »… est-il possible que cette macromolécule devienne le support de l’information tout court ?
– C’est humblement, dans le domaine de la traçabilité, que l’ADN a trouvé un rôle d’espion : Ainsi, au début des années 2000, la société norvégienne ChemTag, tente de développer des systèmes permettant de tracer dans nos océans, le pétrole retrouvé suite à d’éventuels dégazages. L’idée est simple : l’ADN est utilisé comme un code-barre, la succession du code de quatre lettres permet assez rapidement d’obtenir un code (4^n) ne permettant qu’avec une probabilité infime d’être retrouvé par hasard. Il faut savoir que par année, plus d’un million de tonnes de pétrole sont déversées dans les océans. La problématique en vaut la peine, Poséïdon en sera gré. Ici toute la difficulté qui se présentait à la société ChemTag : faire en sorte que l’ADN ait une grande affinité pour le pétrole (évidemment en cas de dégazage si le traceur ADN se dissout dans l’océan… adieu code-barre) et permettre que ce traceur soit purifié le plus simplement possible. L’ADN est une molécule plutôt stable et plutôt facile à « lire », ne modifiant pas les qualités organoleptiques d’une substance… et surtout cette molécule permet un nombre infini de combinaisons.
Moult industriels souhaiteraient être capables d’identifier en toute objectivité (légalement) tout organisme vivant leur appartenant (une souche de production par exemple). A cette fin, le code barre biologique peut être utilisé de deux manières :
– artificiellement, en « incorporant » par manipulation génétique une séquence connue. Cette option est bien souvent écartée parce qu’elle fait appel à la notion d’OGM, mention qui peut effrayer le consommateur.
– naturellement, en connaissant le patrimoine génétique du génome employé dans un procédé industriel. Cela implique de déterminer une séquence singulière qui n’appartiendrait qu’à cette organisme. Souvent ce type de procédures fait appel à une combinaison de séquences ou de loci polymorphes.
L’évolution des techniques de séquençages (augmentation des débits, diminutions des coûts, rapidités des runs) a permis de rendre accessible une séquence complète d’un micro-organisme ou une carte de SNPs haute-densité pour certains eucaryotes supérieurs dont on dispose de puces SNPs.
Aujourd’hui, certains envisagent d’exploiter l’ADN pour en faire un support robuste de l’information numérique.

Depuis quelques semaines, une petite bataille a lieu entre l’équipe de Georges Church de la Medical School de Boston et Christophe Dessimoz de l’EBI.
Le premier a encodé un livre dont il est le co-auteur : Regenesis: How Synthetic Biology Will Reinvent Nature and Ourselves in DNA comportant 53000 mots et 11 images en jpeg accompagné d’un programme Javascript pour un total de 5,37 Mo dans un picogramme d’ADN (un peu moins de 1 Gbase). Le second a encodé du Shakespeare, un discours de Martin Luther King, une photo et la copie d’un article de 1953 décrivant la structure de l’ADN… on s’amuse plutôt pas mal aussi de ce côté de l’Atlantique !
Ces querelles d’encodeurs ont malgré tout un intérêt. Ils démontrent la faisabilité de ce type de système et montrent qu’il est désormais envisageable de sauvegarder pour des échelles de temps très longues, de grandes quantités d’informations qui échapperaient à bien des autodafés.
Les systèmes d’encodage au sein même de l’ADN relégueront nos séquenceurs haut-débit au niveau de lecteurs DVD ou Blu Ray du futur… La limite du système, actuellement, tient plus des synthétiseurs d’ADN et des technologies de séquençage qui n’ont pas réellement été conçus à cette fin. Mais à l’heure où l’on promet d’ici quelques dizaines de mois un séquençage de génome humain pour une centaine de dollars contre quelques milliards il y a 10 ans, tous les espoirs sont permis pour que l’information séculaire contenue dans une bibliothèque patrimoine de l’Humanité devienne une bibliothèque d’Alexandrie du futur, franchissant les âges, échappant aux catastrophes naturelles, aux censures et à l’oubli.
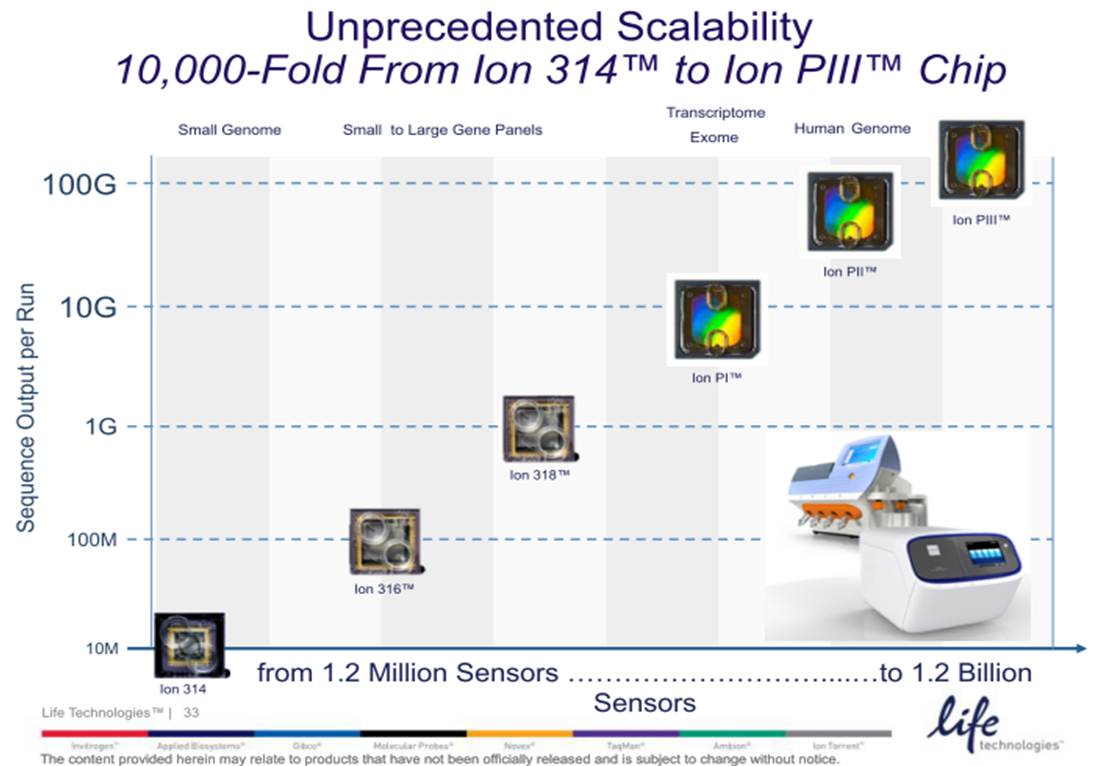 Le mois de décembre 2012 a connu le lancement du kit 400 bases (
Le mois de décembre 2012 a connu le lancement du kit 400 bases (
Ion PGM™ Template OT2 400 Kit) qui est principalement attendu pour les applications de séquençage de novo et de séquençage d’amplicons ciblant le 16S, 18S ou encore ITS2 (respectivement pour l’étude de la diversité bactérienne, eucaryote ou fongique) pour les études métagénomiques. Comme vous pouvez le constater cette application est uniquement supportée par le One Touch 2 devenu nécessaire pour accéder aux reads de 400 bases (c’est en partie pour cette raison que notre plateforme PEGASE-Biosciences a décidé de se doter de cette automate).
L’autre nouveauté est portée par les consommables de séquençage associés au Ion Proton (le grand frère du PGM). En effet, si la Puce Ion PIII est encore virtuelle, la PI et PII, permettent de délivrer : 10 Gb et 70 millions de reads filtrés environ pour la première, contre plus de 280 de millions de reads et plus de 70 Gb pour la seconde (de quoi séquencer un génome humain à 20 X).
Du côté de Life Technologies et de sa technologie Ion Torrent, sans aucun doute, la principale nouveauté de l’année 2013 touche l’amplification clonale (phase où, à partir de la librairie préparée, est réalisée la matrice de séquençage fixée de manière covalente sur des billes qui se logeront dans les puits présents sur la puce). Ainsi, la fameuse (et pour certains la fâcheuse) phase de PCR en émulsion trouvera une alternative avec la chimie isothermique dite « avalanche » (dont vous pouvez voir l’aperçu d’un run ci-dessous). A en croire Life Technologies, cette chimie ouvre la voie à des tailles de séquences générées encore plus longues.
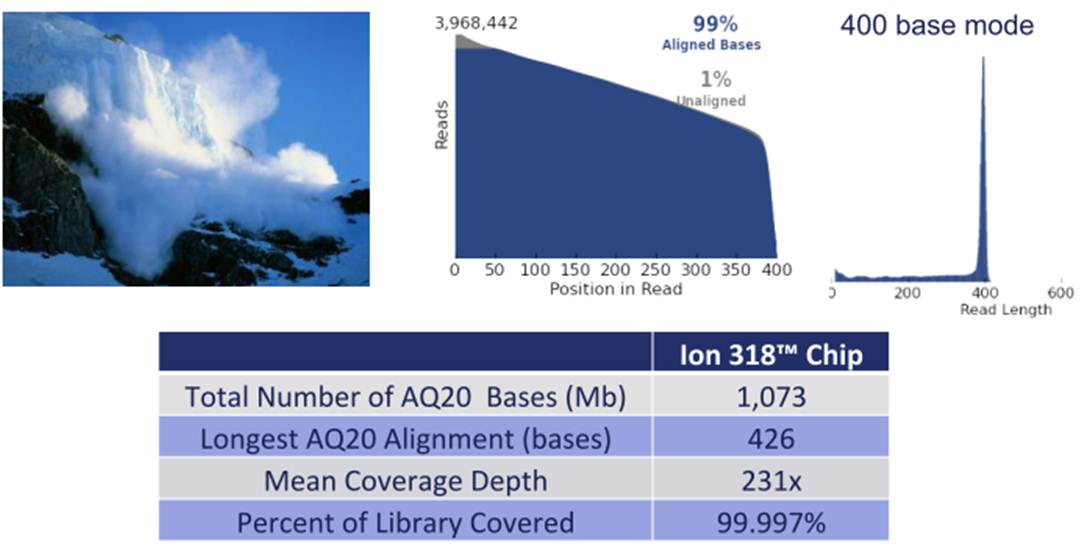
Bien entendu, Life Tech annonce l’arrivée d’un nouvel automate permettant d’intégrer la nouvelle chimie mais aussi de charger les puces (et pour quelques dizaines de k€, d’épargner au manipulateur les deux phases délicates du séquençage).
Pour conclure brièvement, l’une des nouveautés et non des moindres : l’acceptation du PGM comme diagnostic moléculaire CE-IVD (un parcours pas vraiment de tout repos) a n’en pas douter une conformité qui ne devrait pas laisser insensible les chercheurs, praticiens hospitaliers.
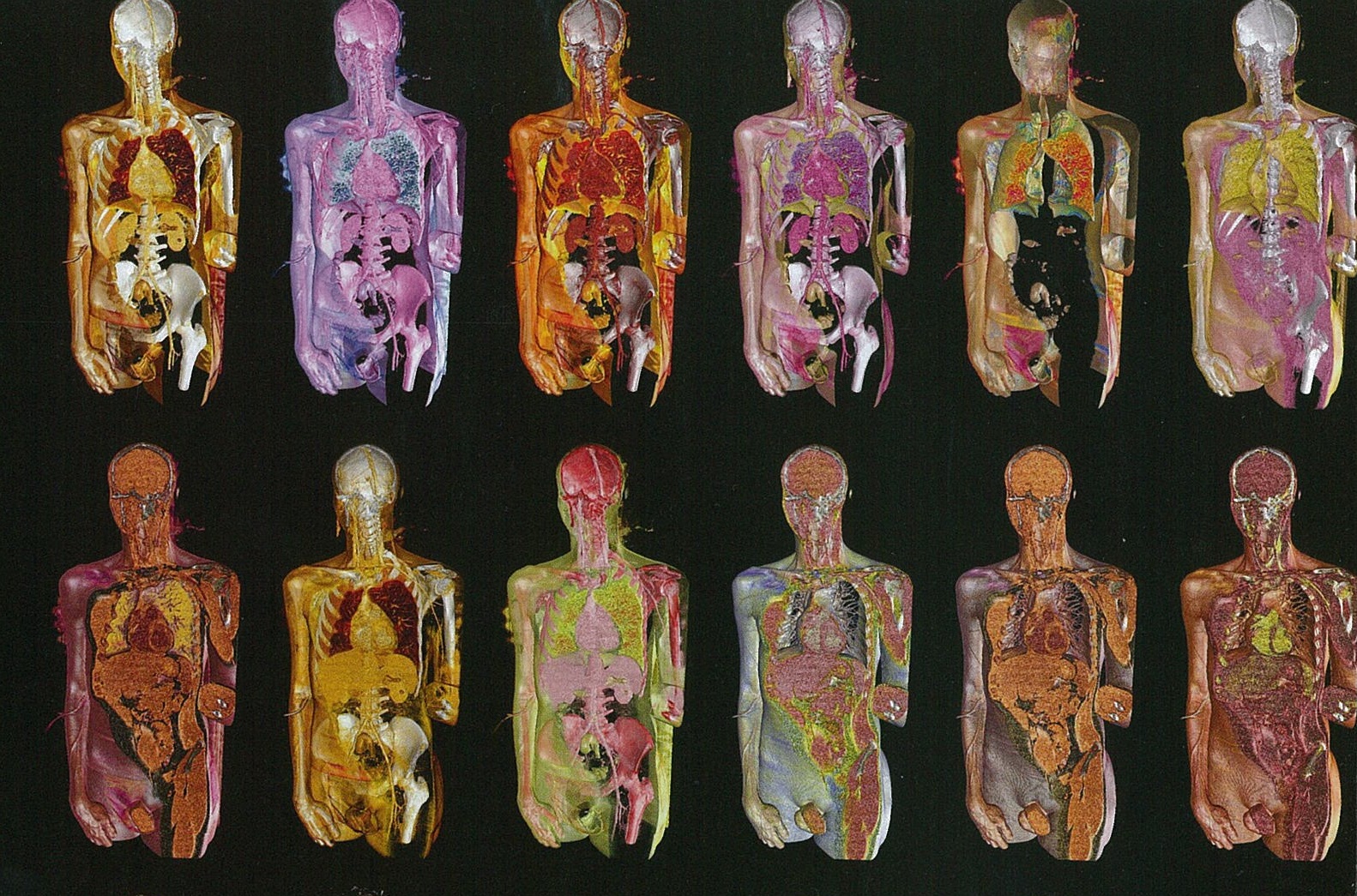 « La réalité virtuelle » ou « la réalité augmentée », « l’art génératif » ou encore « l’art interactif », autant de déclinaisons possibles pour designer « l’art virtuel« . Cette forme d’art nouveau est à la croisée des chemins de l’informatique, de la technologie ou encore du graphisme.
« La réalité virtuelle » ou « la réalité augmentée », « l’art génératif » ou encore « l’art interactif », autant de déclinaisons possibles pour designer « l’art virtuel« . Cette forme d’art nouveau est à la croisée des chemins de l’informatique, de la technologie ou encore du graphisme.
Parmi les plus médiatiques de la discipline, à la fois radiologue et photographe, Rodolphe von Gombergh détourne les techniques de l’imagerie médicale de pointe (ultra-sons, ondes électromagnétiques, rayons X et imagerie 3D) pour créer son propre univers et révéler ses secrets dans l’art de la trans-apparence ( Une des modalités du Bio Art évoqué au cours d’un article précédent).
Dès les prémisses de l’imagerie médicale, il se passionne pour la discipline et, fort d’une équipe de recherche et développement, contribue à l’explosion de l’imagerie médicale moderne. les procédés utilisés permettent de modéliser le corps humain en faisant apparaître les organes comme transparents. Cet explorateur des corps eut l’occasion d’exposer ses premières images médicales au musée Beaubourg. Dix ans plus tard, c’est au musée Guimet que seront livrées les découvertes des scanner des célèbres bouddhas du XI au XVII ème siécle (http://www.dailymotion.com/video/xqtdo2_virtual-art-ou-la-science-qui-se-mele-a-l-art_news).
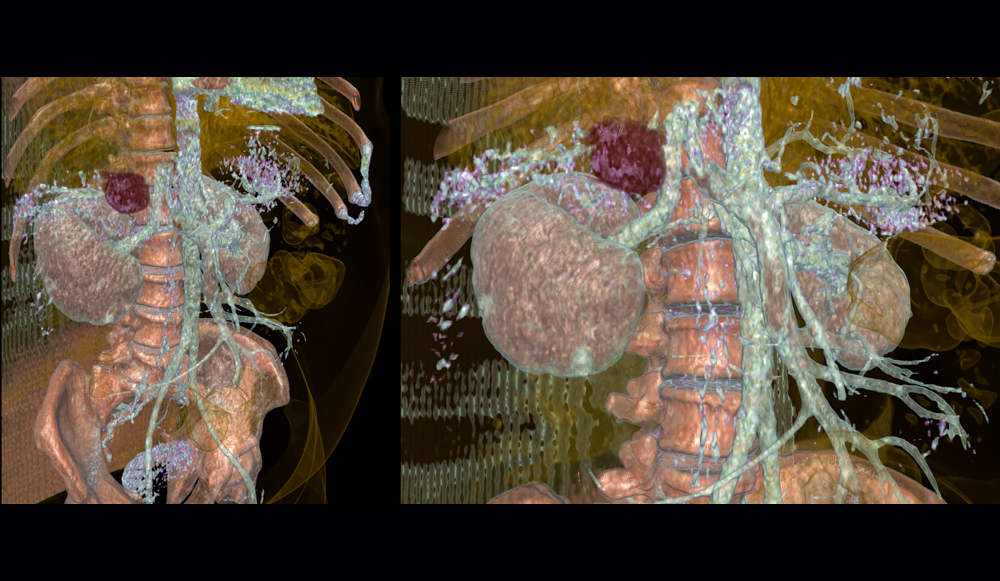
Sur le plan médical, il est aisé d’imaginer la puissance de ces méthodes non invasives pour le dépistage d’anomalies (exemple ci dessous présentant une tumeur de la surrénale), d’autant qu’elles ne nécessitent aucune anesthésie ni ne présentent de risque d’infection pour le patient…
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages