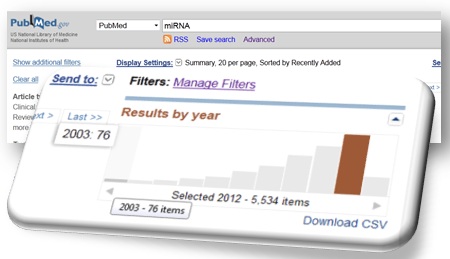
 L’accélération du débit bibliographique faisant référence aux « miRNA » atteste aisément de leur caractérisation récente (Lee RC et al., Cell (1993)) et de l’intérêt lié à leurs potentielles fonctions .
L’accélération du débit bibliographique faisant référence aux « miRNA » atteste aisément de leur caractérisation récente (Lee RC et al., Cell (1993)) et de l’intérêt lié à leurs potentielles fonctions .
Il aura fallu près de dix années supplémentaires pour mettre en évidence leur implication en tant que régulateurs biologiques (notamment au niveau de la régulation de l’expression des gènes) et leurs impacts dans de certains cancers… Aussi, le développement des nouvelles technologies de séquençage à haut débit contribue forcément à cette émergence.
Ce poste est l’occasion de présenter « miRNAtools » qui comme son nom l’indique, regroupe un grand nombre de liens renvoyant vers différents outils dédiés aux miRNA.
– Analyse de données NGS appliquées aux miRNAs (étude des profils d’expression). La liste des 7 softwares présentés n’est pas exhaustive et en voici quelques uns supplémentaires à tester: « mireap », « miRTRAP », « DSAP », « mirena », « miRNAkey », « SeqBuster », « E-mir », … . Une comparaison de l’efficacité de certains de ces outils fera l’objet d’un prochain poste.
– Prédiction de cibles selon les miRNA étudiés.
– Analyse de pathways impliquant les miR d’intérêt. Pour cette dernière application, le soft DIANA LAB – Mirpath proposé, bien que facile d’utilisation et gratuit, a le défaut de ne s’appliquer qu’aux organismes « humain » et « souris ». Dans ce registre et moyennant quelques milliers d ‘euros, « Ingenuity Pathway Analysis » (« IPA ») reste de loin l’outil idéal. En effet, en plus d’identifier les voies métaboliques au sein desquels sont impliqués les miR modulés comme proposé par Mirpath, « IPA » permet également d’intégrer les résultats de modulation de miR et d’expression de gènes pour une même condition d’étude…
 Les outils d’assemblage, de mapping sur référence deviennent relativement pléthoriques… il est de ce fait relativement aisé de choisir l’outil permettant de gérer au mieux les défauts de votre séquenceur, vous n’avez que l’embarras du choix en somme. Même si cette étape n’est pas à négliger, il n’en reste pas moins que le rôle du biologiste restera toujours d’apporter du sens à une ou plusieurs millions de séquences, informations qui viendront tenter d’élucider une question biologique. Les outils permettant de faire parler un ensemble de reads, que vous pratiquiez le de novo RNA-seq ou séquençage génomique de novo sont plutôt restreints, peu diffusés, limités ou tout simplement absents… Ces grands absents constituent le point noir de l’analyse secondaire de vos reads. De nombreux biologistes recherchent un financement (séquencer massivement coûte encore un peu cher !), font séquencer l’objet de leur étude -hors humain, rat, souris- repartent quand tout va bien avec de beaux contigs, et se retrouvent face à un livre écrit dans une langue inconnue. Ce livre est censé renfermer la réponse à beaucoup de leurs questions… on comprend leur frustration d’autant qu’ils ne possèdent pas de pierre de Rosette.
Les outils d’assemblage, de mapping sur référence deviennent relativement pléthoriques… il est de ce fait relativement aisé de choisir l’outil permettant de gérer au mieux les défauts de votre séquenceur, vous n’avez que l’embarras du choix en somme. Même si cette étape n’est pas à négliger, il n’en reste pas moins que le rôle du biologiste restera toujours d’apporter du sens à une ou plusieurs millions de séquences, informations qui viendront tenter d’élucider une question biologique. Les outils permettant de faire parler un ensemble de reads, que vous pratiquiez le de novo RNA-seq ou séquençage génomique de novo sont plutôt restreints, peu diffusés, limités ou tout simplement absents… Ces grands absents constituent le point noir de l’analyse secondaire de vos reads. De nombreux biologistes recherchent un financement (séquencer massivement coûte encore un peu cher !), font séquencer l’objet de leur étude -hors humain, rat, souris- repartent quand tout va bien avec de beaux contigs, et se retrouvent face à un livre écrit dans une langue inconnue. Ce livre est censé renfermer la réponse à beaucoup de leurs questions… on comprend leur frustration d’autant qu’ils ne possèdent pas de pierre de Rosette.
Cet article part d’un constat, l’important est moins de savoir si votre séquenceur fournit les reads les plus propres, les plus longs, si votre assembleur est le plus adapté… car tous ces efforts resteront vains si vous écrivez un livre dont personne ne peut comprendre le sens. J’arrête là de filer la métaphore.

Blast2GO -ici dans sa version gratuite- est un outil qui trouvera vite (trop vite) ses limites mais qui permettra d’apporter du sens à des séquences générées à haut-débit. La publication de Conesa et al. (Bioinformatics – 2005), dévoile le pipeline d’analyse. Grossièrement, il s’agit de blaster des séquences (en 2005 – on envisageait par séquences des reads et non des reads assemblés) automatiquement sur le serveur du NCBI, les résultats de blast sont récupérés, les autres phases du pipeline sont réalisées en interrogeant les bases de données de Blast2GO, in fine des termes GO permettent d’annoter les séquences dont on souhaite décoder le sens. La principale qualité de Blast2GO réside dans son ergonomie, l’interface Java6 permet une prise en main directe (aucune ligne de code). Ses défauts sont multiples… tout d’abord Blast2GO plante… très régulièrement, ensuite pour une séquence d’entrée (dont la taille sera nécessairement inférieure à 7000 bases) vous ne disposerez que d’une annotation pour le meilleur des hits.
 Lors d’articles précédents nous vous avions présenté le logiciel de Workflow Galaxy, qui permet d’analyser et de visualiser toutes sortes de données biologiques à partir d’une interface simple d’utilisation.
Lors d’articles précédents nous vous avions présenté le logiciel de Workflow Galaxy, qui permet d’analyser et de visualiser toutes sortes de données biologiques à partir d’une interface simple d’utilisation.
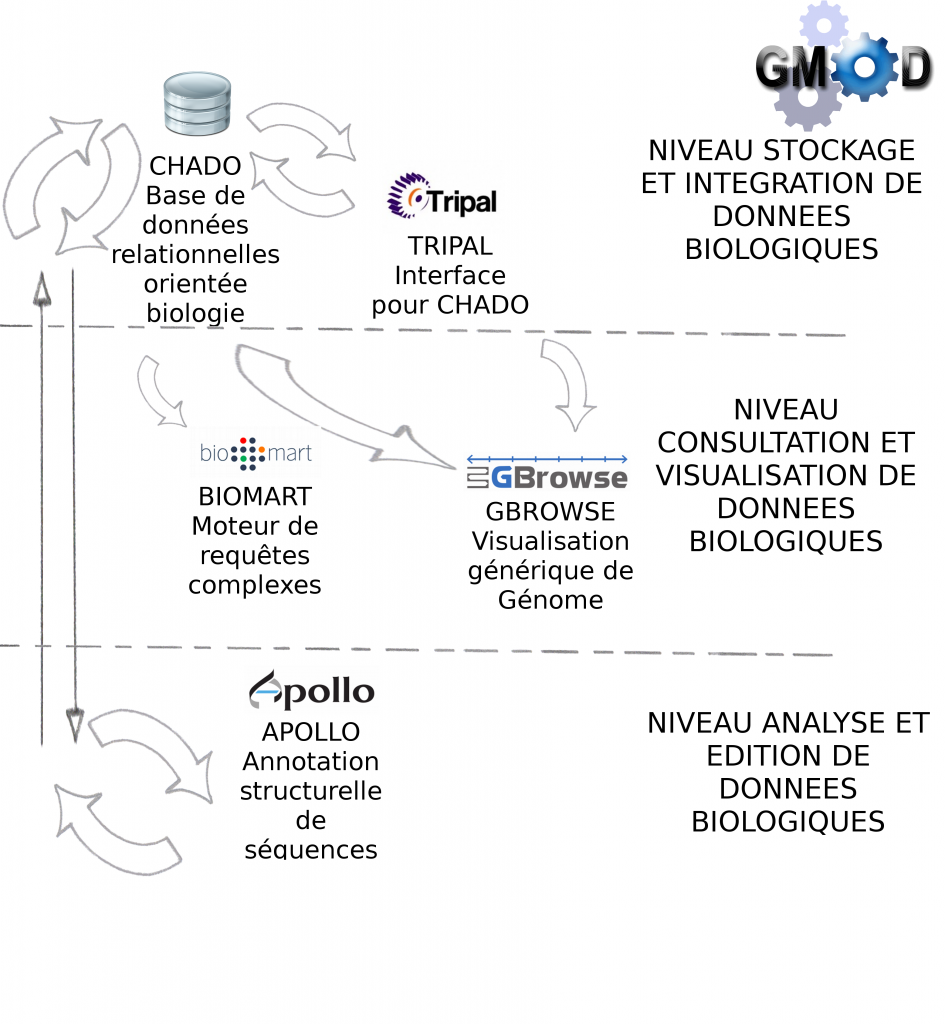
Galaxy est en fait une brique d’une collection d’outils dédiés à l’analyse et au stockage de données biologiques : GMOD ( Generic Model Organism Database )
Le lundi 14 mai a été l’occasion pour nous d’assister à une conférence sur l’utilisation de certains des outils GMOD, dont voici les principaux enseignements :
> Le projet GMOD a pour objectif de fournir à l’utilisateur biologiste un ensemble d’outils interconnectés, libre de droit (open-source), générique (pour tous types de données biologiques) et facile d’utilisation (à travers des services Web principalement)
> Les outils GMOD sont développés (et donc installés) par des bioinformaticiens pour une utilisation par des biologistes.
> Certains outils sont indispensables à GMOD, pour la manipulation de données génomiques, c’est le cas des outils Chado et Gbrowse qui sont respectivement les squelettes pour la manipulation et pour la visualisation des données biologiques.
Le schéma ci-dessous décrit les modules et interactions présentés lors de cette journée thématique :
> GBrowse est un outil de visualisation de données biologiques très puissant et certainement l’outil le plus populaire de la suite GMOD
Description détaillée : GMOD-GBROWSE
> Biomart est un outil de recherches avancées (ou requêtes complexes) pour la base de donnée relationnelle Chado
> Apollo est un module pour la correction manuelle d’annotation structurelle
Description détaillée : GMOD-APOLLO
> Tripal est une interface web développée en PHP pour interrogation de Chado
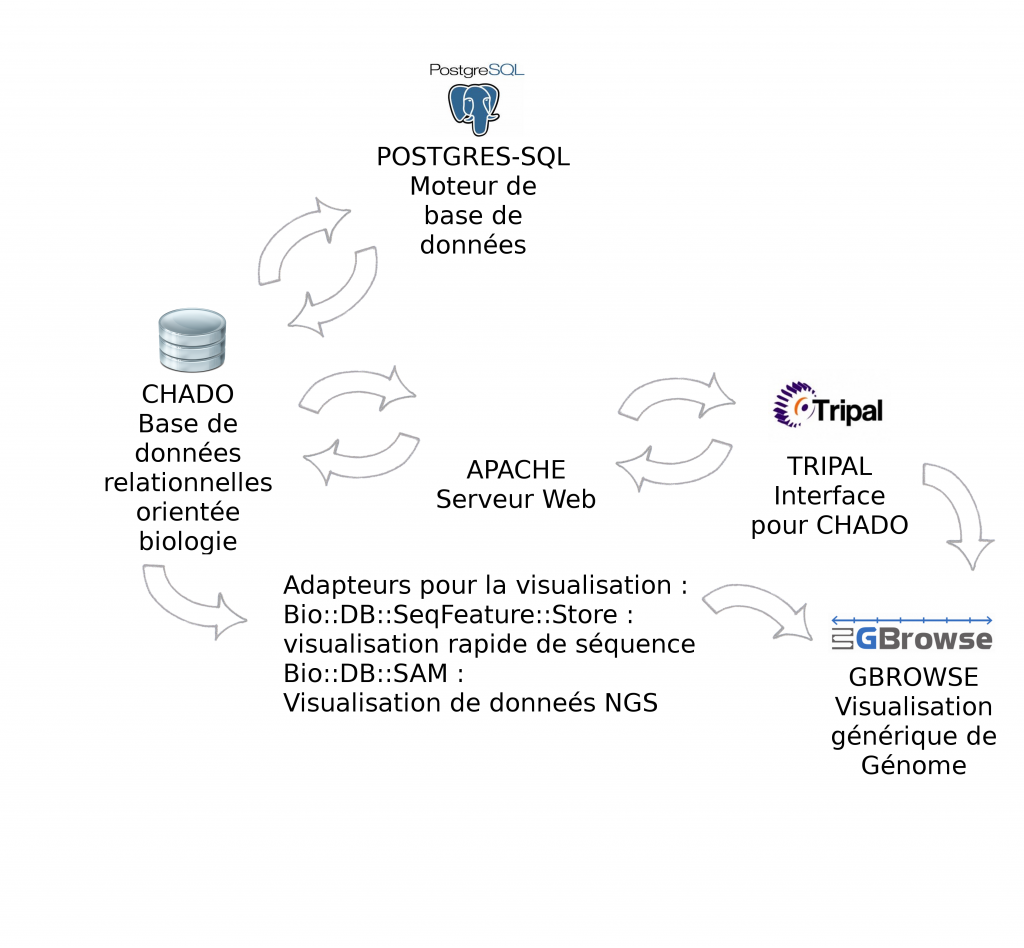
Environnement informatique : CHADO fonctionne par défaut avec Postgres-Sql. Pour l'interfaçage avec Tripal, la solution étant développée avec PHP, il est nécessaire d'installer un serveur Web Apache. Gbrowse peut s'utiliser à partir de l’interface Tripal mais également en "stand-alone". Afin d'accélérer la visualisation des données il est vivement recommandé d'utiliser les adapteurs Bio::DB::*, soit en relais entre Chado et Gbrowse (présentés dans cet article), soit en dupliquant les informations dans les deux bases de données (la visualisation ne se connecte qu'à Bio::DB sans passer par CHADO). Intuitivement, nous privilégions la première solution qui n’entraîne pas de duplication.
Pour conclure, GMOD propose un ensemble de modules pour la standardisation des processus bioinformatique : stockage et manipulation de données biologiques, visualisation et analyses avancées (assemblage, annotation…).
L’utilisation de tels outils (open-source) va dans le bon sens pour le partage scientifique et la standardisation des processus utilisés lors de l’analyse bioinformatique. Cela n’était pas le but de la conférence à laquelle nous avons assistée mais il serait également intéressant de connaître les conditions pour intégrer ces propres outils bioinformatiques en tant que brique GMOD.
GMOD est donc intéressant si :
> vous souhaitez stocker et gérer vos données biologiques
> vous cherchez des solutions d’analyses bioinformatiques déjà développées et robustes
Les biologistes sont les utilisateurs des outils GMOD, en revanche l’installation, l’administration et la formation des utilisateurs ne peuvent échapper à l’intervention, au moins ponctuelle, d’un bioinformaticien.
Si vous projetez d’utiliser ces outils, nous vous conseillons donc dans un premier temps, de regrouper l’ensemble des acteurs, installateurs comme utilisateurs de GMOD, afin de présenter les solutions offertes par l’outil et déterminer les besoins et objectifs pour votre propre utilisation.
Voici un lien sur le site Molecular Biology Freeware for Windows… un lien destiné aux biologistes un peu allergiques à Linux et qui ne souhaitent pas débourser des milliers d’euros dans des logiciels qui ne peuvent s’avérer utiles que sporadiquement (pour certains).
Le site Molecular Biology Freeware for Windows est particulièrement laid mais il permet de référencer ces logiciels utiles pour bon nombre d’applications : phylogénie, alignement de séquences, etc.
L’alliance un peu paradoxale du logiciel gratuit, fiable et utile sous Windows !
Ce site-référence est pour quelques uns d’entre vous… : il classe les logiciels outils de biologie moléculaire en fonction des catégories suivantes :
A. DNA, RNA and genomic analysis
B. Plasmid graphic packages
C. Primer design
D. Protein analysis
E. Viewing three dimensional structures
F. Alignments
G. Phylogeny
H. Miscellaneous
I. Graphic packages
Le réseau régional d’ingénieurs en bioinformatique de Lille et le PPF bioinformatique organisent lundi 14 Mai 2012 une conférence sur l’utilisation des outils GMOD (Generic Model Organism Database). Cette conférence se déroulera de 13h30 à 17h30 dans l’amphithéâtre de l’Institut de Biologie de Lille.
« Le projet GMOD a pour objectif de fournir une série d’outils génériques, clé en main, pour gérer et visualiser différents types de données biologiques. »
Deux interventions sont prévues :
Olivier Arnaiz du CGM Centre de Génétique Moléculaire (UPR 3404 Gif-sur-Yvette) introduira GMOD tout en se focalisant plus particulierement sur deux outils : Chado et Gbrowse. Chado est un schéma de base de données relationnelles qui permet d’intégrer des données « omiques » (génomiques, transcriptomices, séquençages…), tandis que GBrowse est un outil permettant de visualiser des annotations sur un génome.
Joelle Amselem de l’INRA – URGI (BIOGER Versailles) présentera un outil d’annotation développé à l’URGI à partir de modules GMOD. Deux exemples concrets d’utilisation seront développés.
Pour en savoir plus, vous pouvez télécharger le programme ici.
Si vous souhaitez des informations sur GMOD, je vous invite à consulter la présentation de GMOD.
Cet article a pour but d’aider à la mise en place d’un serveur Galaxy en local. Il décrira également les avantages et inconvénients d’une telle approche par rapport à d’autres utilisations (Galaxy en ligne et Galaxy sur cloud).
Pour une définition de ce qu’est le logiciel de workflow Galaxy vous pouvez lire l’article précédemment posté sur ce blog ou accéder à la description PLUME du logiciel.
Préambule :
Il existe trois méthodes pour utiliser Galaxy :
> En local : c’est la méthode que nous décrirons ci-dessous
> En ligne : outre l’instance principale de Galaxy, de multiples organismes ouvrent l’utilisation de leurs serveurs Galaxy à l’extérieur ( pour une liste exhaustive cliquer ici)
> Sur un cloud : On utilise notre propre instance (virtuelle) de Galaxy mais au lieu d’être déployés sur une machine locale, les « jobs » sont envoyés sur le cloud de Amazon
Chaque approche a ses avantages et ses inconvénients, nous les détaillerons plus bas.
Installation :
Un grand nombre d’indications sont extraites du tutorial d’installation de Galaxy en local, disponible, en anglais, à cette adresse – http://wiki.g2.bx.psu.edu/Admin/Get%20Galaxy.
Ce qu’il faut retenir de l’installation de Galaxy en local est qu’il est possible de mettre en place un serveur en moins de 10 minutes!
Brève Explication du processus d’installation (sous un serveur Debian – les étapes seront les même pour Ubuntu):
Ouvrir une invite de commande
– Vérifier sa version de python :
python --version
La version 2.5 (et au-delà) est suffisante pour un bon fonctionnement de Galaxy.
– Installer le système de gestion de version mercurial : la version 1.6 est suffisante. S’ il est déjà installé, vérifier votre version :
hg --version
– Télécharger l’archive dans un répertoire (par exemple « workflow ») approprié :
hg clone http://bitbucket.org/galaxy/galaxy-dist
– Lancer Galaxy! Dans le répertoire galaxy-dist :
sh run.sh --reload
Note : reload permet de fermer correctement l’application sous debian (et ubuntu)
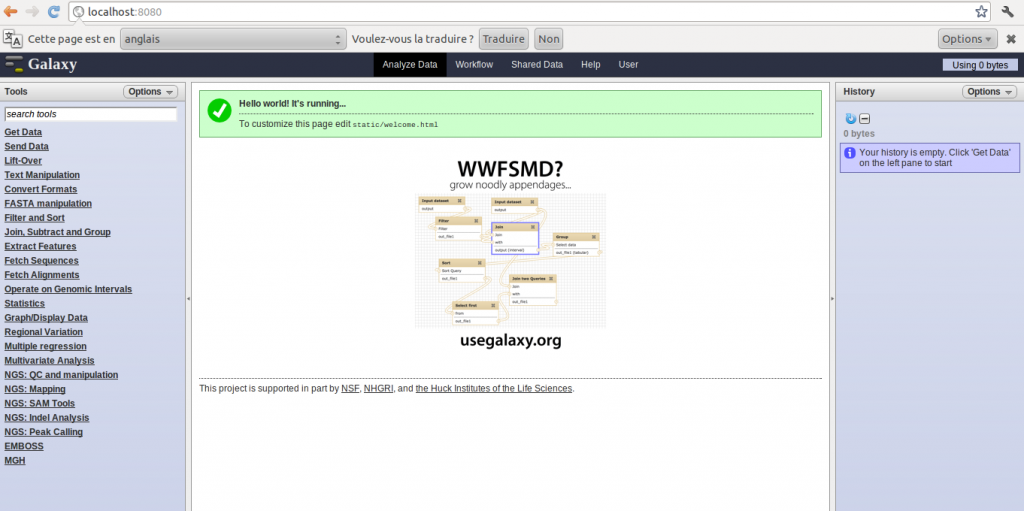
A ce stade, le programme devrait initialiser l’application.
Vous pouvez maintenant accéder à votre serveur Galaxy à l’adresse suivante : http://localhost:8080
Evidemment ce tutoriel est court et ne prend pas en compte la configuration plus fine de l’application. Pour plus de détails sur l’installation mais aussi sur l’utilisation je vous invite à lire (et à tester) les excellentes explications consultables sur le site de Galaxy :
http://wiki.g2.bx.psu.edu/Admin/Get%20Galaxy
http://wiki.g2.bx.psu.edu/Admin/Training/
Pour les (heureux) propriétaires d’un serveur de calcul :
http://wiki.g2.bx.psu.edu/Admin/Config/Performance/Cluster
Pour l’utilisation de Galaxy dans un environnement de production – ou si vous voulez directement installer votre serveur Galaxy en partant sur des bases solides :
http://wiki.g2.bx.psu.edu/Admin/Config/Performance/Production%20Server
Utilisation :
Il faut savoir que Galaxy en local fait souvent un lien vers les logiciels sans que ces derniers ne soient présents « physiquement » sur votre serveur. Ainsi le mapping avec BWA ne marchera pas, à moins que vous installiez au préalable bwa sur le serveur. C’est le cas de pas mal d’outils, donc si vous rencontrez parfois des erreurs lors de l’exécution, il est fort probable qu’il vous faudra d’abord installer les outils utilisés par Galaxy.
Un des inconvénients majeurs de la version en ligne concerne la limitation de chargement de jeux de données à 2 Gb. En séquencage haut-débit cette limite est très souvent atteinte. En ligne il est possible de contourner cette limitation en chargeant ses fichiers par FTP. Mais on ne peut éviter le problème du débit (temps de transfert long) et de la confidentialité des données.
En local, cette la limitation est également fixée à 2 Gb, et il y a également la possibilité de charger ces jeux de données directement dans le logiciel, sans passer par une phase de chargement (« upload ») en installant son propre FTP. Évidement en local, le débit est fortement accéléré.
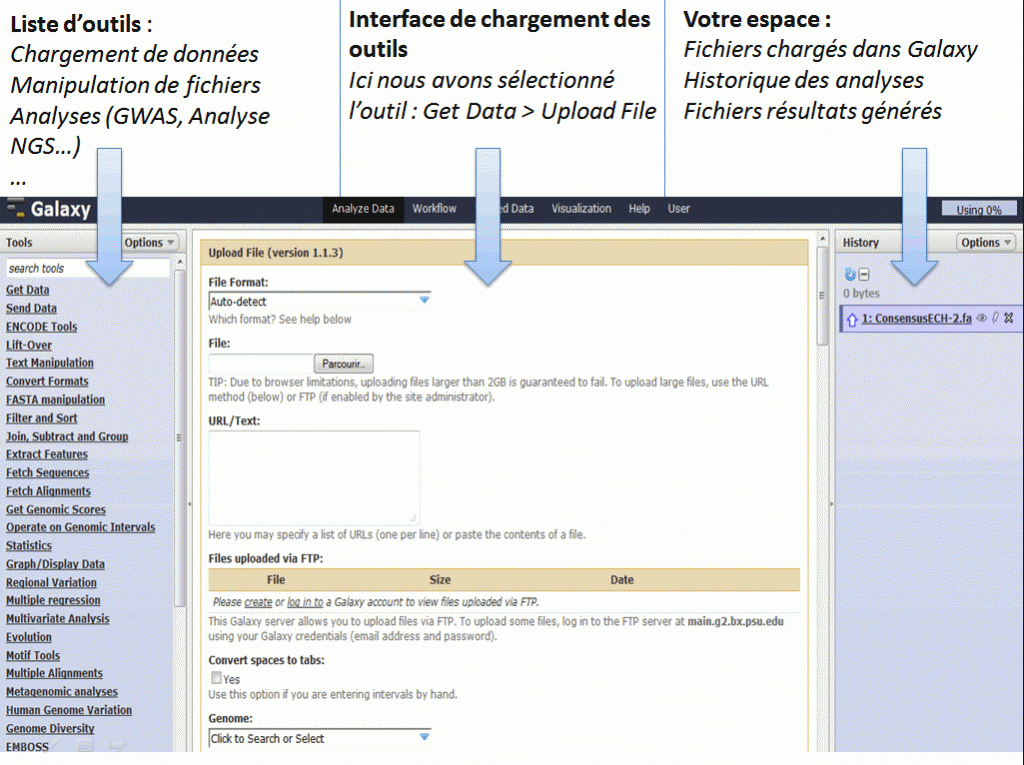
Pour ce faire, vous pouvez lire le tutoriel suivant en anglais ou suivre les indications ci-dessous :
http://wiki.g2.bx.psu.edu/Admin/Config/Upload%20via%20FTP
ou regarder la video tutoriel .
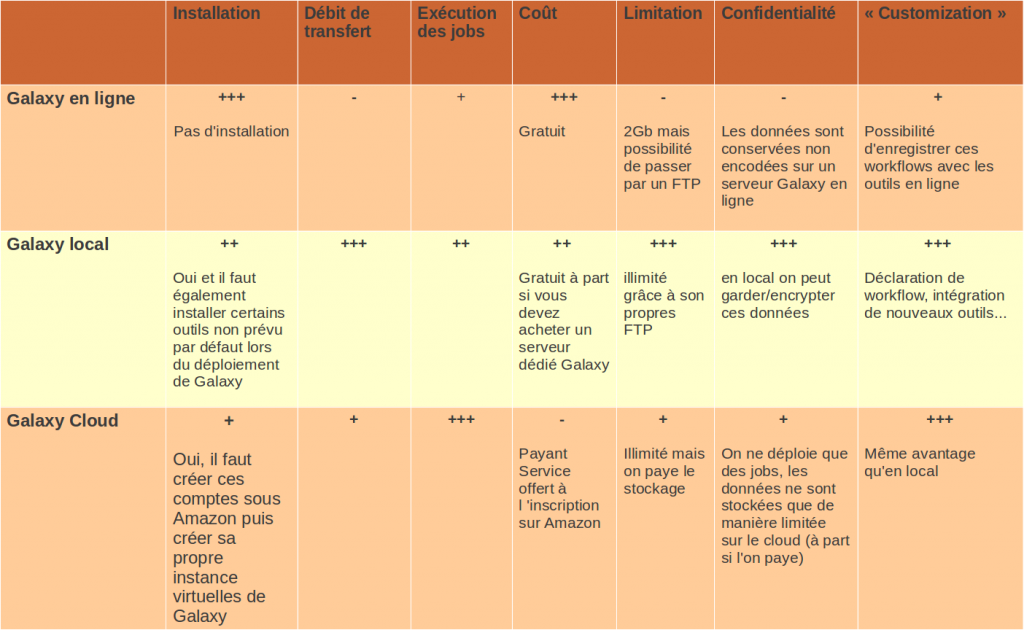
Comparaison entre les méthodes :
Pour résumer, l’installation en local offre plusieurs avantages par rapport à la version en ligne, en terme de confidentialité, de taille de données, de transfert et de temps d’analyses. Quelques inconvénients subsistent notamment la contrainte d’investir dans une machine correcte si l’on veut obtenir un temps d’analyse correct ou la nécessité d’installer certains outils extérieurement à Galaxy.
Si l’on supprime l’aspect coût et les contraintes lors de la première installation, la solution Cloud semble la plus pertinente si l’on a pas accès à des environnements matériels conséquents.
L’aspect « customisation » et particulièrement l’intégration de ses propres outils fera l’objet d’un prochain article.
 Cet article est l’occasion de mettre en avant un site plutôt fourni, SEQanswers… un jeu de mots pour une communauté d’utilisateurs de technologie de séquençage haut-débit. Ce site, un peu bouillon, fondé en 2007 par Eric Olivares (qui a travaillé pour Pacific Biosciences) s’adresse aux biologistes moléculaires plus qu’aux bio-informaticiens. Malgré tout, le lien que nous vous présentons ici, renvoie sur la partie Wiki du site SEQanswers. Cette page liste et ordonne en fonction de leurs domaines d’applications les logiciels (gratuits et commerciaux) utiles pour le devenir de vos reads produits par séquençage haut-débit : http://seqanswers.com/wiki/Software/
Cet article est l’occasion de mettre en avant un site plutôt fourni, SEQanswers… un jeu de mots pour une communauté d’utilisateurs de technologie de séquençage haut-débit. Ce site, un peu bouillon, fondé en 2007 par Eric Olivares (qui a travaillé pour Pacific Biosciences) s’adresse aux biologistes moléculaires plus qu’aux bio-informaticiens. Malgré tout, le lien que nous vous présentons ici, renvoie sur la partie Wiki du site SEQanswers. Cette page liste et ordonne en fonction de leurs domaines d’applications les logiciels (gratuits et commerciaux) utiles pour le devenir de vos reads produits par séquençage haut-débit : http://seqanswers.com/wiki/Software/
Cette page est plutôt bien renseignée et vous donnera un large choix de logiciels : vous y trouverez des assemblers de novo, des logiciels pour réaliser du RNAseq (quantification), des logiciels permettant de trouver des pics après ChipSeq… peu de logiciels indispensables sont absents de cette liste qui comporte un peu moins de 500 logiciels…
- des fiches logiciels (classement, descriptions et lien pointant vers l’hébergeur de ceux-ci)
- des fiches ressources (site internet, évènements, documents utiles…)
- mutualiser les compétences sur les logiciels
- promouvoir les développements internes
- animer une communauté autour du logiciel
- promouvoir l’usage des logiciels libres et la contribution à leur élaboration
La confusion entre mate-pair et paired-end, tant au niveau technologique (selon qu’on lise les notes techniques d’Illumina, de Roche ou de Life) que logiciel nous a mené à rédiger, en collaboration avec Ségolène Caboche, Bioinformaticienne à l’université de Lille2, une note technique dont le contenu est résumé ci-dessous :
– Genèse de la confusion entre mate-pair et paired-end
– Descriptions les deux approches, avec un focus sur les principales technologies de seconde génération de séquenceurs
– Traitement au niveau logiciel et conseils généralistes pour l’utilisation
Le document est consultable dans son intégralité sur notre blog :
Télécharger Paired-end versus mate-pair
Bonne lecture!
Il y a quelques semaines, nous avions discuté de l’utilisation de logiciel de Workflow pour la bioinformatique. Il est temps de passer à la pratique en vous présentant un de ses dignes représentants : Galaxy.
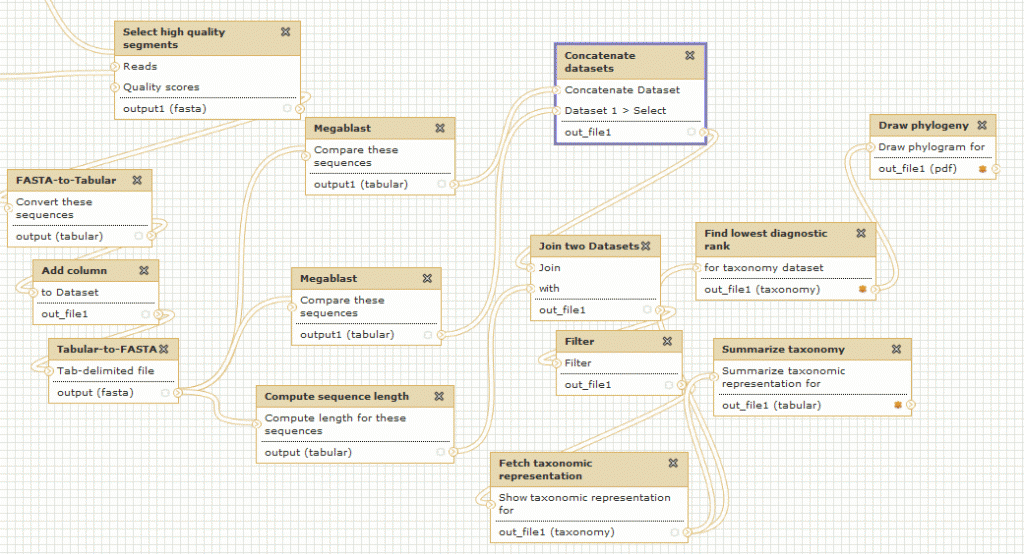
Le workflow Galaxy fournit un ensemble d’outils pour la manipulation et l’analyse de données génomiques. Il est très intuitif dans l’utilisation ce qui en fait une cible de choix pour le biologiste.
Il est possible d’utiliser Galaxy directement depuis le serveur. Avantage conséquent pour les bioinformaticiens il est possible d‘installer sa propre instance de serveur Galaxy, cette option fera l’objet d’un prochain post technique.
Du point de vue de l’interface graphique :
On peut également créer des workflows, les enregistrer dans un espace dédié, les partager, et les exécuter de façon automatique.
Pour exemple ce workflow de métagenomique publié gratuitement par un utilisateur de Galaxy (vous devez être connecté pour visualiser le workflow dans Galaxy)
Les outils dédiés analyse de données NGS sont régulièrement mis à jour et nul doute que d’ici peu, certains seront dédiés IonTorrent.
Les tutoriels sont également très bien faits, on apprend très vite à maitriser l’environnement grâce à des dizaines de vidéos d’aides.
Galaxy offre donc la possibilité d’exécuter des analyses bioinformatiques sans effort de programmation. La version en ligne est intéressante car elle permet de se familiariser aux logiciels et d’exécuter l’analyse depuis un portable, mais la possibilité d’intégrer ces propres outils (nous y reviendrons) est indéniablement un gros avantage de la version locale.
Si nous devions citer un inconvénient, plutôt d’actualité : l’utilisateur est obligé de charger ses données en mémoire dans Galaxy, le temps de chargement peut être très long si l’on manipule des données issues d’expériences NGS. D’autres workflows tels que Ergatis, fonctionnent en local et permettent à l’utilisateur d’utiliser directement les données présentent sur l’ordinateur.
Pour en savoir plus :
La description complète du logiciel Galaxy en Français sur PLUME :
http://www.projet-plume.org/fiche/galaxy
Le Galaxy Wiki :
http://wiki.g2.bx.psu.edu/FrontPage
La publication associée :
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages