Voici un lien sur le site Molecular Biology Freeware for Windows… un lien destiné aux biologistes un peu allergiques à Linux et qui ne souhaitent pas débourser des milliers d’euros dans des logiciels qui ne peuvent s’avérer utiles que sporadiquement (pour certains).
Le site Molecular Biology Freeware for Windows est particulièrement laid mais il permet de référencer ces logiciels utiles pour bon nombre d’applications : phylogénie, alignement de séquences, etc.
L’alliance un peu paradoxale du logiciel gratuit, fiable et utile sous Windows !
Ce site-référence est pour quelques uns d’entre vous… : il classe les logiciels outils de biologie moléculaire en fonction des catégories suivantes :
A. DNA, RNA and genomic analysis
B. Plasmid graphic packages
C. Primer design
D. Protein analysis
E. Viewing three dimensional structures
F. Alignments
G. Phylogeny
H. Miscellaneous
I. Graphic packages
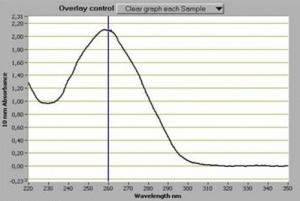
 Souvent perçu comme une évidence, le dosage d’acides nucléiques est un point fondamental. Il faut pourtant le considérer comme le premier « monitoring » important d’une expérience de biologie moléculaire. Que vous soyez équipés d’un NanoDrop™, d’un plus antique spectrophotomètre, d’un fluorimètre… les outils sont parfois complémentaires et ont des spécificités propres qu’il vous faudra évidemment connaître (au moins approximativement).
Souvent perçu comme une évidence, le dosage d’acides nucléiques est un point fondamental. Il faut pourtant le considérer comme le premier « monitoring » important d’une expérience de biologie moléculaire. Que vous soyez équipés d’un NanoDrop™, d’un plus antique spectrophotomètre, d’un fluorimètre… les outils sont parfois complémentaires et ont des spécificités propres qu’il vous faudra évidemment connaître (au moins approximativement).
En matière de dosage des acides nucléiques, il apparaît fondamental (si vous utilisez un spectrophotomètre) de connaître la fameuse loi de Beer où A=Ɛ.l.C
A étant l’absorbance (sans unité) et Ɛ est le coefficient d’absorption moléculaire (unité : L.mol -1 .cm-1)
l : la longueur du trajet optique (valeur fixe, donnée par l’épaisseur d’une cuve ou d’un ménisque sur un Nanodrop) (unité : cm)
C, souvent ce que l’on souhaite déterminer: la concentration d’acides nucléiques (unité : mol.L-1)
Cette formule permet d’estimer assez aisément une concentration en acides nucléiques, elle est vérifiée lorsque la solution est de concentration inférieure à : c < 0,1 mol.L-1
| A260 nm | ||
| acides nucléiques | Ɛ (M-1.cm-1) | pour 1 unité absorbance (µg.mL-1) |
| ADN db | 6200 | 50 |
| ADN ss | 8350 | 37 |
| ARN | 7700 | 40 |
| standard : température ambiante, l=1cm, A260/A280 =1,80 | ||
Assez rapidement, la concentration, le plus souvent en ng.µL-1, est déduite. Le point fort de la méthode spectrophotométrique (comparée à la méthode fluorométrique, plus sensible, quant à elle) est fournie par les informations que l’on tire des ratios suivants:
Ratio A260/A280 : un ratio de 1,80 (+/- 0,1) permet de qualifier une extraction d’ADN comme pure, ce ratio se situe autour de 2,0 pour une extraction d’ARN (+/- 0,1). Si ce ratio est inférieur cela signifie que l’extraction est polluée par des protéines et/ou des composés phénoliques
Ratio A260/A230 : la valeur attendue de ce ratio se situe entre 2,0 et 2,20. Si ce ratio est plus faible cela indique nécessairement qu’un composé absorbe à 230 nm. Ces composés peuvent le plus souvent être de l’EDTA, des sucres, et encore une fois le phénol et particulièrement le TRIzol reagent.
Ces valeurs de ratios peuvent parfois être négligées, cependant elles donnent une indication quant à la présence d’éventuels inhibiteurs qui viendraient perturber le devenir de vos acides nucléiques (PCR et autres réactions enzymatiques). Il est important de réaliser une valeur de blanc sur la solution qui vous aura servi à éluer vos acides nucléiques (car comme il a été précédemment mentionné l’EDTA absorbe à 230 nm… et bien souvent le TE 1X sert à reprendre et conserver des acides nucléiques)
vous trouverez ci-contre le mode d’emploi du Nanodrop 1000.
Une autre méthode alternative, permettant une meilleure sensibilité : méthode picogreen. Cette méthode, bien que très sensible, donne des résultats non valides (car très largement sous-estimés) dès lors que votre solution d’acides nucléiques est polluée par des traces d’agents détergents (type SDS). En effet, ces « adjuvants » quenchent la fluorescence émise par l’agent qui s’intercale au sein des acides nucléiques que l’on souhaite doser.
Note : bien souvent il vous est utile de calculer la quantité de molécules (quantité de copies).
Sachant que la masse molaire moyenne d’une base azotée => Masse Molaire (MM) : 309 g.mol-1
&
avec le nombre d’Avogadro 6,02214129.1023 molécules.mol-1
La formule devient : nombre de copies pour 1 µL = [C (concentration mesurée en ng/µL) x V (volume, ici 1 µL) x 10-9 ]/ [(MM environ 309 g.mol-1) x la taille de votre génome en pb x 2] le tout que l’on multiplie par le nombre d’Avogadro… le tour est joué !
plus simplement : 978 Mb pèsent 1 pg (en avant le produit en croix).
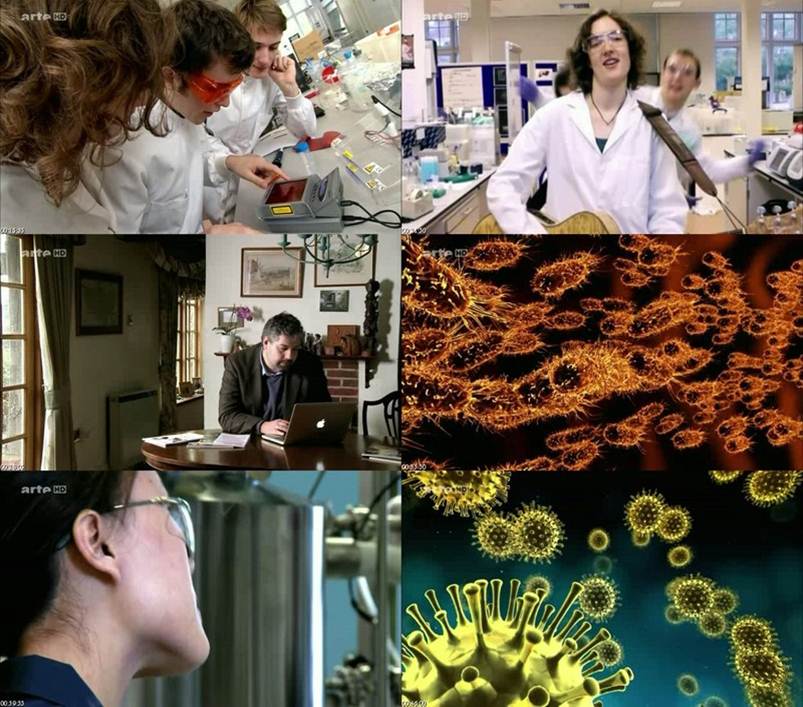
Voici un documentaire diffusé sur Arte qui aborde en un peu plus de 50 minutes la biologie synthétique. Laetita Ohnona, la réalisatrice du film, pondère les interventions des Craig Venter et autres ingénieurs du MIT, technoscientistes par plusieurs interventions d’un lanceur d’alerte, Jim Thomas, filmé devant une campagne rassurante qui contraste avec l’univers d’un laboratoire. Ce documentaire très pédagogique (à mes yeux, il s’agit de sa principale qualité), consacre les ingénieurs face aux chercheurs -de la recherche appliquée vers la recherche fondamentale, en quelque sorte. Ainsi des parallèles sont réalisés entre des Steve Jobs & Bill Gates, deux personnages ayant révolutionné les applications liées à l’informatique et des Randy Rettberg & Tom Knight (co-fondateurs du prix Igem), des ingénieurs-chercheurs ayant œuvré en informatique pour terminer leur carrière dans les biotechnologies.
L’expérience de Philippe Marlière est développée en fin de documentaire pour apporter un début de solution au contrôle des organismes synthétiques comme outils de production.
Ce documentaire avivera la curiosité des scientifiques en herbe attiré par l’image d’Épinal de biogeek construisant un biofermenteur dans le garage de papa, effraiera les sceptiques, et très certainement produira un peu ces deux effets sur les initiés. On pourra simplement regretter l’absence d’un regard bioéthique dans ce documentaire qui permettrait de prendre un certain recul, évitant les allers-retours entre fascination technologique et peur de fin du monde, un monde ravagé par des bactéries tueuses luminescentes…
Ci-dessous : le synopsis d’Arte concernant ce documentaire
Les spécialistes sont unanimes : la biologie connaît une révolution aussi décisive que la découverte de l’ADN ou le séquençage complet du génome humain. La biologie de synthèse doit permettre à moyen terme à l’homme de se faire démiurge, pour écrire des séquences partielles ou entières d’ADN – et donc créer ainsi artificiellement la vie. Une perspective tellement nouvelle qu’elle suscite des vocations en dehors des laboratoires eux-mêmes. Dans de simples garages ou des hangars, les « biohackers » sont aujourd’hui capables d’assembler des séquences d’ADN synthétiques (via des DNA cassettes ou biobricks disponibles sur Internet). Peut-être donneront-ils vie à de nouvelles cellules, voire à des organismes entiers. Certains experts posent la question sans détour : l’homme va-t-il, pour la première fois, prendre la main sur l’évolution naturelle des espèces ? Et si oui, quelles seront les conséquences pour les écosystèmes les plus fragiles, et l’équilibre des espèces vivantes ? De nombreux acteurs, dont les plus gros laboratoires, investissent en tout cas dans la biologie de synthèse, jusqu’à Bill Gates, qui finance un programme colossal contre la malaria reposant sur elle. C’est cette révolution en marche que ce film souhaite montrer pour la première fois, en partant à la rencontre de ceux qui la portent.
(France, 2012, 52mn)
ARTE
Date de première diffusion : Jeudi 26 avr. 2012, 22h25
Date(s) de rediffusion : Samedi 5 mai 2012, 10h45
Vidéo disponible sur : http://videos.arte.tv/fr/videos/fabriquer_le_vivant-6609804.html
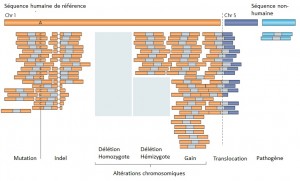
Le séquençage massif parallélisé palliant les limites des anciennes méthodes, a depuis ces dernières années amorcé de nouvelles perspectives, comme l’étude d’altérations génomiques pour la compréhension de nombreuses maladies rares (Plan National Maladies Rares). De plus, la mise sur le marché des derniers séquenceurs de paillasse aux visées hospitalières (positionnement de marché de Ion torrent avec son PGM) contribue également au développement de nombreux diagnostics cliniques.
Face à cette révolution, il convient de faire face au goulot d’étranglement de l’analyse et de l’interprétation, en faisant appel à une stratégie plus appropriée que le séquençage de génome complet et qui consiste à focaliser les efforts de décodage sur les régions codantes enrichies (Exome, gènes cibles, etc…) par une étape préliminaire de d’enrichissement d’exons. Tout en contribuant à une diminution des coûts (on ne séquence que ce qui semble pertinent…), elle permet entre autre d’augmenter la profondeur de séquençage des régions d’intérêts, paramètre indispensable pour l’identification d’altérations génomiques.
Succinctement, les techniques d’enrichissement sont multiples et peuvent être regroupées en trois catégories sur la base de leur principe technique:
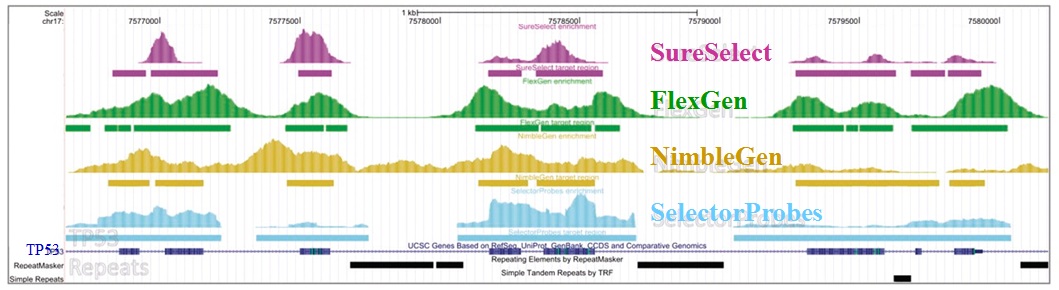
– Enrichissement par capture d’hybrides : La capture par hybridation des régions cibles est initiée à partir de la librairie de séquençage. Elle peut être effectuée soit en solution (billes magnétiques) soit sur support solide (microarrays). Récemment, de nombreuses solutions (commerciales ou « à façon ») ont été développées: SureSelect, Agilent – SeqCap EZ, Roche NimbleGen – Sequence Capture Arrays, Roche Nimblegen – FleXelect, FlexGen – MYselect, MYcroarray – TargetSeq, Life Technologies .
– Enrichissement par circularisation de fragments d’ADN : Cette méthode est supérieure à la précédente en terme de spécificité. En amont de la préparation de la librairie, les fragments d’ADNg (fragmentation enzymatique ou mécanique selon la méthode) sont enrichis via une sonde constituée d’une séquence universelle flanquée aux extrémités de séquences spécifiques de la région cible. HaloPlex, Agilent.
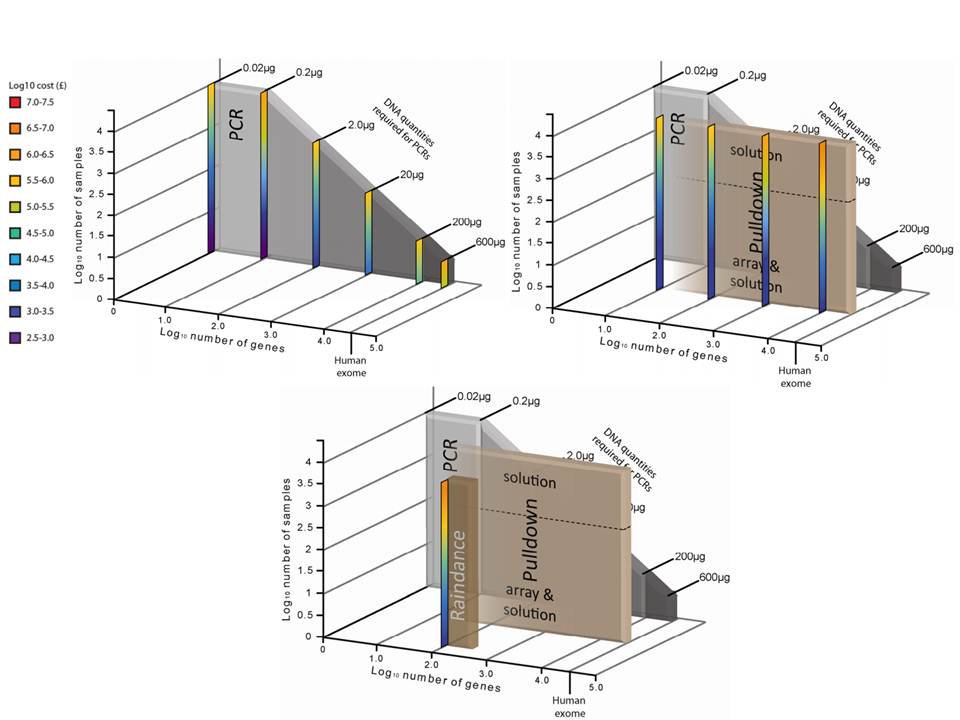
– Enrichissement par PCR : Cette approche intervient avant la préparation de la librairie et consiste grossièrement en une PCR multipléxée ciblant les régions d’intérêts. Une étape préliminaire de design des amorces est requise. SequalPrep, Life technologies. Tout juste disponible, Ion AmpliSeq Designer, Life Technologies propose un PCR Ultra-Plex (jusqu’à 1536 amplicons) avec un étape de digestion des amorces permettant de ne conserver que les régions d’intérêts lors du séquençage.
Une technologie récente et élégante faisant appel à la microfluidique et l’émulsion PCR permet l’amplification multipléxée (jusqu’à 20000) en micro-gouttelettes (une paire d’amorces par microréacteur) en un seul tube. Multiplicom, Fluidigm – RainDance.
Ceci étant, les considérations fondamentales dans l’analyse de variants résident notamment dans:
– la profondeur de séquençage
– l’homogénéité de couverture des régions d’intérêt
– la reproductibilité de la méthode
– la quantité d’ADN « Input » requis
– le nombre d’échantillons à traiter
– le coût global
L’étude récemment menée par Florian Mertes et al. (nov. 2011) souligne l’importance de ces notions et fait le postulat de performances de captures de régions cibles significativement différentes selon la méthode d’enrichissement employée.
Il est impossible d’identifier une méthode d’enrichissement comme étant la meilleure notamment parce que ces approches sont en continuelle évolution et amélioration. Chacune d’elles répondent à des caractéristiques particulières et à des applications distinctes.
Si l’enrichissement par circularisation offre davantage de spécificité mais moins d’uniformité, l’inverse est aussi valable pour l’enrichissement par capture d’hybrides. Parmi les considérations dans le choix d’une méthode, le nombre d’échantillons et la taille des régions cibles sont essentiels. Si la capture d’hybrides est privilégiée pour l’analyse de mégabases (étude de l’exome) avec un nombre limité de cas, cette méthode sera délaissée au profit d’une approche PCR multipléxée pour l’étude d’un nombre restreint de régions cibles de petite taille appliquée à un grand nombre d’échantillons (Diagnostic).
L’association des méthodes d’enrichissement au séquençage haut débit offre des capacités technologiques ouvrant la voie vers de nombreuses perspectives. Toutefois, si l’émergence des séquenceurs de troisième génération s’accompagne à nouveau d’une diminution des coûts de séquençage, on est en droit de se demander si cette option restera aussi attractive.

Cet article revient sur une publication de janvier 2012 de Science (voir image ci-dessous) qui exploite des données de biologie haut-débit pour répondre à deux questions biologiques liées : « comment a été permise la photosynthèse ? » – « toutes les plantes ont elles un ancêtre commun?«
Les auteurs exploitent des données génomiques et transcriptomiques d’un glaucophyte : Cyanophora paradoxa. Cyanophora paradoxa est le biflagellé le plus étudié des glaucophytes, pris pour modèle dans l’étude de l’endosymbiose et pour ce qui a trait à la fonction du proplaste à l’origine des chloroplates.
Ces proplastes ayant pour origine l’intégration par endosymbiose d’une cyanobactérie (les mitochondries quant à elles, ayant pour origine l’endosymbiose d’une alpha-protéobactérie). L’intégration de ces données apporte la preuve que toutes les plantes seraient issues d’un seul ancêtre commun: les plantes formeraient une lignée monophylétique.
Il y a plus d’un milliard d’années a eu lieu la rencontre entre une cyanobactérie et une cellule eucaryote : de cette rencontre a émergé des cellules capables d’exploiter la lumière solaire pour synthétiser de la matière organique. L’étude présentée ici distingue :
– les EGT (Endosymbiotic Gene Transfer) avec les échanges de gènes entre le génome de l’hôte et celui de la cyanobactérie initiale venant grandement enrichir le génome de cet hôte. La participation des gènes de cyanobactérie représenterait entre 6 % (pour Chlamydomonas reinhardtii) et 18 % (pour Arabidopsis thaliana). Chez C. paradoxa, 274 gènes semblent avoir une origine cyanobactérienne. L’étude de la séquence mitochondriale a permis de positionner les glaucophytes très près du point de divergence des algues vertes et rouges.
– une autre source d’apport de matériel génétique venant enrichir ce système : les transferts de gènes horizontaux (HGT). Les auteurs dénombrent 15 gènes qui sont partagés par les 3 taxons de plantes à l’origine d’une diversité de plantes, et qui auraient une origine bactérienne – des séquences proches de celles retrouvées chez des Chlamydiae et Legionella, des parasites intracellulaires de cellules eucaryotes.
Ainsi les gènes apportés par le parasite ont permis au couple cyanobactérie-cellule de fournir des gènes nécessaires aux échanges de « nourriture » (UhpC-type hexosephosphate transporters, des sous-unités de translocons). Cette étude démontre que ces échanges de gènes ont une signature unique chez les plantes, les algues et les glaucophytes confortant la thèse d’une lignée végétale monophylétique, un coup porté aux tenants de la thèse de la « paraphylie végétale ».
Cet article de Science est un bel exemple de l’exploitation, de l’intégration et enfin de l’interprétation donnant sens aux données génomiques et transcriptomiques générées à haut-débit (des données de transcriptomiques -réalisées sur la plateforme Illumina GAIIx- sont disponibles dans la banque du NCBI : Sequence Read Archive avec les n° d’accès suivants SRX104482, SRX104481, SRX104480).
La confusion entre mate-pair et paired-end, tant au niveau technologique (selon qu’on lise les notes techniques d’Illumina, de Roche ou de Life) que logiciel nous a mené à rédiger, en collaboration avec Ségolène Caboche, Bioinformaticienne à l’université de Lille2, une note technique dont le contenu est résumé ci-dessous :
– Genèse de la confusion entre mate-pair et paired-end
– Descriptions les deux approches, avec un focus sur les principales technologies de seconde génération de séquenceurs
– Traitement au niveau logiciel et conseils généralistes pour l’utilisation
Le document est consultable dans son intégralité sur notre blog :
Télécharger Paired-end versus mate-pair
Bonne lecture!
 Sequenom est une autre société californienne de biotechnologie (cotée au NASDAQ) basée à San Diego. Cette société développe des outils d’analyse génomique avec deux principales divisions:
Sequenom est une autre société californienne de biotechnologie (cotée au NASDAQ) basée à San Diego. Cette société développe des outils d’analyse génomique avec deux principales divisions:
– l’une d’elles développant des systèmes d’analyse génétique (type MassARRAY)
– l’autre se concentrant sur la recherche et développement de réactifs transférables sur la plateforme technologique développée par la première division.
Depuis 2008, Sequenom développe des outils analytiques de paillasse, les systèmes ‘MassARRAY’, appareillage compact permettant une mesure directe de la masse des acides nucléiques, offrant une précision inégalée quant à leur nature et leur quantification. Ce système, sur la base d’un spectromètre de masse, multi-MALDI-TOF MS, trouve des applications pour le génotypage, l’ analyse de la méthylation et l’expression génique. Sequenom continue d’affiner ces applications et de développer plusieurs nouvelles applications pour l’analyse de CNV (Copy Number Variation) par exemple.
Cette technologie est chahutée par les séquenceurs haut-débits de paillasse et autres systèmes permettant de génotyper une grande quantité d’échantillons tels que les systèmes proposés par Fluidigm. Le système MassARRAY bénéficie d’une sensibilité hors pair, les kits diagnostiques développés sur leur plateforme permettent à Sequenom de faire perdurer leur technologie. Les kits diagnostiques prénataux comme le MaternitT21 offre une alternative à l’amniocentèse pour diagnostiquer les trisomies 21, (en février 2012, Sequenom a lancé le MaterniT21 PLUS, permettant de diagnostiquer les trisomies 21, 18 et 13) de manière non invasive ciblant l‘ADN fœtal circulant dans le système sanguin maternel. Après des suspicions quant à un délit d’initié potentiel, des cadres de Sequenom sont surveillés par la SEC (Securities and Exchange Commission), de plus le développement des kits diagnostiques prénataux a connu des difficultés en 2010. En effet, la société a été condamnée à verser 14 millions de USD pour régler un recours (en « class-action ») : des actionnaires se sont retournés sur la société considérant que le développement du kit diagnostique de la trisomie 21 avait été plus que mal géré (notamment au niveau des essais cliniques).
La technologie MassARRAY est encore très employée pour le passage de cohortes suite à des études d’associations génétiques (les puces haute-densité sont employées dans un premier temps, suivi du passage des loci candidats sur une plateforme plus souple, telle que le MassARRAY, permettant d’interroger les mutations putativement associées au phénotype étudié). Un document en lien ici, reprend les principales caractéristiques de la plateforme de génotypage.
Bien que la plateforme MassARRAY trouve vite ses limites, la société Sequenom s’emploie à développer des solutions diagnostiques qui retardent l’obsolescence de leur technologie. La société a connu des soubresauts économico-judiciaires dont elle a su se relever, elle tente d’accélérer la diffusion de plusieurs de ces kits diagnostiques avant que sa technologie ne soit dépassée par la concurrence.
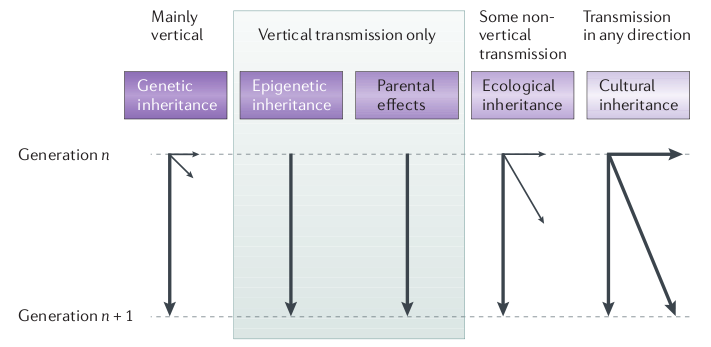
Pierre Barthélémy, journaliste scientifique au monde, a publié hier sur son blog (passeurdesciences.blog.lemonde.fr) une interview de Etienne Danchin (directeur de recherche CNRS et directeur de l’EDB) qui nous parle de la nécessité de s’extraire du dogme du « tout-génétique » pour s’intéresser plus précisément à d’autres mécanismes qui régissent l’hérédité, tel que l’épigénétique, l’hérédité culturelle et environnementale.
Vous pouvez retrouver l’interview dans son intégralité ici :
Cette interview fait suite à la publication d’une synthèse sur l’hérédité non génétique et son rôle dans l’évolution parue dans Nature Reviews Genetics en Juillet 2011 que vous pouvez consulter en intégralité ici.
Etienne Danchin n’est pas le seul à s’interroger sur la part réelle de la génétique dans les mécanismes de l’hérédité, nous vous conseillons une nouvelle fois l’excellente émission de Jean Claude Ameisen (à écouter dans ce post) qui aborde ces aspects de la transmission génétique et non génétique, entre générations.
L’hérédité culturelle est une composante en général peu discutée (notamment dans le domaine animal): quelle est la part de variation phénotypique qui peut être expliquée par une transmission sociale (ou transmission par apprentissage)? A noter que le rôle de la composante culturelle dans l’hérédité a été formalisé dans les années 1970, ça ne date donc pas d’hier, mais comme toutes les composantes non génétiques impliquées dans l’hérédité elle s’est retrouvée minimisée, voire ignorée, au profit de la composante génétique.
Quoi qu’il en soit, à l’heure de l’arrivée de la 3eme génération de séquenceur haut-débit et de la génération toujours plus astronomique de données génomiques, il est toujours bon de rappeler que le seul transfert de gènes n’explique qu’une proportion des mécanismes étudiés.
Pierre Barthélémy, journaliste scientifique à publié hier sur son blog (passeurdesciences.blog.lemonde.fr) une interview de Etienne Danchin (directeur de recherche CNRS) qui nous parle de la nécéssité de sortir du dogme du « tout-génétique » pour s’intérésser plus précisement à d’autre mécanisme qui régisse l’hériédité, l’épigénétique, l’hérédité génétique et environementale.
Vous pouvez retrouver l’interview dans son intégralité ici :
Cet interview est le résultats d’une synthèse sur l’hérédité non génétique et son rôle dans l’évolution publié dans Nature Reviews Genetics en Juillet 2011 que vous pouvez consulter en intégralité ici.
Etienne Danchin n’est pas le seul à s’intéroger sur la place réelle de la génétique dans les mécanisme de l’hérédité, nous vous conseillons une nouvelle fois l’excellente émission de Jean Claude Ameisen (à écouter dans ce post).
La HRM est une technique accessible (en temps/finance) sous diffusée. Loin d’avoir l’ambition de réparer cette injustice (à mes yeux), cet article vise à aborder une technique analytique de biologie moléculaire survolée, méconnue et snobée à l’heure de la biologie haut-débit.
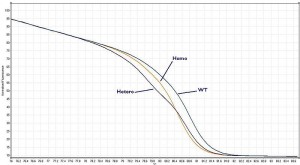
La HRM (High Resolution Melt, Courbe de Fusion à Haute-Résolution) permet de distinguer des amplicons (produits PCR) variant d’un minimum de 1 base… (avec plus ou moins de difficulté selon que la mutation concernée est une transversion A/T ou un INDEL par exemple). Cette technique est l’alliance de machine qPCR permettant un contrôle fin de la température (pour ce qui nous concerne nous sommes les heureux possesseurs d’un RotorGene 6000, de Corbett dorénavant vendu sous le nom de Rotor-Gene Q, Qiagen) et d‘agents intercallants de l’ADN saturants et moins « toxiques » pour la PCR comme peut l’être l’EvaGreen. Au rayon des molécules fluorescentes pour cette application vous pourrez trouver le LC Green et LC Green Plus , le ResoLight, Chromofy, SYTO 9 et le SybrGreen I bien sûr ! (de toute évidence sur la plateforme RotorGene, cette molécule permet de bons résultats). Ainsi que le montre les courbes ci-dessous, après amplification, les produits de PCR sont chauffés jusqu’à complète dénaturation par pas de température les plus fins possibles, plus ceux-ci sont petits, plus la technique est résolutive. Le point d’inflexion (l’endroit où la dérivée s’annule) reporté en abscisses est le Tm du produit PCR préalablement amplifié. Or le Tm d’un produit PCR est dépendant de sa concentration, de la concentration en sels du milieu dans lequel il est solubilisé mais surtout de sa composition en bases azotées.
L’hétéroduplexe (la forme hétérozygote) montrera une courbe avec 2 points d’inflexions (ainsi que vous pouvez l’observer sur la courbe ci-dessus). Les 3 populations : homozygotes AA et BB, ainsi que la forme hétérozygote AB se distinguent entre elles.
Au niveau des stratégies de design puis d’analyse, un document plutôt bien réalisé est disponible ici. Les plateformes permettant de réaliser de « vraies » courbes HRM possèdent un logiciel permettant une normalisation qui facilite grandement l’interprétation des résultats.
Les applications de cette techniques sont évidentes :
– le génotypage à bas débit et très bas coût : de simples amorces suffisent (nul besoin de sondes internes doublement marquées) un mastermix contenant un fluorochrome et le tour est joué (il sera judicieux d’avoir sous la main des échantillons références homozygote des deux formes, au minimum). Bien designé, ce diagnostic de génotypage permet une discrimination des substitutions A/T (SNP de classe 4) les plus difficiles à distinguer compte tenu de la faible différence de Tm (autour des 0,2 °C) entre les deux formes.
– le criblage (screening) de mutants : cette application permet de réaliser un premier filtre en vue d’un séquençage (par exemple) sur des zones courtes de l’ADN potentiellement impliquées dans un phénotype particulier. La reproductibilité et la sensibilité de la technique est correcte comme le montre l’article Tù Nguyen-Dumont et al., BMC 2011
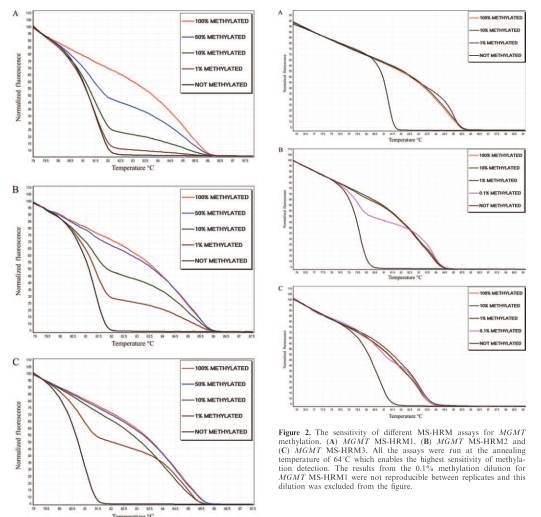
– méthylation de l’ADN : recherche de base 5-méthylcytosine (à l’aide d’un protocole bi-sulfite permettant de « remplacer » les cytosines non méthylées par des uraciles) à l’instar de ce que développe la publication dans NAR de Tomasz K. Wojdac et Alexander Dobrovic. Cette étude montre que l’intermédiaire de la MS-HRM (Methylation-Sensitive High Resolution Melting) il est possible de distinguer des zones méthylées à 0,1 % (ainsi que les courbes tirées de l’article sus-cité montrent pour des sensibilités descendant jusqu’à 1% d’amplicons méthylés, visibles ci-dessous).
En conclusion, la HRM est une technique largement méconnue et sous-employée, relativement simple à développer. Il est nécessaire de normaliser la concentration des échantillons (afin que ceux-ci aient des courbes d’amplification franchissant le seuil autour des 25 Ct). Dans l’idéal, la concentration en sels ne doit pas varier entre les échantillons… appuyé sur un bon design… et le tour est joué. De plus amples informations sont disponibles sur le site gene-quantification.info (la référence en matière de qPCR) ainsi que sur le site highresolutionmelt.co.uk.
Dernier né de la gamme des séquenceurs de paillasse, le « Ion Proton Sequencer » (149.000 $) est le premier de sa catégorie à permettre le séquençage en quelques heures du génome humain et à faible coût (1.000 $). A l’instar du PGM Ion Torrent, cette nouvelle plateforme est basée sur la technologie des semi-conducteurs (et la détection de la libération d’ions H+ suite à la polymérisation de dNTP).
La puce « Ion Proton ™ I »,disponible mi-2012, aura une capacité de séquençage 1000 fois supérieure au premier format de puce 314 du PGM Ion torrent soit 10 Gb (davantage orientée pour le séquençage d’exome humain). Le second format (puce « Ion Proton ™ II ») , disponible environ six mois plus tard aura une capacité de 100 Gb (Séquençage Génome humain).
Compte tenu de l’apparente similarité entre les deux plateformes de chez Life technologies, l’évolution réside essentiellement dans l’architecture de la puce avec une augmentation du nombre de puits ( 165 millions de puits pour la puce « Ion Proton ™ I » et 660 millions pour la puce « Ion Proton ™ II » soit respectivement 100 et 1000 fois plus que la puce 314 du PGM ), l’une des bases de la capacité de séquençage (en complément de la longueur des reads générés).
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages