Voici l’arrivée et la mise en exploitation du premier Ion Proton (Life Technologie) au Baylor College of Medicine à Houston.
36 heures entre la réception et la génération des premières données (avec des préparations de librairies réalisées en amont de la réception de la machine…).
Une présentation assez sympathique et ludique de la machine de Jonathan Rothberg… en avant première.
 Le représentant européen des séquenceurs haut-débit de troisième génération, Oxford Nanopore va communiquer à l’AGBT… dans quelques jours. La société en profitera pour annoncer la commercialisation de ses premières machines permettant de séquencer en 24 heures un génome humain pour moins de 1000 $ (soit le même prix qu’un génome humain séquencé par un Ion Proton en 2 heures sur une plateforme de deuxième génération).
Le représentant européen des séquenceurs haut-débit de troisième génération, Oxford Nanopore va communiquer à l’AGBT… dans quelques jours. La société en profitera pour annoncer la commercialisation de ses premières machines permettant de séquencer en 24 heures un génome humain pour moins de 1000 $ (soit le même prix qu’un génome humain séquencé par un Ion Proton en 2 heures sur une plateforme de deuxième génération).
Oxford Nanopore souhaite commercialiser sa machine (en direct) avant la fin 2012. Un réelle course a lieu, puisqu’à cette échéance sera commercialisé le Ion Proton. Nous assisterons donc avant fin 2012 a un chevauchement technologique où 2ème et 3ème générations de séquenceurs risquent de se télescoper… les scientifiques en cours d’élaboration de dossiers de demandes de financements devront (vite) choisir leur camp… : partir sur une technologie qui a d’ores-et-déjà fait ses preuves (Ion Proton) donc de 2ème génération ou une technologie de 3ème génération totalement innovante… mais dont on ne possède pas de retour indépendant sur la qualité des données générées. La « guerre » technologique est lancée entre le transfuge de Solexa (technologie des séquenceurs Illumina), John Milton, le responsable scientifique d’Oxford Nanopore et la biotech’ rock-star Jonathan Rothberg, responsable scientifique de Ion Torrent chez Life Technologies.
A Davos en Suisse (dans la patrie de Roche) au forum économique mondial, Jonathan Rothberg présente son séquenceur Ion Proton, une évolution de son PGM de Life Technologies. La dépêche AFP (mise en forme par 20minutes est disponible en cliquant sur l’image ci-dessous) est dithyrambique replaçant la machine de Life Technologies dans le contexte et le champ d’application de la médecine personnalisée… jouant au passage du violon de l’affect. Le Ion Proton, fort de la démonstration menée par son petit frère le PGM depuis un an (200 machines PGM en Europe !) s’appuie sur l’efficience du séquençage à l’aide de semi-conducteurs.
Il parait évident que la médecine personnalisée aura un futur au sein des pays développés… mais il ne faut pas oublier qu’il s’agit d’un futur que l’on augurait aux puces à ADN, il y a plus de 10 ans, sans réelle démocratisation au sein des cliniques. Avant toute chose, permettre de séquencer l’équivalent d’un génome humain ou de 2 exomes humains pour la somme soutenable de 1000 $ (en 2 heures) laisse entrevoir une accélération des recherches biomédicales mais aussi de toutes les autres ! Cet outil qui torpille un MiSeq (Illumina) sorti trop tard, diffusera dans les laboratoires… il remporte, malgré ses défauts d’ores et déjà connus, la bataille du séquençage 2ème génération. Le prochain pari (plus aléatoire) porterait plutôt sur le séquençage 3ème génération dont la promesse est une lecture d’une molécule d’ADN ou d’ARN (non amplifiée artificiellement) dans son « état » originel (méthylé ou non par exemple). La 3ème génération unifie génomique et épigénétique.
Le Ion Proton devrait être livrable à partir d’octobre 2012, quelques chanceux pourront malgré tout réaliser des runs dès le milieu de l’année (il faut en acheter un minimum de 4 pour être un parmi ces privilégiés).
Un Ion Proton devrait avoir un coût proche de 140 k€ auxquels il vous faudra ajouter 65 k€ de serveur informatique.
Dernier né de la gamme des séquenceurs de paillasse, le « Ion Proton Sequencer » (149.000 $) est le premier de sa catégorie à permettre le séquençage en quelques heures du génome humain et à faible coût (1.000 $). A l’instar du PGM Ion Torrent, cette nouvelle plateforme est basée sur la technologie des semi-conducteurs (et la détection de la libération d’ions H+ suite à la polymérisation de dNTP).
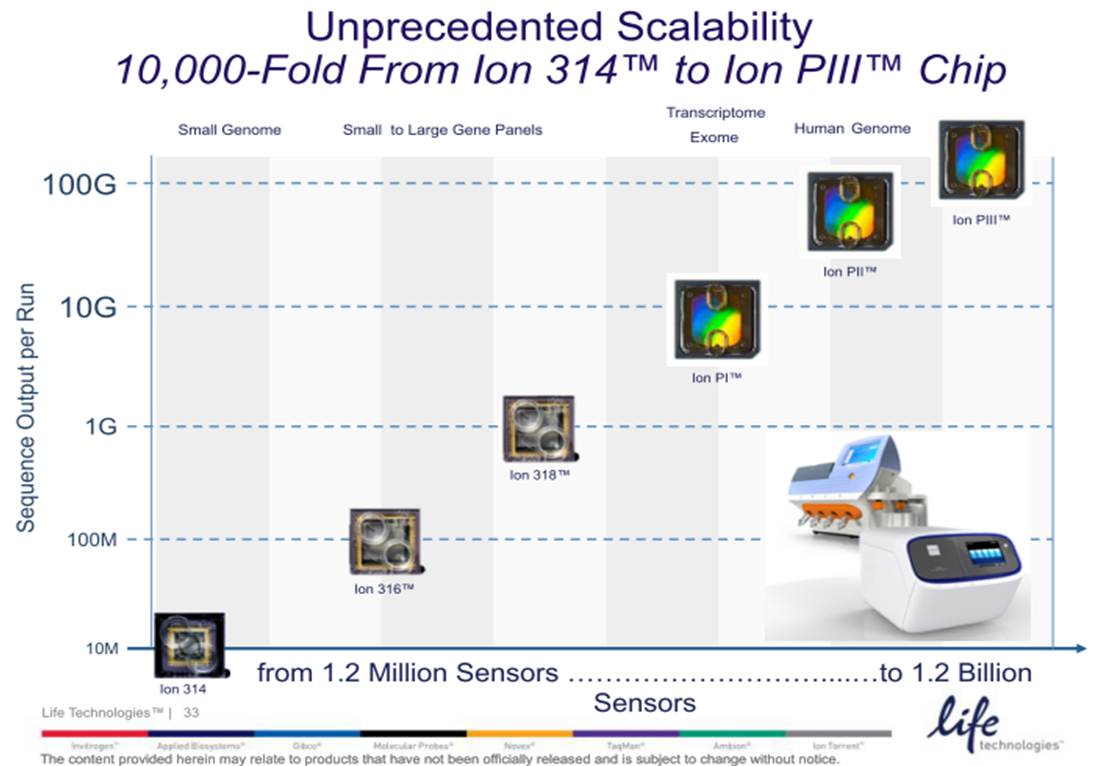
La puce « Ion Proton ™ I »,disponible mi-2012, aura une capacité de séquençage 1000 fois supérieure au premier format de puce 314 du PGM Ion torrent soit 10 Gb (davantage orientée pour le séquençage d’exome humain). Le second format (puce « Ion Proton ™ II ») , disponible environ six mois plus tard aura une capacité de 100 Gb (Séquençage Génome humain).
Compte tenu de l’apparente similarité entre les deux plateformes de chez Life technologies, l’évolution réside essentiellement dans l’architecture de la puce avec une augmentation du nombre de puits ( 165 millions de puits pour la puce « Ion Proton ™ I » et 660 millions pour la puce « Ion Proton ™ II » soit respectivement 100 et 1000 fois plus que la puce 314 du PGM ), l’une des bases de la capacité de séquençage (en complément de la longueur des reads générés).
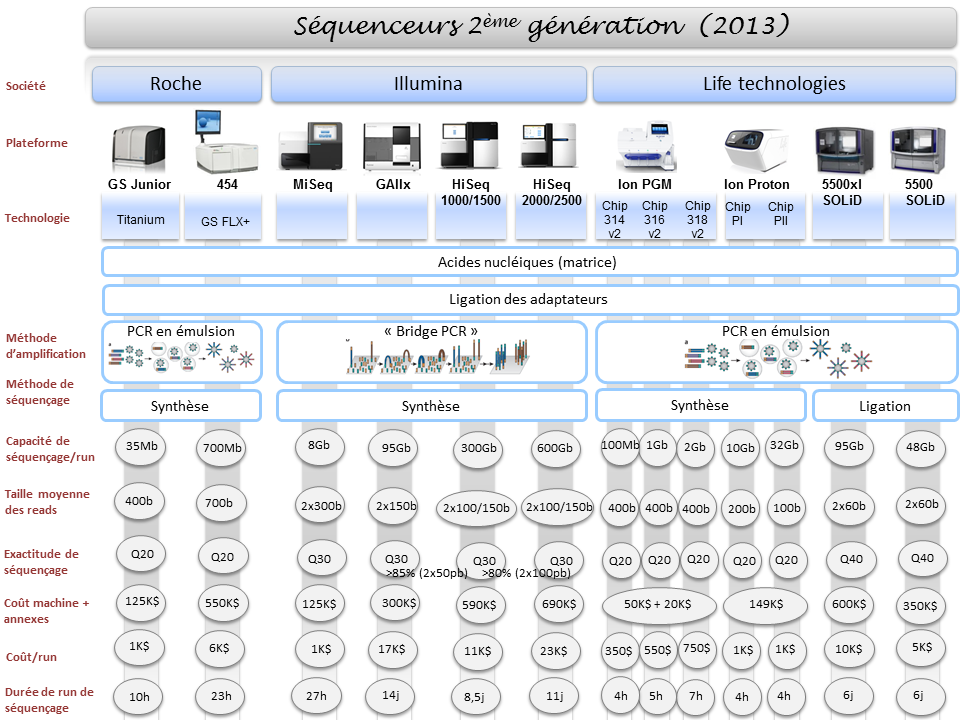
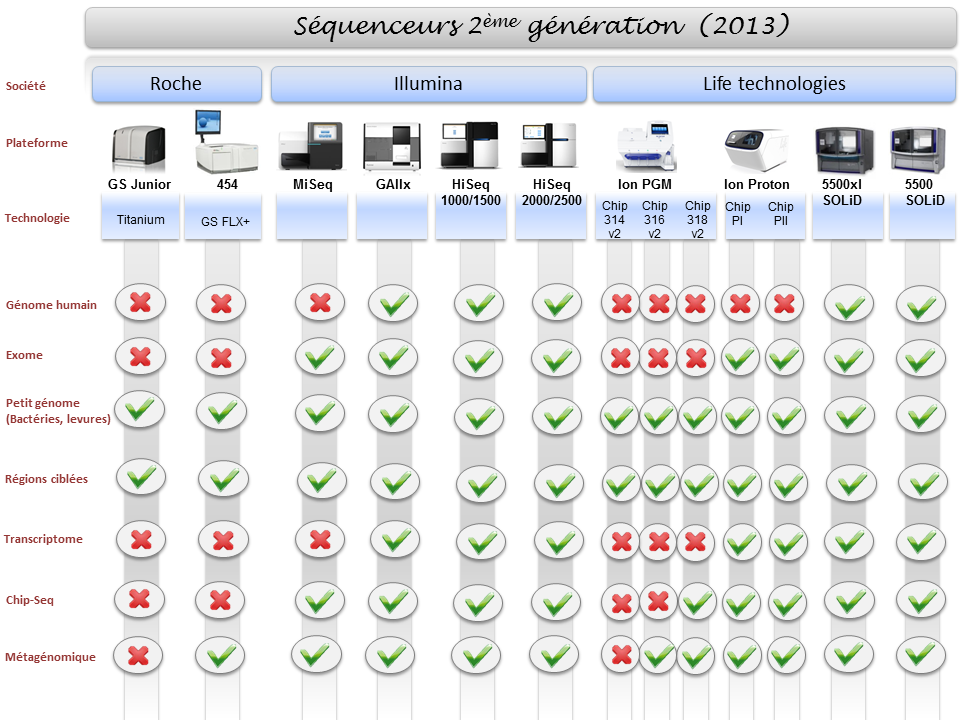
La diversité du parc technologique des séquenceurs de deuxième génération n’est plus une surprise pour personne. Ceci étant, il devenait indispensable de mettre à jour l’ensemble des informations postées sur ce site, il y a exactement deux ans (2011), faisant un état de l’art des différentes caractéristiques technologiques des séquenceurs, ainsi que les possibles applications biologiques associées.
Pour ne mentionner que les trois plus gros fournisseurs du marché, les sociétés Roche, Illumina et Life Technologies n’ont cessé de faire évoluer leur gamme, tant sur le plan des équipements que sur le plan des capacités de séquençage.
Par voie de conséquence, ce survol est l’occasion de refaire le point sur les technologies appropriées selon l’application biologique recherchée. A noter que le Ion proton, dernier en date sur le marché des séquenceurs de deuxième génération disposera au cours de l’année 2014 d’une puce « PIII » permettant de générer environ 64Gb. Cette capacité de séquençage permettra à Ion torrent de se positionner sur le séquençage de génome humain à partir d’un séquenceur de paillasse et accèdera ainsi à la gamme complète des applications citées ci-dessous.
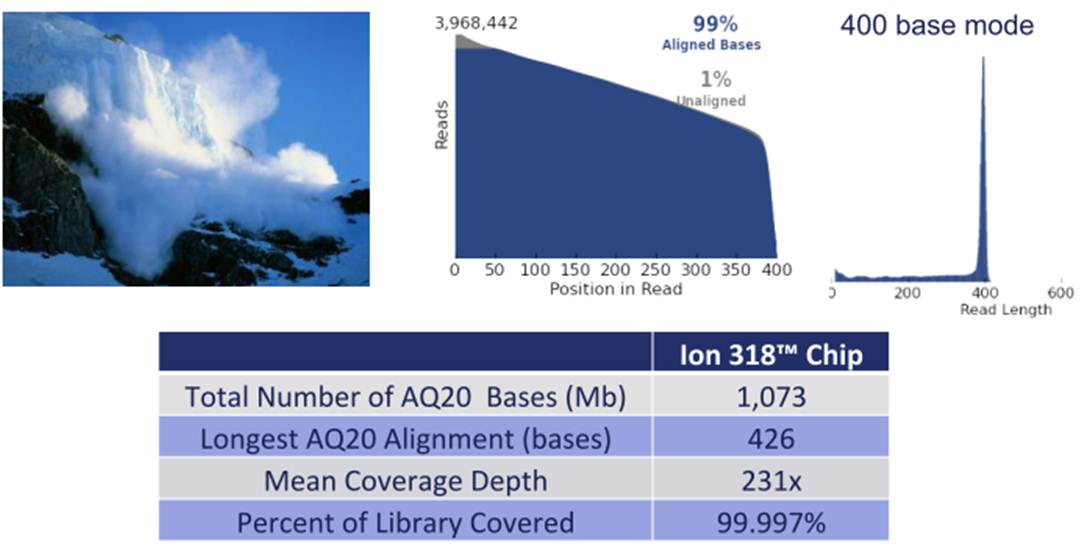
 Le mois de décembre 2012 a connu le lancement du kit 400 bases (
Le mois de décembre 2012 a connu le lancement du kit 400 bases (
Ion PGM™ Template OT2 400 Kit) qui est principalement attendu pour les applications de séquençage de novo et de séquençage d’amplicons ciblant le 16S, 18S ou encore ITS2 (respectivement pour l’étude de la diversité bactérienne, eucaryote ou fongique) pour les études métagénomiques. Comme vous pouvez le constater cette application est uniquement supportée par le One Touch 2 devenu nécessaire pour accéder aux reads de 400 bases (c’est en partie pour cette raison que notre plateforme PEGASE-Biosciences a décidé de se doter de cette automate).
L’autre nouveauté est portée par les consommables de séquençage associés au Ion Proton (le grand frère du PGM). En effet, si la Puce Ion PIII est encore virtuelle, la PI et PII, permettent de délivrer : 10 Gb et 70 millions de reads filtrés environ pour la première, contre plus de 280 de millions de reads et plus de 70 Gb pour la seconde (de quoi séquencer un génome humain à 20 X).
Du côté de Life Technologies et de sa technologie Ion Torrent, sans aucun doute, la principale nouveauté de l’année 2013 touche l’amplification clonale (phase où, à partir de la librairie préparée, est réalisée la matrice de séquençage fixée de manière covalente sur des billes qui se logeront dans les puits présents sur la puce). Ainsi, la fameuse (et pour certains la fâcheuse) phase de PCR en émulsion trouvera une alternative avec la chimie isothermique dite « avalanche » (dont vous pouvez voir l’aperçu d’un run ci-dessous). A en croire Life Technologies, cette chimie ouvre la voie à des tailles de séquences générées encore plus longues.
Bien entendu, Life Tech annonce l’arrivée d’un nouvel automate permettant d’intégrer la nouvelle chimie mais aussi de charger les puces (et pour quelques dizaines de k€, d’épargner au manipulateur les deux phases délicates du séquençage).
Pour conclure brièvement, l’une des nouveautés et non des moindres : l’acceptation du PGM comme diagnostic moléculaire CE-IVD (un parcours pas vraiment de tout repos) a n’en pas douter une conformité qui ne devrait pas laisser insensible les chercheurs, praticiens hospitaliers.
 L’AGBT qui a eu lieu du 15 au 18 février, à Marco Island, a fait la part belle à la technologie d’Oxford Nanopore, ainsi qu’il avait été prévu. La société dont il est question a profité du rassemblement pour lever le voile sur 2 produits : le MinION et le GridION, il est à noter que le MinION, mini-système de séquençage de la taille d’une grosse clé USB (photo ci-contre) a une dénomination commerciale tout particulièrement adaptée au marché français.
L’AGBT qui a eu lieu du 15 au 18 février, à Marco Island, a fait la part belle à la technologie d’Oxford Nanopore, ainsi qu’il avait été prévu. La société dont il est question a profité du rassemblement pour lever le voile sur 2 produits : le MinION et le GridION, il est à noter que le MinION, mini-système de séquençage de la taille d’une grosse clé USB (photo ci-contre) a une dénomination commerciale tout particulièrement adaptée au marché français.
La technologie d‘Oxford Nanopore a été évoquée dans plusieurs de nos articles. Elle permet le séquençage et l’analyse à haut-débit de reads de taille ultra longue (plusieurs kb) en temps réel pour pas très cher : la promesse d’un séquençage de 3ème génération démocratisé. Clive G. Brown (directeur de la technologie chez Oxford Nanopore) a présenté ses deux nouveaux jouets :
– Le MinION (dont le prix serait inférieur à 900 $) est un consommable et séquenceur (les deux à la fois) jetable qui devrait permettre de générer 1 Gb de données
– Le GridION (que vous pouvez empiler à foison, voire photo ci-dessous) permet quant à lui de générer, par module, plusieurs dizaines de Gb / jour (on pencherait pour un minimum de 25 Gb) sachant que selon nos informations un module aurait un coût voisin de 30 k$. Oxford Nanopore insiste sur le fait qu’à la Gb générée ils seront concurrentiels en terme de coût des consommables. En outre, le volume de données générées s’adapte à la problématique de l’utilisateur puisqu’en effet tant que l’appareil séquence -d’où leur slogan « Run Until« – il génère des données (le débit journalier associé à une technologie prend tout son sens ici). La cartouche –consommable de séquençage– associée à la technologie GridION possède actuellement 2000 pores individuelles -en 2013, il est prévu de passer à un consommable en comportant 8000- avec cette évolution il sera donc possible avec 20 modules GridION (environ l’investissement équivalent à un HiSeq2000) de séquencer un génome humain en 15 minutes ! Une autre façon de voir les chose est la suivante, dans sa version « actuelle » à 2000 pores disponibles : pour un prix équivalent à celui d’une configuration de type Ion Proton, 5 modules GridION seront capables de séquencer un génome humain à 30 X (cela leur prendrait une demie journée).
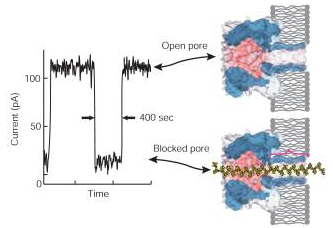
D’autres éléments ont filtré lors de l’AGBT. En effet, il semblerait que la technologie d’Oxford Nanopore subisse un taux d’erreurs sur séquences brutes encore assez élevé de 4 % (comparé aux plus de 10 % pour la technologie de Pacific Bioscience). Clive G. Brown aurait laissé entendre que ce taux d’erreurs serait uniforme et le fruit d’une majorité d’erreurs systématiques (ce qui est plutôt bon signe, en vue d’une rapide amélioration du système). Au niveau du système de détection, une puce GridION comporte 2K capteurs (un par pore). Chaque capteur permet de distinguer 64 signaux différents, ceci a permis d’analyser le passage de triplets de base afin de pouvoir discriminer 4x4x4 profils différents (j’avoue que j’attendais plus : quid de la prise en compte d’un signal différentiel en cas de présence d’une 5-méthylcytosine ?).
Au niveau préparation des échantillons, un séquenceur de 3ème génération ne nécessite pas de phase d’amplification. Hormis une phase préalable de légère fragmentation de l’échantillon rien ne semble envisagé. Pour palier leur problème de fiabilité, on imagine aisément qu’à l’instar de Pacific Bioscience, une circularisation de l’ADN de l’échantillon permettra d’engendrer en séquençage un nombre suffisant de répétitions venant atténuer ce point négatif.
Un élément important -mais pas surprenant- réside dans la politique commerciale affichée : une distribution directe des machines ainsi qu’une adaptation tarifaire (en usant de forfaits) offrant la possibilité d’acheter la machine à prix réduit avec un report sur le coût des consommables devrait permettre à Oxford Nanopore de conquérir quelques marchés n’en doutons pas !
Ce post fait naturellement suite à celui dédié à la seconde génération de séquenceurs multi-parallélisés, et conserve la même approche, à savoir un tour d’horizon des technologies et une évocations des informations générales sur le sujet.
A l’instar du PGM de Ion torrent mis sur le marché depuis un an (10Mb – reads 100b – 06.2011 / 100Mb – reads 200b -11.2011 / 1Gb – reads 400b – prévu début 2012), la seconde génération de séquenceurs haut débit tend vers une production de reads de plus en plus longs et de moins en moins chère. Toutefois, on est en droit de se demander quelle sera leur pérennité face à la 3éme génération répondant à un cahier des charges assez similaire et la possibilité de bénéficier de nouvelles applications.
Le principe de la 3ème génération peut être symbolisé par le séquençage d’une molécule d’ADN sans étape de pré-amplification (contrairement à la génération actuelle type 454 Roche, SOLiD Life technologie, Ion Proton, PGM Ion torrent, HiSeq Illumina, …) en conservant l’incorporation de nucléotides, par cycles ou non ( dans ce dernier cas, le terme de « Séquençage d’ADN simple molécule en temps réel » est approprié).
Les technologies « SMS » pour « Single Molecule Sequencing » peuvent être regroupées selon trois catégories:
– Technologies de séquençage en temps réel impliquant la synthèse du brin d’ADN complémentaire via une ADN polymérase.
– Technologies de séquençage par détection des bases successives d’une molécule d’ADN au travers de nanopores.
– Technologies de séquençage basées sur des techniques de microscopie.
En combinant les dernières avancées dans la nanofabrication, la chimie de surface et l’optique, Pacific Biosciences (Pacbio RS) a lancé une plateforme technologique puissante appelée technologie de molécule unique en temps réel, ou « SMRT » pour « Single Molecule Real-time sequencing ». Parmi ses concurrents directs, Helicos Biosciences (Helicos) qualifié « tSMS » pour « True Single Molecule Sequencing ». Malgré le recours à une technologie analogue, la mention « Temps réel » auquel il échappe est simplement liée à une incorporation cyclique des nucléotides fluorescents.
D’autres technologies, à des degrés de développement plus ou moins avancé, sont dans les tuyaux et qui sait de Noblegen, Starlight, Cracker Bio, NABSys, Halcyon, ou autres… révolutionnera encore un peu plus cet univers du haut débit et suivra le chemin emprunté dernièrement par Oxford Nanopore …
 Le séquençage haut-débit utilisant des analogues de nucléotides « portant des groupes bloquants en 3′ qui sont utilisés pour produire une terminaison de chaîne réversible appliqués pour le séquençage par synthèse nucléotidique » (je sais cette phrase est « un peu » lourde…) est un brevet de Caliper Technologies -autrement dit de Perkin Elmer : la firme qui n’a pas su saisir sa chance lors de la révolution du séquençage haut-débit. Le séquençage par synthèse (avec réversion du blocage des nucléotides fluorescents polymérisés -un peu moins lourde cette phrase là…) est le cœur de la technologie faisant une partie du chiffre d’affaire d’Illumina (l’intégralité de sa gamme de séquenceurs fonctionne sur ce principe).
Le séquençage haut-débit utilisant des analogues de nucléotides « portant des groupes bloquants en 3′ qui sont utilisés pour produire une terminaison de chaîne réversible appliqués pour le séquençage par synthèse nucléotidique » (je sais cette phrase est « un peu » lourde…) est un brevet de Caliper Technologies -autrement dit de Perkin Elmer : la firme qui n’a pas su saisir sa chance lors de la révolution du séquençage haut-débit. Le séquençage par synthèse (avec réversion du blocage des nucléotides fluorescents polymérisés -un peu moins lourde cette phrase là…) est le cœur de la technologie faisant une partie du chiffre d’affaire d’Illumina (l’intégralité de sa gamme de séquenceurs fonctionne sur ce principe).
Le principe assez simple du séquençage SBS (Sequencing By Synthesis) est en train de se décliner au sein de machines haut-débit plus accessibles en terme de coût… il s’agit là de l’arrivée sur le marché d’une nouvelle concurrence : des séquenceurs génériques tels que ceux que proposent le distributeur Azco Biotec, Inc. Ces séquenceurs viennent directement concurrencer ceux d’Illumina accompagnés de leurs réactifs « génériques » tels que ceux distribués par la société Proteigene…
Ces séquenceurs « génériques » ne révolutionnent rien, leur part de marché est actuellement marginale… les laboratoires académiques préfèrent souvent l’original à la copie, malgré tout la concrétisation de la « promesse » –qui l’a formulée au juste ?– d’un génome humain pour 100 $ d’ici 2015 passe par la large diffusion de machines dont la technologie a fait ses preuves… Le retard pris par les séquenceurs de 3ème génération laisse le champ libre à la 2ème. Le marché risque de se saturer en séquenceurs de paillasse (séquenceur de 2ème génération tels que le MiSeq d’Illumina et les PGM et Ion Proton de Life Technologies).
Un document paru dans la Ion Community (site de partage autour de la technologie Ion Torrent et Ion Proton de Life Technologies) commence à faire parler de lui. En effet, des scientifiques de Life Technologies montrent et démontrent que le MiSeq d’Illumina produit (aussi) des erreurs au niveau des homopolymères.
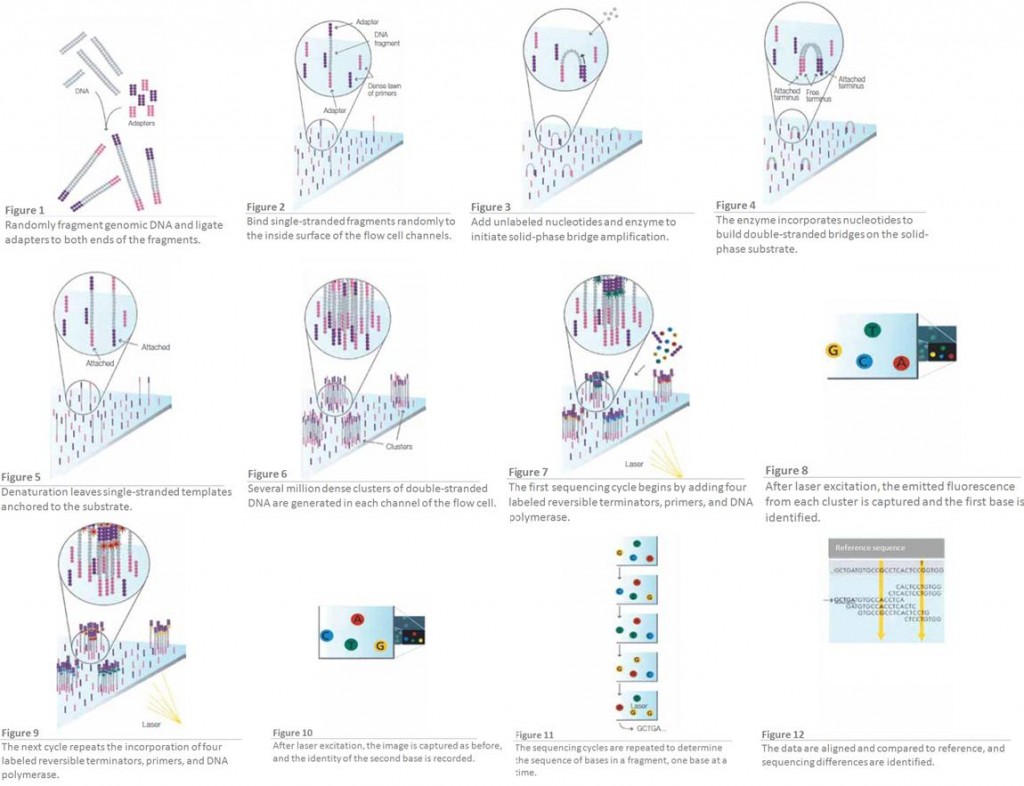
Nous savions depuis longtemps que les séquenceurs de type 454 ou Ion Torrent montraient des biais quant à détermination correcte de ce type de séquences… Ici, pour la première fois les méthodes de SBS (Sequencing By Syntesis) dont le principe pour la technologie Illumina est rappelée par le schéma disponible ci-dessous, montrent des biais étonnement importants pour la juste détermination de séquences adjacentes d’homopolymères (particulièrement concernant les régions GC % riches). La note qui met le feu dans l’argumentaire commercial d’Illumina est disponible ici-même. Une hypothèse quant à la génération de ce type d’erreurs est formulée.
Nous vous laissons le soin de lire la note, intéressante malgré la partialité de la source. La démonstration quoique scientifique, montre l’âpreté de la bataille qui se joue entre les acteurs majeurs du séquençage de paillasse de deuxième génération.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages