Il convient tout d’abord de distinguer deux types de bioinformaticiens :
-Ceux qui sont intégrés dans une unité de recherche : ils sont souvent spécialisés sur une thématique ou même un projet précis : généralement isolés, non permanent, impliqués et informés des problématiques biologiques
-Ceux qui font partie d’une unité de bioinformatique : en émulation avec d’autres bioinformaticiens, moins précaires, plus loin des problématiques biologiques
Ce document se veut une libre opinion sur la bonne intégration d’un, ou d’une équipe, de bioinformaticiens au sein d’un groupe de recherche et les clés pour la bonne réussite de celle-ci. Il est en grande partie inspiré de l’éditorial « On the organization of bioinformatics core services in biology-based research institutes » agrémenté de commentaires plus personnels.
Pour le bien-être interne et externe d’une plateforme de bioinformatique, il convient de :
-Bien séparer les tâches dîtes de prestation et les activités de recherches.
-Scinder les sujets traités par tâches bien définies : par exemple, support en développement et maintenance de base de données, analyse statistique de données haut-débit, support pour le déploiement d’applications sur environnement HPC, analyse de donnée NGS…
-Mettre en place un comité d’utilisateurs incluant des utilisateurs biologistes/bioanalystes chargés d’établir les priorités
-Mettre en place des outils pour suivre l’activité bionformatique de la façon la plus transparente possible
-Mettre en place au maximum des projets transversaux pour favoriser l’interface entre biologiste et bioinformaticiens, les interactions pluridisciplinaires sont les plus enrichissantes et souvent les plus productives. Comme toutes disciplines, la bioinformatique peut devenir endogame, particulièrement dans ou le cadre où l’on parle de bioinformatique comme une science à part entière, et non comme un outil au service de problématiques biologiques : une équipe de bioinformatique peut très bien travailler sur des sujets « porteurs » (donc plus facile à financer) sans interagir, ou seulement sporadiquement, avec des biologistes pour valider le bien-fondé du travail. C’est une des dérives du système d’évaluation actuel, pour plus d’information, lire la libre opinion publiée dans ce blog : Chemins de traverses : la recherche d’excellence ou quand les mots perdent leur sens.
-Mettre en place des formations pour les biologistes : collaborer avec des personnes connaissant un minimum les problématiques bioinformatiques est plus efficace
-Dans le même esprit, nommer un bioinformaticien responsable pour le support utilisateurs pour assister les biologistes dans leurs tâches quotidiennes de bioanalyse est très utile pour entretenir de bonne relation avec les laboratoires de recherche
-Pour les bioinformaticiens intégrés dans une unité, il est primordial de trouver des structures permettant de rencontrer et de partager avec d’autres bioinformaticiens, à défaut, passer quelques heures par semaine à effectuer de la veille technologique peut être un palliatif.
Pour conclure, une équipe de bioinformaticiens doit communiquer un maximum avec leurs partenaires biologistes afin de favoriser une saine interaction, un bioinformaticien, seul, entourer de biologistes se doit de rester à la page, idéalement en partageant et en communiquant sur ces projets à d’autres bioinformaticiens.
 Cet article est le prolongement sous forme de fiche technique, de notre article abordant le bio-art.
Cet article est le prolongement sous forme de fiche technique, de notre article abordant le bio-art.
Commençons par quelques définitions :
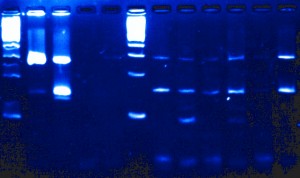
fingerprinting = signature moléculaire exploitant les polymorphismes individuels
fingerprinting = (exploitation des polymorphismes pour singulariser un prélèvement) + (séparation sur gel) + (visualisation par hybridation de sondes spécifiques marquées)
L’ensemble du génome humain est commun à tous aux mutations près. Ces mutations se produisent au hasard et se produisent à une moyenne proche de 1 ‰.
Lorsque l’on digère le génome humain (3,5 x 109 bp pour un génome haploïde) par une enzyme de restriction dont le site est à 6 bp (probabilité moyenne de coupure (1/4)6=1/4096 soit 1 site tous les 4 kbp), on obtient de l’ordre de 106 fragments qu’on ne peut examiner un à un pour déterminer la taille d’un fragment particulier. Il faut un moyen de repérer la région où la mutation est susceptible de s’être produite. Une caractéristique unique d’une région particulière d’ADN est sa séquence (à condition de n’être pas de l’ADN répété). Ainsi, après séparation des fragments selon leur taille, un blotting (ou buvardage) sera utilisé pour réaliser la signature moléculaire à l’aide de sondes marquées par un radio-isotope ou avec une molécule « traçante ».
Le southern blot est une technique permettant, après une phase de séparation sur gel, de visualiser spécifiquement des loci ciblés. Cette technique a été mise au point par E. M. Southern qui a combiné électrophorèse, transfert et hybridation avec une sonde marquée pour permettre une détection spécifique d’un locus cible (1975).
Après électrophorèse …
1. Le gel d’électrophorèse est immergé dans une solution alcaline permettant la dénaturation de l’ADN
2. Une membrane de nitrocellulose (ou de nylon) imprégnée d’une solution saline concentrée est appliquée directement sur le gel, un poids la maintient en sandwich entre une couche épaisse de substance absorbante et le gel. Trois moyens sont disponibles pour attirer la solution saline à travers le gel :
– par capillarité
– par électrophorèse
– par succion sous vide
3. La membrane est alors chauffée, dans le cas de nitrocellulose, ou exposée au rayonnement ultra-violet pour le cas du nylon, afin de fixer de manière permanente l’ADN sur la membrane. Une étape de préhybridation permettant de traiter les sites laissés libres, ils seront saturés par une solution contenant une protéine telle que de la sérum albumine et / ou traités à l’aide d’un détergent comme le laurylsulfate de sodium.
4. Une sonde spécifique marquée complémentaire de l’ADN cible est mise en contact de la membrane sur laquelle il a été transféré.
5. Une phase de « visualisation » de l’hybridation aura lieu soit par autoradiographie dans le cas de l’emploi d’une sonde marquée à l’aide d’un radio isotope (phosphore 32 par exemple) ou par développement d’un film photographique impressionné après réaction de chimioluminescence.
Cette technique est de moins en moins utilisée dans les laboratoires d’identification (police scientifique, laboratoires vétérinaires), elle a été remplacée par l’électrophorèse capillaire après une PCR sur des régions flanquantes de microsatellites (cette technique, beaucoup plus automatisable et standardisable a supplanté le SouthernBlot). Aujourd’hui cette technique tend à être remplacée par des panels de SNPs.
Deux techniques analytiques de biologie moléculaire dérivent du Southern Blot par goût du jeu de mot, elles ont été nommées :
– Northern Blot, technique analytique des ARN (James Alwine, David Kemp, and George Stark at Stanford University, 1977)
– Western Blot, technique analytique des protéines (W. Neal Burnette, 1979-1981)
Le quatrième point cardinal, le Eastern Blot, beaucoup plus marginal consiste en l’analyse des modifications post traductionnelles des protéines.
Depuis une petite dizaine d’années, une partie du monde de la recherche s’indigne souvent en silence contre un outil de mesure : les principes de l’évaluation scientifique.
Le pouvoir politique français garant du bon usage des fonds publics alloués à la recherche a calqué son appareil de mesure sur ce qui a déjà cours dans le monde anglo-saxon (l’AERES s’est inspiré notamment de l’ERIH, l’European Reference Index for the Humanities). Ainsi, ont fleuri un grand nombre de facteurs dont l’objectif est d’objectiver l’impact d’une recherche sur une société qui la finance ou plutôt d’évaluer la diffusion d’une innovation, d’une découverte (et non son impact réel sur la société).
Ci-dessous un exemple des items tirés de la « fiche de données bibliométriques » qu’un chercheur remplit en vue de satisfaire aux exigences lui permettant de voir sa carrière progresser (dossiers de contractualisation de la vague C, CNRS):
– Nombre d’articles réf Web of Science sur 2004-2007
– Nombre total d’articles réf. Web of Science, Npa
– Nombre total de citations Nc,totb
– Nombre total de citations hors autocitations
– Nombre de citations par articles Nc,tot/Npd
– Facteur h
– Facteur h relatif, h/Npf
– Facteur d’impact max. de la discipline
– Facteur d’impact moyen de la discipline
– Facteur d’impact max. pour le chercheur
– Facteur d’impact moyen pour le chercheur
Pour paraphraser un chercheur imminent, Pierre Bourdieu, parmi les bons élèves d’une classe il y a ceux qui ont compris comment les notes sont décernées et ceux qui ont compris…
Cet outil de mesure certainement nécessaire peut apparaître comme critiquable à plusieurs titres :
– l’instrument de mesure peut modifier la variable qu’il tente de mesurer (ainsi des pratiques telles que le « name-droping » des auteurs associés à une publication, les citations de convenances etc.)
– l’évaluation telle qu’elle est menée engendre des modes à l’instar de ce que relate la nouvelle de Léo Szilard, La fondation Mark Gable. Léo Szilard s’amusait des solutions qui engendreraient un retard du progrès scientifique… le présent peut par certains égards supplanter son ironie.
– ce type d’évaluation basée sur la bibliométrie engendre que « l’objectif principal des scientifiques n’est plus de faire des découvertes mais de publier autant que possible», de sorte que « l’utilité, la qualité et l’objectivité des articles se sont dégradées », c’est ce que de manière synthétique l’article du Monde Diplomatique, (comment devenir le chercheur du mois) tend à démontrer.
Par dessus tout, l’évaluation induit la mise en concurrence systématique de tous contre tous. Cette conscience même de la mise en concurrence entre équipes travaillant sur une même thématique incite ces mêmes équipes à attirer vers elles les fonds alloués à la recherche pendant que d’autres tentent de justifier leur propre existence… autant d’énergie qui n’est pas consacrée à la recherche en tant que telle : beaucoup de temps pour remplir des formulaires, beaucoup de temps pour communiquer, de moins en moins de temps pour réfléchir, développer et réaliser le travail de fond…
En définitive, le bon élève assimile et critique le système mais peut rapidement finir par exploiter les vices de l’outil de mesure : la notation. Ayant compris, il se plie aux exigences des critères de la recherche d’excellence… il remplit des formulaires, cherche comment il peut financer sa recherche, en n’oubliant pas les mots clés attendus. Pour prétendre à ces financements, il lui faudra justifier d’une certaine visibilité donc publier autant que possible… la publication devient l’alpha et l’oméga du chercheur… Ceci ayant pour effet, la dilution de l’information… et donc une perte de temps à rechercher l’information réellement pertinente.
Le constat peut paraître sans nuances mais il convient de l’inscrire dans une évolution du monde où la justification prend le pas sur la réflexion et l’action. Même s’il apparaît nécessaire d’évaluer ce que deviennent les fonds publics, il semble qu’un mauvais outil de mesure peut pervertir tout un système. C’est ainsi que l’on se gargarise à l’envi des terminologies novlangues où « l’excellence » remplace la science.
« Evaluer tue » – logo retravaillé de : http://forumpsy.wordpress.com/
 L’œuvre d’art contemporain fait débat et pour le mener à bien il semble toujours possible d’exhumer Duchamp ou bien encore de profiter des écrits d’un Yves Michaud. Humblement, cet article est une évocation de ce qui peut être qualifié d’art, simplement parce qu’il en porte le nom : le bio art.
L’œuvre d’art contemporain fait débat et pour le mener à bien il semble toujours possible d’exhumer Duchamp ou bien encore de profiter des écrits d’un Yves Michaud. Humblement, cet article est une évocation de ce qui peut être qualifié d’art, simplement parce qu’il en porte le nom : le bio art.
Commençons par ce qui n’est pas du bio-art… l’art qui imite la nature (ou l’inverse).
Il est parfois délicat de qualifier les courants et orientations de l’art contemporain, il ne faut pas se méprendre : un arbuste taillé (même par un Edouard aux mains d’argent lui-même créature bio artistique) en forme de dinosaure par exemple ne bénéficierait pas de l’appellation de bio art, sinon que dire de la période art nouveau où la végétation des boiseries et ferronneries domestiquées par Victore Horta s’invitait en douces volutes dans les escaliers des maisons des années 1900. Ces derniers exemples nous placent toujours sous la sentence d’Aristote : l’art imite la nature.
Des artistes exploitent le vivant comme médium artistique mais plutôt dans un but contemplatif et du laisser faire, tel Michel Blazy et ses fameuses sculptures pourrissantes qui ont ravi ou dégoûté le public du palais de Tokyo en 2007.
Une des modalités du bio art consiste à exploiter les moyens analytiques scientifiques pour les ériger en œuvre d’art à part entière, en œuvres porteuses de sens, une forme de rencontre entre le numérique et l’organique. Jean Claude Ameisen avec une mise en parallèle d’une image tridimensionnelle d’un kyste de Pneumocystis carinii et du portrait tourmenté de Francis Bacon, nous propose une lecture ironique et lyrique en inversant les termes du dogme aristotélicien que l’art contemporain cherche à dépasser (ici la nature imite l’art).
Qu’est ce que le bio art…? Un art qui joue des oxymores en inventant une nature synthétique …
Le bio art (ou art biotech) est une forme de l’art contemporain qui modifie les processus de vie. Cette forme d’art utilise trois modalités possibles :
Le bio art cherche en grande partie à en créer une nouvelle (nature), telle un Monsanto de salon dirait ses détracteurs. L’art réalise la nature par l’intervention non divine de la science.
1) amener de la biomatière à des formes inertes ou à des comportements spécifiques. Beaucoup d’artistes travaillent sur la frontière entre naturel et artificiel, comme le mobile perpétuel à base photosynthétique d’Amy Franceschini et Michael Swaine.
2) détourner des outils et des processus biotechnologiques pour en réaliser une œuvre en soi (ou à soi…), ce point sera plus particulièrement abordé puisqu’il a trouvé une application commerciale.
3) inventer ou transformer des organismes vivants sans réels objectifs scientifiques ou industriels. Ainsi, Joe Davis, un artiste américain notamment affilié au MIT a encodé des textes en prenant les quatre bases A, T, G, C de l’ADN pour réimplanter ceci dans des organismes vivants. Il a encodé par exemple un fragment génétique codant une phrase d’Héraclite – » Le dieu dont l’oracle est à Delphes ne révèle pas, ne cache pas mais il signifie » dans le gène d’une drosophile (gène responsable de la vue chez cette mouche, cette modification n’altère pas le phénotype de l’animal).
Cette dernière approche de transformation du vivant est la plus radicale et a pour objectif déclaré de susciter un débat que la bio-éthique mène depuis les années 60.
Une œuvre marquante, troublante et génétiquement gênante a été réalisée par Eduardo Kac avec son projet de lapine transgénique, vert fluorescente appelée « Alba« , ce projet a pu être mené à bien par des techniques transgéniques utilisées dans l’art. Ce lagomorphe chimérique rendu fluorescent grâce à un gène de méduse devait provoquer un débat sur le statut des animaux transgéniques (de la transgression à la transgénèse, en somme…)
Quelques groupes ont émergé pour pousser l’investigation bio-artistique. Parmi quelques groupes de recherche institutionnels, deux d’entre eux symbiotica et bioteknica, réciproquement rattachés à l’université de Western Australia et à l’université de Québec à Montréal, proposent un grand nombre d’expositions et d’ouvrages plutôt bien accueillis par la communauté scientifique. Outre des biologistes et des plasticiens ce type de structures accueille des philosophes, sociologues et anthropologues afin de permettre l’analyse des interactions entre les biotechnologies et la société, entre l’art et la biologie, l’Homme et son environnement…
L’innovation et la recherche du spectaculaire qui a aussi cours dans le monde des sciences et notamment dans le monde de biologie cellulaire et moléculaire (dont rappelons le, grand nombre de techniques cherchent à montrer de manière indirecte ce qui demeure invisible) devient un médium que le bio art exploite pour des fins tant esthétiques qu’égotistiques. En effet, au carrefour entre le bio art, la biologie moléculaire et Ikea, ont fleuri des marchants d’œuvre d’art relativement bon marché proposant votre propre emprunte ADN (détournant ainsi les techniques de « fingerprinting » moléculaire) imprimée qui deviendra ornement d’intérieur, un vernis commercial transformant le bio art en « ego art ».
Ainsi se sont développées sur internet des sociétés (telle que la société DNA 11, par exemple) proposant de mettre en œuvre ce type de techniques qui désertent les laboratoires pour se banaliser dans un champ d’application inattendu.
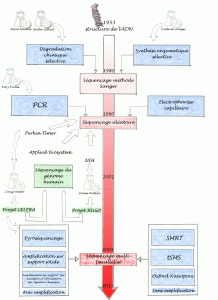
Un bref historique des évolutions du séquençage de l’ADN permet de comprendre ce que certains nomment révolution technologique… certainement concernant l’univers de la biologie moléculaire une innovation comparable à celle la PCR.
Allan Maxam , Walter Gilbert (USA) et Frederick Sanger (Royaume-Uni), les pionniers de la conquête de la séquence, ont mis au point deux méthodes très différentes permettant d’accéder à la lecture de la séquence. Maxam, Gilbert ont exploité des stratégies de dégradation chimique sélective quand Sanger choisit une stratégie de synthèse enzymatique sélective. Pour ces découvertes, Gilbert et Sanger ont été récompensés par le prix Nobel de chimie en 1980. L’histoire ne retient effectivement que Sanger, à juste titre, puisque cette stratégie bénéficiant de l’invention de laPCR (invention de Kary Mullis publiée en 1986, lui aussi nobélisé par le prix de chimie en 1993) et du développement de l’électrophorèse capillaire, permettant de simplifier la partie séparative et analytique. C’est ainsi que des sociétés comme Perkin Elmer, Beckman Coulter et Applied Biosystem ont investi le marché sur le principe même du séquençage Sanger sans le révolutionner mais en permettant son automatisation et l’augmentation du nombre de réactions de séquences analysables par jour. Une fois amorti le coût des séquenceurs, le coût de revient d’une séquence n’a cessé de baisser permettant de banaliser l’accès à la séquence.
Bien que se montrant de plus en plus performant, des applications comme le séquençage complet de génomes eucaryotes supérieurs, les approches métagénomiques (nécessité de cloner), les études de modulation de transcrits (la méthode SAGE les permettait sur la base du séquençage Sanger, cette méthode est lourde et nécessite toujours une phase de clonage) ont connu des limites quasi infranchissables (méthodes nécessitant trop de temps et de capitaux). Prenons à titre d’exemple les projets de séquençage du génome humain. Ces projets auront nécessité plus de 10 ans de travail et de 300 millions de USD pour le projet privé de Celera et un peu moins de 3 milliards de USD pour le projet HUGO (HUman Genome Organisation), le match entre Craig Venter (fondateur de Celera avec le soutien de Perkin Elmer) et James Watson (premier directeur du NIH coordinateur du consortium international) se solde par un match nul entaché de polémiques avec les publications en 2001 des premières séquences du génome humain.
L’année 2005 a connu à grands bruits, l’arrivée de nouvelles méthodes de séquençage à haut débit. Ces méthodes de séquençage massif font appel aux techniques de clonage et d’amplification moléculaire, leur spécificité relevait de leur stratégie de lecture.
En effet, la société 454 (rachetée par Roche en 2007), utilise des méthodes de pyroséquençage (luminescence par libération de pyrophosphate) de fragments d’ADN isolés dans des micro-gouttes comme micro-réacteur de PCR isolés au sein d’une émulsion, la société Solexa (rachetée par Illumina en 2007), utilise des méthodes d’amplification sur support solide permettant l’incorporation de bases terminateurs de chaîne réversibles marqués par des fluorochromes. La société Agencourt (rachetée par Applied Biosystem en 2006), a quant à elle basé son système de détection sur le principe de l’amplification par émulsion et hybridation-ligation chimique.
Depuis 2007, une certaine frénésie s’empare du monde de la biologie moléculaire, un très grand nombre de machines que l’on qualifie tour à tour de NGS (Next Generation Sequencing), HTS (High Throughput Sequencing), ou encore de manière plus appropriée sequençage multi-parallélisé se trouvent disponibles et évoluent vers le plus de profondeur, ou vers le moindre coût, rendant obsolètes les versions précédentes (la société Illumina à titre d’exemple depuis le rachat de Solexa a lancé un modèle de séquenceur tous les ans).
Cette révolution technologique est sortie des laboratoires pour attirer l’attention d’investisseurs et autres banquiers conscients qu’une ruée vers l’or accompagnant la ruée vers la séquence pouvait s’engager. Dans ce sens nous mettons à disposition un document datant de 2007 réalisé pour le compte de la Deutsche Bank (ce document très complet a été co-réalisé par nombre de consultants en biotechnologies, il permet de décortiquer assez finement les 3 technologies présentes sur le marché à partir de 2005 et de montrer les attentes des mondes scientifique et financier)
Afin de terminer ce bref tour d’horizon il semble nécessaire de hiérarchiser ces nouvelles technologies. Il est possible de distinguer tout d’abord deux grands groupes : les technologies sans amplification (permettant de séquencer une seule molécule d’adn matriciel, citons exemple de la technologie SMRT de Pacific Biosciences, la technologie tSMS de Helicos Biosciences ou encore celle toujours en développement mais très attendue développée par Oxford Nanopore) et avec amplification (toutes les technologies amplifiant une matrice clonale d’adn issu d’une librairie)
Parmi cette seconde classe, il est possible de distinguer trois grandes classes de matériel :
– les usines à produire de la séquence (type Hiseq 2000 de Illumina permettant de séquencer jusqu’à 200 Gb par run de 8 jours
– les séquenceur de paillasse (type PGM de IonTorrent de taille et prix beaucoup plus modeste permettant de réaliser des run autour de 100 Mb avec une perspective de 1 Gb pour début 2012)
– les séquenceurs haut débit en voie d’obsolescence (454 de Roche, les versions des séquenceurs Illumina antérieures au HiSeq 1000 etc.)
Cet historique est bien entendu un prétexte pour planter le décor et présenter les forces en présence. Après l’âge des pionniers (Maxam et Sanger), celui de la douce exploitation (séquenceur capillaire) vient celui de l’accélération que certains nomment révolution… ces technologies marquent quoi qu’il en soit un tournant pour la génomique et pour les applications actuelles et en devenir qui y sont liées. Enfin la diffusion de ces technologies va de paire avec un changement de physionomie des laboratoires : la biotechnologie est une science composite où bio-informatique, biologie cellulaire et moléculaire mais aussi physique et statistique deviennent de plus en plus interdépendantes.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages