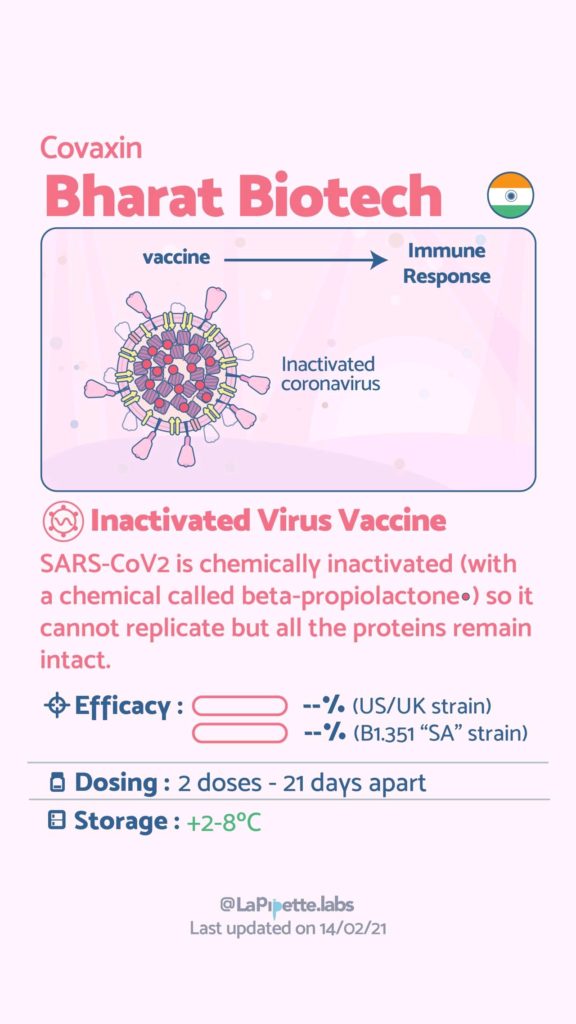
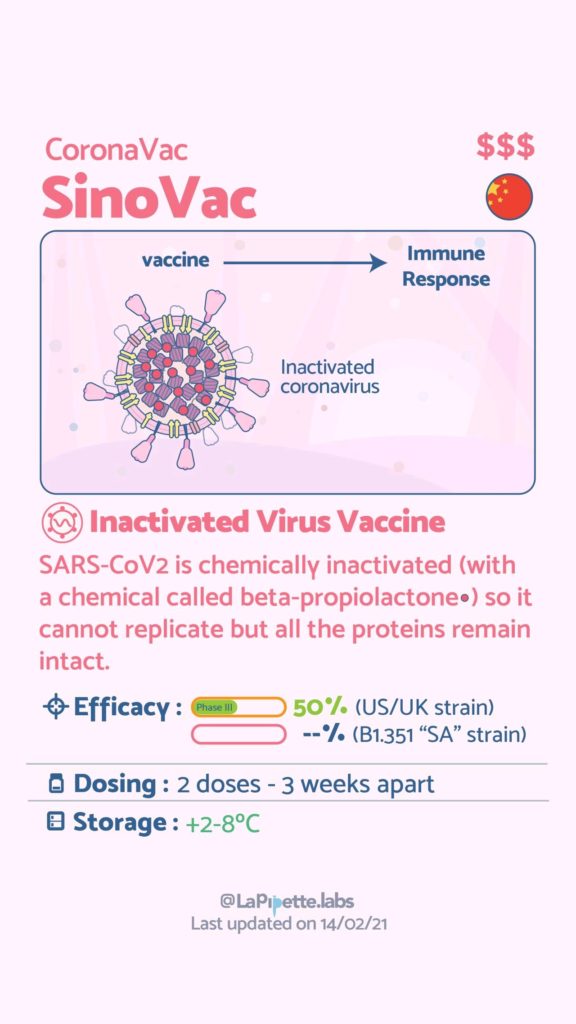
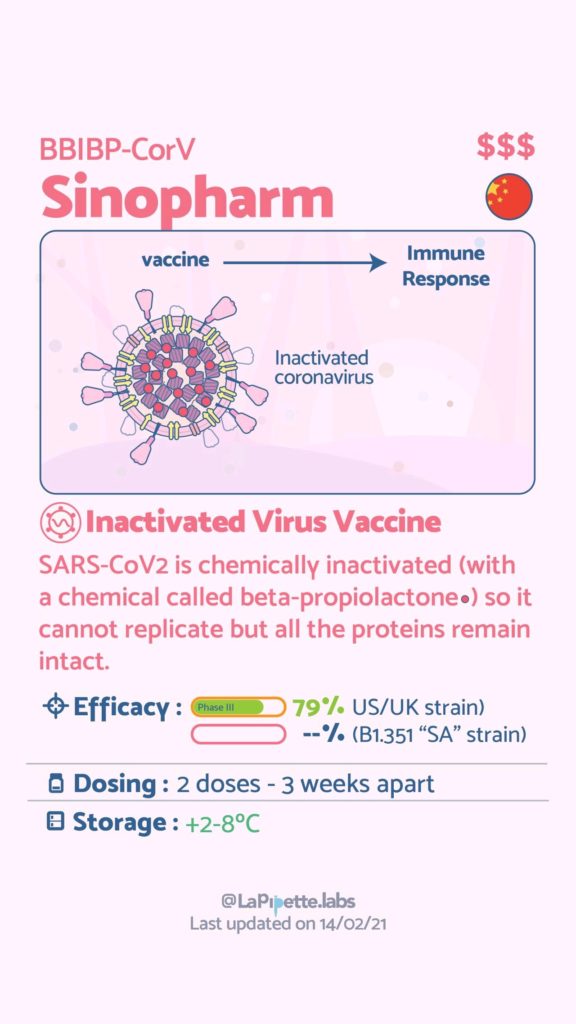
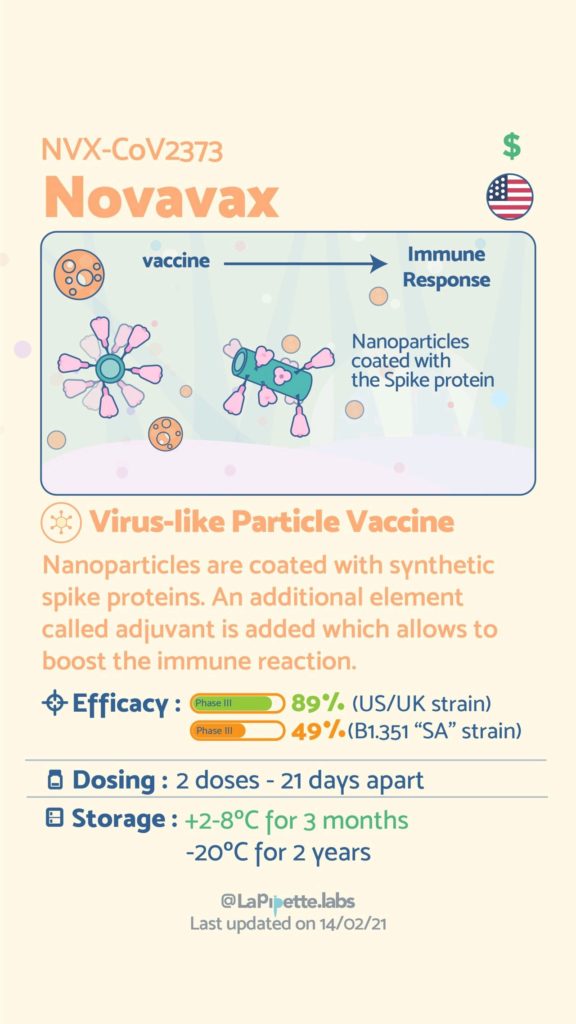
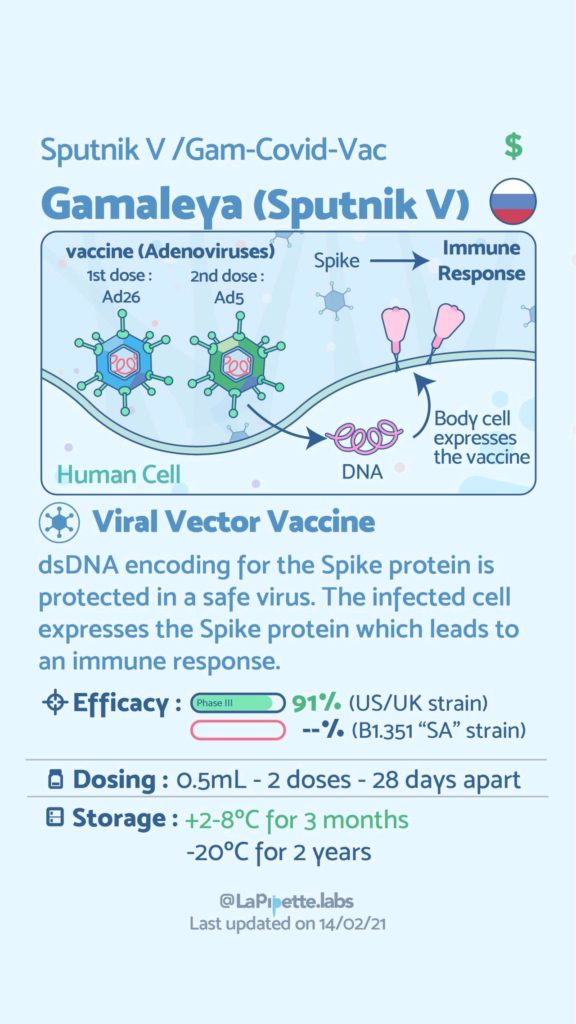
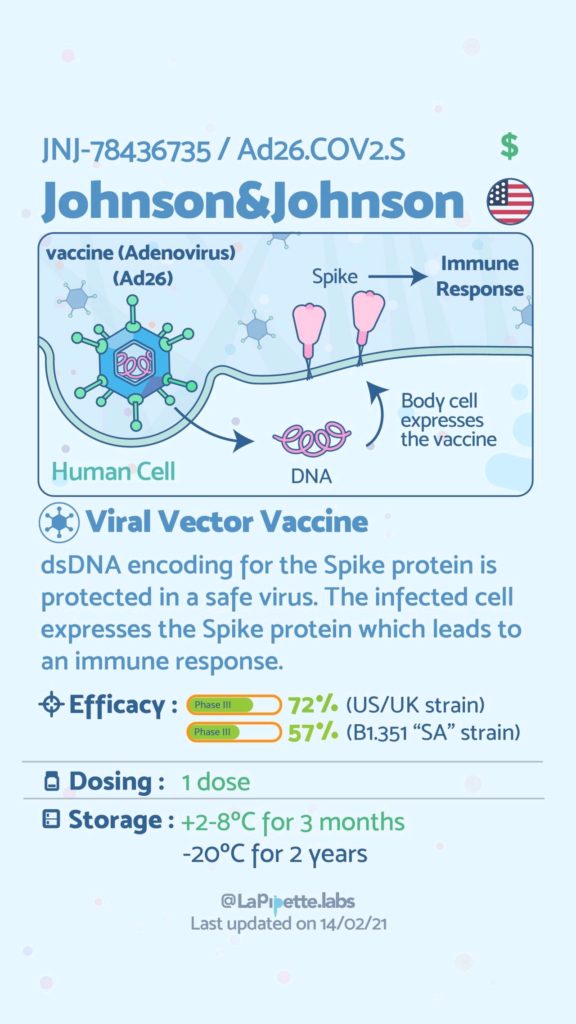
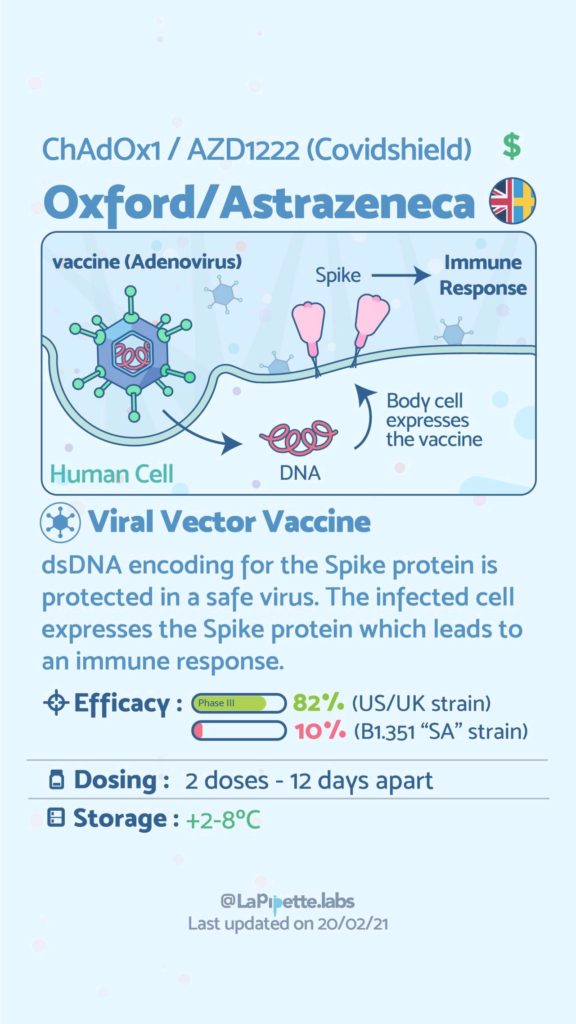
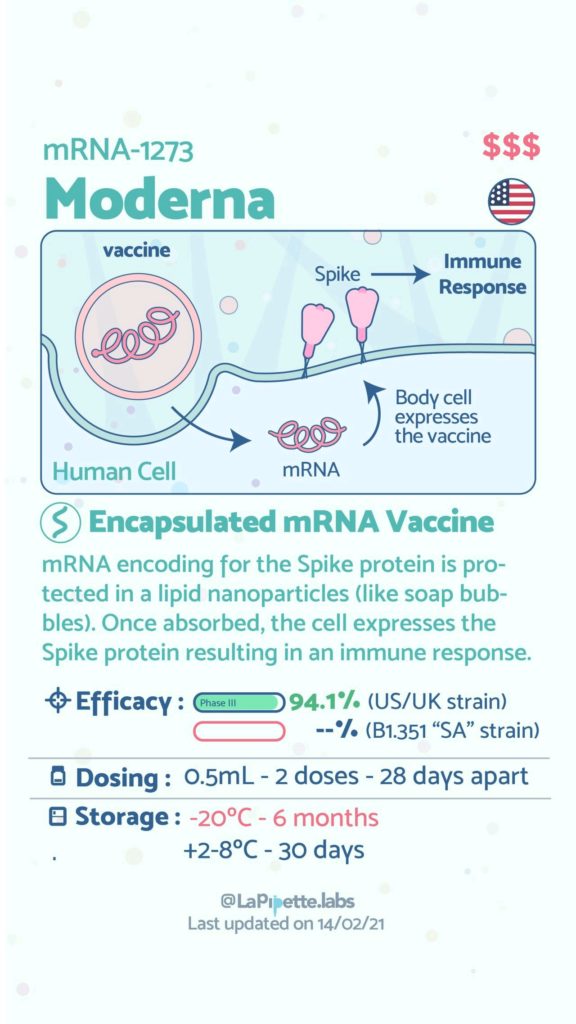
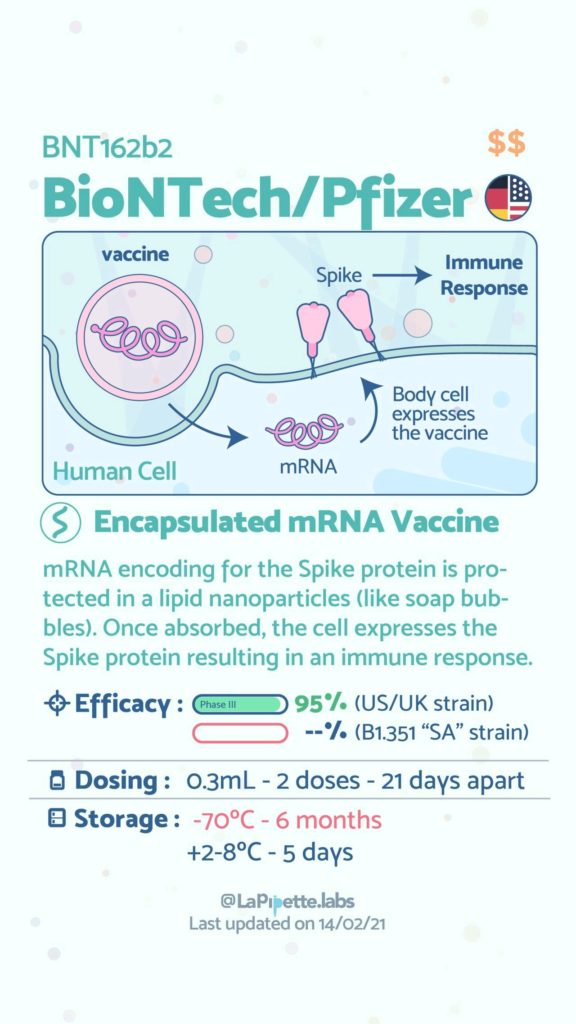
Ces excellentes infographies créditées à LaPipette.labs (https://www.instagram.com/lapipette.labs/?hl=fr) font le tour des technologies employées dans les 9 vaccins ayant franchi l’ultime étape les amenant à leur mise sur le marché.
Nous avions pu, il y a deux ans (en 2018), aborder cet usage : l’ADN comme support d’informations numériques
Cette article fait suite à notre post sur l’intervention du professeur Arnaud Fontanet de l’Institut Pasteur sur le Coronavirus COVID-19.
Dans sa présentation, le professeur Fontanet renvoie vers trois sites web qui permettent de mieux comprendre le coronavirus.
Chacun dans leur contexte (observation/simulation/étude), ces sites montre la rapidité avec laquelle les chercheurs peuvent développer des outils bioinformatiques de data visualisation pertinents pour la communauté.
Ceci étant bien sur rendu possible à partir du moment où le partage de données épidémiologiques, génétiques, génomiques (…) est effectué.
Pour observer :
Coronavirus COVID-19 Global Cases by Johns Hopkins CSSE
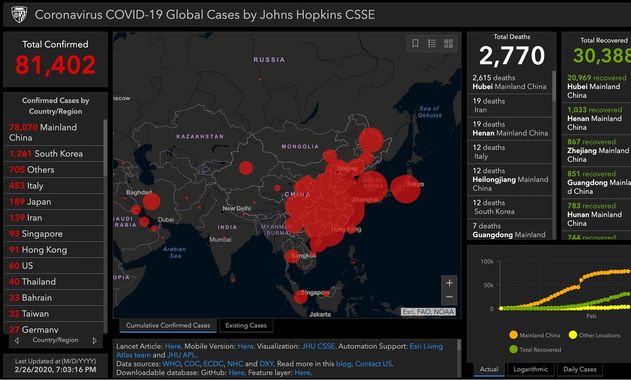
L’université John Hopkins maintient une carte avec des données en temps réel sur le nombre de patients diagnostiqués avec le nouveau coronavirus, le nombre de patients décédés et le nombre de patients guéris. Ces chiffres sont basés sur des informations provenant, entre autres, de l’Organisation mondiale de la santé (OMS) et du Centre européen de prévention et de contrôle des maladies (ECDC). Il peut y avoir de légères différences dans les chiffres réels .
Pour connaître les derniers chiffres confirmés, nous renvoyons aux sites web de l’OMS et de l’ECDC
Github – entrepôt de données : https://github.com/CSSEGISandData/COVID-19
Pour anticiper :
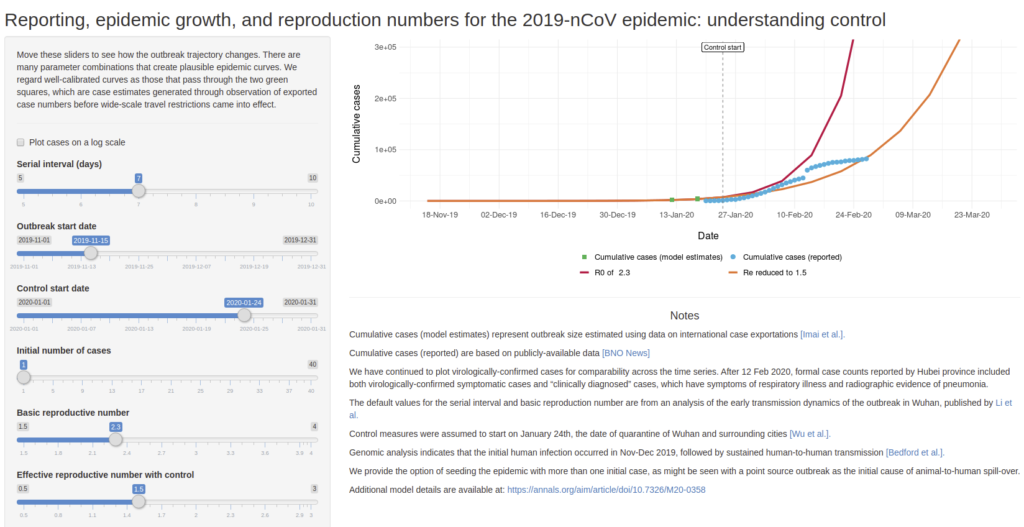
Permet de simuler des scénario de croissance de l’épidémie de COVID-19 en faisant varier quelques paramètres comme :
Serial interval (days) : nombre de jours avant de tomber malade
Outbreak start date : date de début de la maladie
Control start date : date de mise en place de controle (quarantaine, confinement,…)
Initial number of cases : nombre de cas initialement détectés
Basic reproductive number : nombre de personne à leur tour infecté par un malade si aucun contrôle n’est mis en place
Effective reproductive number with control : nombre de personnes à leur tour infecté par un malade si un contrôle est mis en place
Développé par Ashleigh Tuite et David Fisman, Dalla Lana School of Public Health, Université de Toronto
Pour étudier :
Genomic epidemiology of novel coronavirus (HCoV-19)
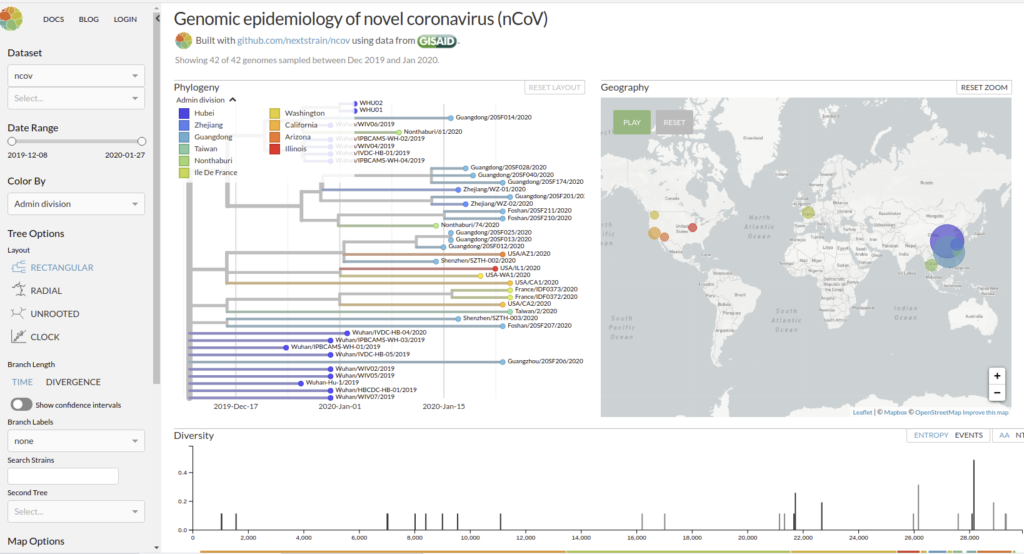
Nextstrain est un projet à open-source visant à exploiter le potentiel scientifique et de santé publique des données sur le génome des agents pathogènes. ils fournissent une vue continuellement mise à jour des données accessibles au public ainsi que de puissants outils d’analyse et de visualisation à l’usage de la communauté. L’objectif est d’aider à la compréhension épidémiologique et d’améliorer la réponse aux épidémies.
Il permet de visualiser les divergences phylogeniques entre les différentes génomes de COVDIR-19 séquencés à ce jour [ 20/02/2020 ]
En savoir plus : Hadfield et al., Nextstrain: real-time tracking of pathogen evolution, Bioinformatics (2018)
Github de l’application : https://github.com/nextstrain/ncov
Des explications précises et instructives sur l’état actuel (au 20 février 2020) des connaissances concernant l’épidémie provoquée par ce nouveau Coronavirus (COVID-19) – Durée : 35Min
Le professeur Arnaud Fontanet revient en détail sur l’histoire de ce virus, de la découverte des premiers cas, à l’enquête sur son mode de transmission jusqu’à son séquençage extrêmement rapide.
Il donne également beaucoup d’informations sur la durée d’incubation, la contagion, les symptômes associés, les mesures sanitaires et le travail des épidémiologistes et des chercheurs pour endiguer la propagation.
Les conséquences économiques ainsi qu’un parallèle pas inintéressant avec la grippe saisonnière permettront, pour certains, de mettre en perspective cette épidémie par rapport à notre monde actuel…
Cette intervention fait partie du MOOC de l’Institut Pasteur « Virus émergents et réémergents ».
Selon une étude : La consommation (modérée) de vin rouge liée à une meilleure santé intestinale…
Une étude du King’s College de Londres tend à montrer que les personnes qui boivent du vin rouge présenteraient une plus grande diversité de microbiote intestinal (un signe de santé intestinale) que les buveurs de vin non rouge.
Dans un article publié le 28 août dans la revue Gastroenterology, une équipe de chercheurs du Department of Twin Research & Genetic Epidemiology au King’s College de Londres a étudié l’effet de la bière, du cidre, du vin rouge, du vin blanc et des spiritueux sur le microbiote intestinal et sur la santé qui en découle chez un groupe de 916 jumelles britanniques. Pourquoi des jumelles? Pour que le fond génétique soit identique, donc la différence de microbiote intestinal entres les jumelles sera en grande partie dû à l’environnement.
Ils ont constaté que le microbiote intestinal des buveurs de vin rouge était plus diversifié que celui des buveurs de vin non rouge. Ceci n’a pas été observé avec la consommation de vin blanc, de bière ou de spiritueux.
La première auteure de l’étude, Caroline Le Roy, du King’s College de Londres, a déclaré : « Bien que nous connaissions depuis longtemps les bienfaits inexpliqués du vin rouge sur la santé cardiaque, cette étude montre qu’une consommation modérée de vin rouge est associée à une plus grande diversité et à un microbiote intestinal plus sain qui explique en partie ses effets bénéfiques sur la santé. »
Le microbiome est la collection de micro-organismes dans un environnement et joue un rôle important dans la santé humaine. Un déséquilibre entre les « bons » microbes et les « mauvais » microbes dans l’intestin peut entraîner des effets néfastes sur la santé, comme un affaiblissement du système immunitaire, un gain de poids ou un taux de cholestérol élevé.
Le microbiote intestinal d’une personne ayant un nombre plus élevé d’espèces bactériennes différentes peut-être considéré comme un marqueur de la santé intestinale.
L’équipe a observé que le microbiote intestinal des consommateurs de vin rouge contenait un plus grand nombre d’espèces bactériennes différentes que celui des non-consommateurs. Ce résultat a également été observé dans trois cohortes différentes au Royaume-Uni, aux États-Unis et aux Pays-Bas. Les auteurs ont tenu compte de facteurs tels que l’âge, le poids, le régime alimentaire régulier et le statut socio-économique des participants et ont continué à voir l’association.
Les auteurs croient que la raison principale de cette association est due aux nombreux polyphénols présents dans le vin rouge. Les polyphénols sont des produits chimiques de défense naturellement présents dans de nombreux fruits et légumes. Ils ont de nombreuses propriétés bénéfiques (y compris des antioxydants) et agissent principalement comme un carburant pour les microbes présents dans notre système.
L’auteur principal, le professeur Tim Spector du King’s College de Londres, a déclaré : « Il s’agit de l’une des plus importantes études jamais réalisées sur les effets du vin rouge sur l’intestin de près de trois mille personnes dans trois pays différents et elle montre que les niveaux élevés de polyphénols dans la peau du raisin pourraient être responsables d’une grande partie des bienfaits controversés pour la santé lorsqu’ils sont utilisés avec modération. »
« Bien que nous ayons observé une association entre la consommation de vin rouge et la diversité du microbiote intestinal, boire du vin rouge rarement, comme une fois toutes les deux semaines, semble suffisant pour observer un effet. Si vous devez choisir une boisson alcoolisée aujourd’hui, c’est le vin rouge qu’il faut choisir, car il semble exercer un effet bénéfique sur vous et sur vos microbes intestinaux, ce qui peut aussi aider à réduire le poids et le risque de maladies cardiaques. Cependant, il est toujours conseillé de consommer de l’alcool avec modération, vous n’avez pas à boire du vin rouge, et vous n’avez pas à commencer à en boire si vous ne buvez pas », a ajouté le Dr Le Roy.
Effectivement, comme dans toute étude, corrélation n’est pas raison! Ainsi et même si la catégorie socio-économique est prise en compte dans l’étude, il pourrait exister d’autres facteurs, non mesurés dans cette étude, qui expliqueraient en partie la bonne santé microbienne des individus. Rappelons que ça n’est pas l’alcool qui est associé avec une meilleure santé intestinale mais d’autres composants du vin rouge (l’hypothèse étant que ce sont les polyphénols), que l’on trouvera aisément dans d’autres aliments (fruits, légumes, noix, cacao…) .
Emplacement de la publication originale :
https://www.sciencedirect.com/science/article/abs/pii/S0016508519412444
L’abus d’alcool est dangereux pour la santé, consommez avec modération
 Que feriez vous avec un séquenceur qui tient dans la paume de votre main… ? Alors qu’il y a quelques années était annoncée l’arrivée de troisième génération de séquenceur, toujours plus sensibles (permettant de séquencer l’ADN natif, non pré-amplifié comme cela peut être le cas dans les technologies de séquençage de 2ème génération), générant des reads toujours plus longs, entachés de beaucoup plus d’erreurs… C’est aujourd’hui, avec le séquençage Minion de Nanopore que se pose réellement la question du changement d’applications qu’induirait le fait de posséder ce type de technologies.
Que feriez vous avec un séquenceur qui tient dans la paume de votre main… ? Alors qu’il y a quelques années était annoncée l’arrivée de troisième génération de séquenceur, toujours plus sensibles (permettant de séquencer l’ADN natif, non pré-amplifié comme cela peut être le cas dans les technologies de séquençage de 2ème génération), générant des reads toujours plus longs, entachés de beaucoup plus d’erreurs… C’est aujourd’hui, avec le séquençage Minion de Nanopore que se pose réellement la question du changement d’applications qu’induirait le fait de posséder ce type de technologies.
Cet article paru ce mois-ci dans la revue Médecine/Sciences, invite à réfléchir sur les conséquences de l’introduction de cette technologie en milieu hospitalier : Séquençage par nanopores – Perspectives d’applications en santé humaine essaie de faire le tour de la question.
Comment réagiriez vous si l’on vous « offrait » votre génotypage complet, ouvrant la possibilité de prédire d’éventuelles maladies, réactions à certains médicaments etc. ? Certes, dans le cas de l’Estonie (contrairement à ce que propose des sociétés privées telles 23andMe) il s’agit d’une démarche s’inscrivant en santé publique pour le meilleur du bien public et non pour le pire du bien privé… Néanmoins, l’ampleur de la cohorte humaine visée n’est pas sans poser des questions.
L’Estonie a lancé un programme visant à recruter et à génotyper 100.000 nouveaux participants à la biobanque (pour une population nationale totale de 1,316 million de citoyens estoniens !) dans le cadre de son programme national de médecine personnalisée. Cette biobanque comportait déjà 50.000 génotypes de citoyens estoniens. Le gouvernement veut développer un système de soins de santé en offrant à un maximum de ses résidents un génotypage scannant le génome qui sera traduit en rapports personnalisés. Ce rapport serait intégré à la pratique médicale quotidienne par l’entremise du portail national de cybersanté.
Cette biobanque a été initiée à trois fins (Leitsalu et al., International Journal of Epidemiology, 2015) :
- promouvoir le développement de la recherche génétique ;
- recueillir des informations sur l’état de santé de la population estonienne, ainsi que des informations génétiques
- utiliser les résultats de la recherche génétique pour améliorer la santé publique.

Au sein de l’article de Leitsalu et al., les auteurs abordent les forces et faiblesses de leur entreprise :
La principale force de la Biobanque estonienne consiste au fait qu’il s’agit d’une biobanque basée sur la population avec une base de données longitudinales et prospectives. Cela signifie qu’un large éventail de groupes d’âge et de phénotypes sont représentés. Alors que les populations urbaines ont généralement tendance à être surreprésentées, ce n’est pas le cas pour la cohorte de la Biobanque estonienne. La biobanque dispose d’ADN, de plasma et de globules blancs pour chaque donneur. Cela signifie qu’il est possible d’analyser les effets directs des variants de séquence sur le métabolisme. De plus, il est possible de transformer les cellules en lignées cellulaires ou en cellules souches pluripotentes induites (iPS) et de réaliser directement des expériences de biologie moléculaire ou de génétique. Une autre force est fournie par la HGRA (the Estonian Human Genes Research Act) ainsi que par le formulaire de consentement général qui permet de participer à un large éventail de projets de recherche sans avoir à communiquer de nouveau et à demander un nouveau consentement. La HGRA et le formulaire de consentement permettent également aux donneurs de demander la divulgation de leurs données génétiques, de leurs caractéristiques héréditaires et des risques génétiques obtenus à partir de la recherche génétique menée. Cela permettrait à terme de mener des projets sur les tests du génome personnel, la perception des risques et la gestion des risques en milieu industriel.
De plus, la HGRA permet à la Biobanque d’obtenir des renseignements supplémentaires en reliant les dossiers aux registres électroniques nationaux et aux principaux hôpitaux. Tous les registres sont reliés de façon centralisée par une infrastructure technique à l’échelle nationale qui permet l’échange sécurisé de données entre les bases de données. La HGRA a également imposé des restrictions sur les activités de l’EGCUT (Estonian Genome Center of the University of Tartu) et les données collectées dans la Biobanque estonienne. La participation devait être entièrement volontaire – seules les personnes intéressées pour rejoindre la Biobanque estonienne, après en avoir entendu parler soit lors d’événements promotionnels spéciaux, soit par les médias, soit par des amis, soit au cabinet du médecin de famille ou à l’hôpital, sont recrutées. L’EGCUT n’a pas été autorisé à envoyer les lettres d’invitation à leur adresse domiciliaire. Par conséquent, la biobanque ne représente pas un échantillon aléatoire classique et pourrait être sujette à un biais de recrutement. Une proportion considérable de la population recrutée pourrait toutefois compenser ce biais. Par conséquent, bien qu’elle ne soit pas aléatoire sur le plan classique, la cohorte peut quand même être considérée comme représentative de la population. Bien que le recrutement était ouvert à tous, il y a une disproportion d’Estoniens ethniques et de Russes ethniques dans la biobanque, les Estoniens étant surreprésentés (81% dans la biobanque contre 70% dans la population générale) et les Russes sous-représentés (16% dans la biobanque contre 25% dans la population générale). Une autre faiblesse est la profondeur limitée de certains sous-questionnaires. Par exemple, un questionnaire relativement bref sur la fréquence des aliments a été administré sans information détaillée sur l’apport en énergie ou en nutriments ; les mesures des traits glycémiques à jeun, comme le niveau d’insuline, ne sont disponibles que pour un nombre limité d’échantillons. La profondeur limitée des données recueillies peut parfois limiter le nombre de projets dans lesquels les données peuvent être utilisées. Toutefois, des questionnaires plus complets auraient exigé des durées d’entrevue encore plus longues et auraient coûté beaucoup plus cher, ce qui aurait pu entraîner une réduction de la taille de la cohorte.
Le pays dispose de nombreuses solutions numériques sécurisées incorporées dans les fonctions gouvernementales qui relient les diverses bases de données du pays par des voies cryptées de bout en bout. Un site Web a été créé dans le cadre du projet, afin que les Estoniens puissent se porter volontaires et donner leur consentement à être génotypés. La génération des données est assurée par l’institut de génomique de l’Université de Tartu (aujourd’hui les 50.000 premiers génotypages ont été réalisés et analysés, les 100.000 pousseront à une population estonienne à 10 % génotypée)
Les efforts internationaux ont permis d’identifier des milliers d’associations entre les variants génétiques et les maladies, ou traits génétiques, et de créer des cartes des variations uniques au sein des populations.
« Aujourd’hui, nous avons suffisamment de connaissances sur le risque génétique des maladies complexes et la variabilité interindividuelle des effets des médicaments pour commencer à utiliser systématiquement ces informations dans les soins de santé au quotidien « , a déclaré Jevgeni Ossinovski, ministre de la Santé et du Travail. « En coopération avec l’Institut national pour le développement de la santé et l’Université de Tartu, nous allons permettre à 100.000 autres personnes de rejoindre la biobanque estonienne, afin de stimuler le développement de la médecine personnalisée en Estonie et de contribuer ainsi à l’avancement des soins de santé préventifs. «
Le gouvernement estonien a alloué 5 millions d’euros au programme au cours de l’année 2018. Le projet sera coordonné par l’Institut national pour le développement de la santé, dont la tâche est d’élaborer et de mettre en œuvre des procédures et des principes pour la mise en œuvre efficace de la recherche scientifique dans la pratique médicale.
Andres Metspalu, directeur du Centre estonien du génome à l’Université de Tartu, se félicite de l’initiative du ministère des Affaires sociales d’augmenter le nombre de participants à la biobanque. « Nous sommes heureux qu’avec le soutien de ce projet, les résultats des travaux à long terme du centre de génomique seront transférés en médecine pratique et donneront un nouvel élan à nos recherches futures. L’Université contribuera également à la création d’un système de rétroaction pour les participants de la biobanque, et à la formation des professionnels de la santé pour qu’ils puissent donner aux patients une rétroaction fondée sur l’information génétique« .
Le projet sera mis en œuvre sur la base de la loi estonienne sur la recherche sur les gènes humains et du même formulaire de consentement général qui a été utilisé pour les 50.000 premiers participants. Le prélèvement officiel d’échantillons a débuté le 2 avril 2018. A voir si l’expérience relativement pionnière ,à cette échelle, menée en Estonie, fera école ou tâche d’huile dans d’autres pays de l’Union Européenne !
on en est pas loin avec cet outil publié aujourd’hui dans Genome Biology
A lire… pour se forger un avis… et puis l’oublier pour continuer comme avant :
Article initialement publié sur https://tempsreel.nouvelobs.com/rue89/rue89-sciences/20160519.RUE2928/je-ne-publierai-plus-jamais-dans-une-revue-scientifique.html

« Je ne publierai plus jamais dans une revue scientifique »
Olivier Ertzscheid, enseignant-chercheur et blogueur renommé, explique pourquoi le système des revues scientifiques – depuis l’évaluation par les pairs jusqu’aux abonnements exorbitants – va à l’encontre du travail scientifique et de sa diffusion au plus grand nombre.
Enseignant-chercheur, je ne publie plus que vraiment très occasionnellement dans des revues scientifiques. Et ce pour plusieurs raisons.
Monde de dingue
D’abord le modèle économique de l’oligopole (voire du quasi monopole dans le cas des SHS), qui gère aujourd’hui la diffusion des connaissances au travers de revues, est celui d’une prédation atteignant des niveaux de cynisme (et de rente) de plus en plus hallucinants.
A tel point que de plus en plus d’universités préfèrent carrément renoncer à l’ensemble de leurs abonnements chez Springer ou Elsevier. La dernière en date est celle de Montréal.
Cette situation est hallucinante et ubuesque :
- Hallucinante tant les tarifs d’Elsevier (ou de Springer) et les modalités d’accès proposées relèvent du grand banditisme et de l’extorsion de fonds.
- Ubuesque car nous sommes dans une situation où des universités doivent renoncer, j’ai bien dit renoncer, à accéder à des revues scientifiques. Monde de dingue.
Un peu comme si des agriculteurs devaient renoncer à certaines graines et semences du fait des pratiques de certaines firmes agro-alimentaires. Monde de dingue au carré.
D’autant qu’on sait que dans ce choix extrêmement délicat effectué par l’université de Montréal, l’existence de Sci-Hub, (site « illégal » dont je vous reparlerai un peu plus tard dans ce billet), pourrait avoir largement pesé dans la balance.
Parce que c’est ahurissant mais c’est ainsi, pour faire de la recherche scientifique aujourd’hui en France (et ailleurs dans le monde), il faut nécessairement passer par des bibliothèques clandestines (Shadows Libraries).
Ensuite les « éditeurs » desdites revues ont arrêté depuis bien longtemps de produire le travail éditorial qui justifiait le coût et l’intérêt desdites revues : ils se contentent le plus souvent d’apposer leur « marque », toutes les vérifications scientifiques (sur le fond) sont effectuées gratuitement par d’autres chercheurs, et les auteurs eux-mêmes se coltinent l’application de feuilles de style la plupart du temps imbitables.
Un système totalement biaisé
Alors bien sûr vous allez me dire que l’intérêt des publications scientifiques dans des revues c’est que des « pairs », d’autres universitaires, vérifient que l’on ne raconte pas de bêtises. Et moi je vais vous répondre en un mot comme en 100 : B-U-L-L-S-H-I-T. Total Bullshit. Hashtag Total Bullshit même.
Bien sûr que l’évaluation par les pairs c’est important. Sauf que même à l’époque où je publiais encore régulièrement dans des revues soumises à l’évaluation par les pairs, (et en l’occurrence « soumises » n’est pas un vain mot), ladite évaluation de mes pairs se résumait neuf fois sur dix à m’indiquer :
- Que je n’avais pas, ou insuffisamment, cité les travaux de tel ou tel mandarin (ou de l’évaluateur lui-même…).
- Que c’était très intéressant mais que le terme « jargon 1 » prenait insuffisamment en compte les travaux se rapportant au terme « Jargon 2 ». Jamais, je dis bien jamais aucun débat scientifique, aucune idée neuve, aucune confrontation d’idée, juste une relecture tiédasse.
- Que ce serait mieux si je changeais cette virgule par un point-virgule.
Mais nonobstant, c’est vrai que la vraie évaluation par les pairs c’est important. Sauf que JAMAIS AUCUN CHERCHEUR NE S’AMUSERA A PUBLIER DES CONNERIES juste pour voir si ses pairs s’en rendront compte ou pas.
Parce que, d’abord, en général, les chercheurs sont plutôt des gens instruits, relativement compétents, et relativement soucieux de contribuer à l’avancée des connaissances.
Et aussi parce que SI TU PUBLIES UN ARTICLE AVEC DES CONNERIES SCIENTIFIQUES OU DES METHODOLOGIES FOIREUSES ben tu te fais immédiatement aligner et ta carrière est finie. Sauf bien sûr si c’est pour faire une blague ; -)
Des revues lues par personne
Alors soyons clair, nul n’est heureusement infaillible et, à moi aussi, il m’est arrivé de publier des articles sur mon blog de chercheur contenant sinon des conneries, en tout cas quelques inexactitudes ou imprécisions.
Lesquelles m’ont été immédiatement signalées de manière tout à fait constructive par les lecteurs dudit blog, qui sont loin d’être tous des scientifiques-chercheurs-universitaires.
Bref le syndrome Wikipédia. Oui il y a des erreurs dans Wikipédia, mais non il n’y en a pas plus que dans les encyclopédies classiques, et oui, à la différence des encyclopédies classiques, elles sont presque immédiatement signalées et corrigées.
Parce que ces putains de revues scientifiques ne sont lues par personne !
Ai-je besoin de développer ? Des milliards (oui oui) d’euros de budget par an versés à quelques grands groupes que je n’ose même plus qualifier « d’éditoriaux » et un lectorat proportionnellement équivalent à celui du bulletin paroissial de Mouilleron Le Captif (au demeurant charmante bourgade de Vendée avec un patronyme trop choupinou).
Ça n’est pas notre métier
Celle qui surclasse toutes les autres. La vraie raison c’est que notre putain de métier n’est pas d’écrire des articles scientifiques et de remplir des dossiers de demande de subvention qui nous seront refusés plus de trois fois sur quatre (chiffres officiels de l’AERES).
Notre putain de métier c’est d’enseigner, de produire des connaissances scientifiques permettant de mieux comprendre le monde dans lequel nous vivons ET DE PARTAGER CES PUTAINS DE CONNAISSANCES AVEC LES GENS. Pas JUSTE avec nos gentils étudiants ou JUSTE avec nos charmants collègues, AVEC LES GENS.
Notre putain de métier ce n’est pas d’attendre deux putains d’années que d’improbables pairs qui auraient par ailleurs bien mieux à faire – de la recherche ou des cours – aient bien constaté que nous n’écrivions pas n’importe quoi pour nous donner, au bout de deux ans, la permission de voir nos écrits diffusés avec un niveau de confidentialité qui rendrait jaloux les banques suisses et avec un coût d’accès qui … rendrait aussi jaloux les banques suisses.
Parce qu’il y’a ceux qui ont un pistolet chargé, et ceux qui creusent
Et que ceux qui creusent, on les connaît. A commencer par les présidents d’université auxquels j’avais déjà par le passé témoigné de toute mon admiration pour le côté visionnaire de leur immobilisme.
Sans oublier bien sûr tous mes charmants collègues qui, à force de « c’est trop compliqué », « j’ai pas le temps », et autres « c’est pas la priorité » ou « les éditeurs de revues ne veulent pas », ne déposent même pas la version auteur de leurs articles dans des archives ouvertes, et qui mettent donc une hallucinante énergie mortifère à creuser leur propre tombe (ça c’est leur problème) mais hélas, aussi et surtout, la tombe de la diffusion des connaissances et de l’accès aux savoirs.
Parce que tant qu’il y aura des couilles éditoriales en or, y’aura des lames d’Open Access en acier
Je vous avais déjà parlé d’Alexandra Elbakyan. S’il y avait un Panthéon des militants de l’accès aux connaissances scientifiques (et du courage scientifique du même coup), elle siègerait aux côtés d’Aaron Swartz.
Cette femme a créé le site Sci-Hub qui est tout simplement à l’heure actuelle la plus grosse bibliothèque scientifique clandestine du Web, plus de 50 millions d’articles scientifiques, et dont la controverse qu’il suscite ne va pas assez loin.
Bien sûr Elsevier lui colle un procès, bien sûr diverses manipulations plus ou moins légales tentent de faire disparaître ce site, qui heureusement, résiste et résiste encore. Pour s’y connecter actuellement, si l’adresse sci-hub.cc ne répond pas, tentez sci-hub.ac ou carrément l’IP 31.184.194.81 : -)
Open Access Guerilla Manifesto
Parce que ces requins du grand banditisme éditorial sont partout et qu’ils ont bien compris d’où venait le danger. A tel point que l’on apprenait il y a quelques jours qu’Elsevier ,(encore…), avait réussi à racheter une archive ouverte en sciences sociales (et pas l’une des moindres… SSRN). Carrément.
Alors figurez-vous que y’a pas que Martin Luther King qui fait des rêves. Moi aussi j’ai fait un rêve.
- J’ai rêvé que les acteurs publics de la recherche publique (l’Etat, les universités, les présidents d’université, les enseignants-chercheurs, les bibliothèques universitaires) lisaient, adhéraient et appliquaient à la lettre le manifeste pour une guerilla de l’Open Access d’Aaron Swartz.
- J’ai rêvé que plus un centime d’argent public ne soit versé à ces escrocs mais que la totalité dudit argent public soit consacré à développer, construire et soutenir des initiatives comme Sci-Hub ou toute autre forme d’archive ouverte ou de libre accès, que ces initiatives soient légales ou illégales.
- J’ai rêvé que des gens qui disposent majoritairement d’un bac+8 soient capables de comprendre et d’entendre que le fruit de leur travail (de recherche), que leur rôle dans la société (faire avancer les connaissances et mettre ces connaissances à disposition du public), que tout cela était non seulement menacé, mais en train d’être entièrement détruit depuis déjà des dizaines d’années par un système devenu totalement dingue et atteignant un niveau de cynisme ahurissant et piloté par quelques grands groupes qui osent encore se dire « éditoriaux » quand la réalité de leurs pratiques les constitue en autant de mafias.
- J’ai rêvé que des gens qui disposent d’un bac+8, d’un salaire confortable, et d’un temps de cerveau disponible non-entièrement dédié à Coca-Cola, soient capables d’entendre et de comprendre que pour des populations entières sur cette planète, que pour des millions de personnes souffrantes, malades, exploitées ou tout simplement… curieuses, la privatisation des connaissances était littéralement, je dis bien littéralement, mortifère.
Et comme depuis plus de 15 ans que je fais ce rêve, je me suis réveillé avec une putain de gueule de bois, à peine atténuée par la récente médaille d’or de l’innovation attribuée à Marin Dacos.
Bon et là vous vous dites : « ok il est énervé », « ok c’est réjouissant », mais « ok il exagère »
Parce que vous me connaissez hein. Pondéré. Pas sanguin pour deux sous. Raisonnable au-delà des attendus du devoir de réserve. La faconde de la grande muette à moi tout seul. Donc devinette.
Qui a écrit :
« Les éditeurs et les scientifiques insistent sur l’importance cruciale de l’évaluation par les pairs. Nous la dépeignons au public comme si c’était un processus quasi-sacré qui aide à faire de la science notre accès le plus objectif à la vérité. Mais nous savons que le système d’évaluation par les pairs est biaisé, injuste, non fiable, incomplet, facilement truqué, souvent insultant, souvent ignare, parfois bête, et souvent erroné. »
Un punk à chien ? Non, le rédacteur en chef de la revue « The Lancet ».
Qui a écrit :
« Nous avons peu de données quant à l’efficacité réelle du processus, mais nous avons la preuve de ses défauts. En plus d’être peu efficace pour la détection de défauts graves et presque inutile pour la détection des fraudes, il est lent, coûteux, distrait le chercheur de son laboratoire, est très subjectif, tient de la loterie, et peut facilement abuser. Vous soumettez une étude pour un journal. Elle entre dans un système qui est en fait une boîte noire, puis une réponse plus ou moins justifiée sort à l’autre extrémité. La boîte noire est comme la roulette, et les profits et pertes peuvent être grands. Pour un universitaire, une publication dans un journal important comme Nature ou Cell équivaut à toucher le jackpot. »
Un marxiste léniniste ? Non, Richard Smith du Journal of Royal Society of Medicine.
Qui a écrit : qu’il n’enverrait plus jamais d’articles à des revues comme « Nature, Science ou Cell dans la mesure où ces revues à comité de relecture faussent le processus scientifique, et constituent une tyrannie qui doit être brisée. »
Un agitateur anarcho-autonome ? Le prix nobel de médecine 2016.
Résultat en libre accès
Du coup, je me sens un peu moins seul, et pas uniquement du fait de ma schizophrénie. Donc non, je ne publierai plus jamais dans des « revues scientifiques », (et s’il m’arrive de le faire une ou deux fois à titre exceptionnel pour des gens que j’estime intellectuellement ou amicalement, la version intégrale – pas juste la version « auteur » – sera toujours disponible sur ce blog et dans une ou plusieurs archives ouvertes). Et s’il faut pour cela être dans « l’illégalité », j’y serai plutôt deux fois qu’une, et ce ne sera pas la première fois…

Je ne publierai plus jamais dans des revues scientifiques qui ne me permettent pas de mettre simultanément en libre accès le résultat de ma recherche. Et j’espère sincèrement que nous serons de plus en plus nombreux à le faire.
Dix ans de perdus ?
Il y a à peine plus de dix ans, le 15 mars 2005 très précisément, un autre universitaire avait pris un tel engagement. Celui de « ne plus jamais publier dans des revues scientifiques qui ne me permette pas au minimum les libertés d’une licence Creative Commons Attribution NonCommercial. »
Ce type, c’était Lawrence Lessig. Sur son blog. Lawrence Lessig. Le même qui, lors de l’enterrement d’Aaron Swartz après son suicide, prononçait avec une immense peine ces quelques mots, le 12 Janvier 2013 :
« Mais quiconque affirme qu’il y a de l’argent à faire avec un stock d’articles scientifiques est soit un idiot, soit un menteur. »
Dix ans plus tard je vais m’autoriser à aller un peu plus loin : quiconque affirme aujourd’hui qu’en acceptant de publier dans des revues scientifiques sans systématiquement déposer son texte dans une archive ouverte et/ou avec une licence d’attribution non-commerciale, ignore, ou feint d’ignorer, sa part de responsabilité dans la situation catastrophique de privatisation de la connaissance que mettent en œuvre quelques grands groupes éditoriaux à l’échelle de la planète. Celui-là est à la fois un idiot, un menteur et surtout un irresponsable.
Alors je ne publierai plus jamais d’articles dans des revues scientifiques. Et je continuerai de m’opposer, de manière légale ou illégale, à toute forme de privatisation de la connaissance. Et vous ?

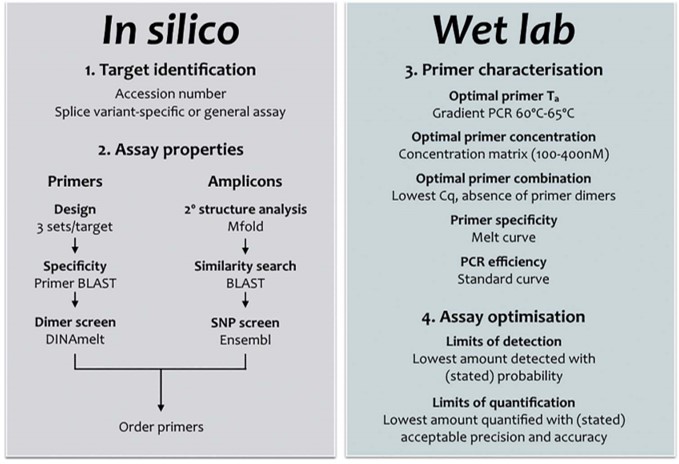
La qPCR est une méthode permettant de doser la quantité d’acides nucléiques ciblés introduits dans une réaction de PCR. Pour des raisons de rapidité, de sensibilité et de coût, souvent l’option de travailler avec un agent intercalant (sans sonde) est choisie. La bonne vieille qPCR SybGreen nécessitant le seul design d’une paire d’amorces…
Simple ? Pas nécessairement, car cette approche, certainement plus que la version qPCR Taqman nécessite un travail in silico et de validation/optimisation expérimentales comme passages obligés. C’est ce que montre la publication de Stephen Bustin et Jim Huggett dans Biomolecular Detection and Quantification. Cette publication incontournable pour les férus de qPCR SybrGreen est un beau travail pour lequel la publication vous est mise à disposition en cliquant ci-dessous. On attend ardemment une déclinaison Taqman, HRM, MolecularBeacon de ce type de revues permettant de formaliser des procédures visant à optimiser l’approche d’optimisation.
La quantification par qPCR SybrGreen suppose une relation linéaire entre le logarithme de la quantité initiale introduite en PCR et la valeur Cq obtenue lors de l’amplification. Ceci permet de calculer l’efficacité d’amplification d’un test et de borner ses limites de détection et de quantification. Les caractéristiques d’un test qPCR (bien) optimisé sont les suivantes:
• Une excellentissime spécificité révélée par un pic unique lors de l’établissement de la courbe de fusion
• Une efficacité d’amplification élevée (95-105%)
• Une courbe étalon linéaire (R2 > 0,980)
• Une bonne répétabilité
• Peu ou prou de dimères d’amorces
Pour paraphraser la conclusion de l’article, afin de finir par convaincre de lire cet « essentiel » de la qPCR :
La conception, le design d’une PCR est souvent au cœur de tout projet de recherche visant à quantifier les acides nucléiques. Il doit être réalisé avec soin, mais peut être simplifié en suivant un flux de travail simple, comme décrit ci-dessus (cf. diagramme workflow design qPCR).
Cela signifie généralement une spécificité absolue, l’absence de structures en épingle à cheveux ou de potentielles dimérisations croisées. Une bonne conception des essais doit tenir compte de la structure de l’amplicon (paramètre souvent négligé) et veiller à ce que les cibles de l’amorce soient exempts de structure secondaire. Il existe de nombreuses opinions et lignes directrices; une recherche sur Internet pour les termes « qPCR Assay Design » renvoie 695.000 pages. Cependant, bon nombre de ceux-ci sont basés sur des mythes ou peuvent être appropriés pour la PCR mais nécessitent des modifications subtiles (ou moins subtiles) pour être utilisés pour développer une qPCR. Chaque « nouveau » dosage doit être correctement validé, la validation in silico servant de filtre initial pour éliminer des designs ne permettant pas d’aboutir à une bonne qPCR. L’optimisation et la validation empirique sont une partie essentielle, mais souvent négligée, de toute expérience qPCR. Cela s’applique aussi bien aux essais nouvellement conçus qu’aux essais obtenus en reprenant des amorces issues d’une publication, par exemple. Avec tant d’essais prêts à l’emploi, on peut se demander pourquoi quelqu’un voudrait se donner la peine de concevoir un autre essai. D’autant plus que l’on a l’impression que la conception de son propre test est beaucoup plus complexe et peu commode que de simplement l’acheter à un fournisseur commercial, qui en tout cas aura validé chacun de ses tests. Cette perception est erronée pour deux raisons:
1° il se peut que les amorces commerciales ou les conditions d’analyse n’aient pas été validées ou optimisées de façon expérimentale.
2° on ne peut pas présumer qu’un ensemble d’amorces produira les mêmes résultats dans des conditions expérimentales différentes puisque la performance du dosage peut varier selon les méthodes d’extraction utilisées pour purifier les acides nucléiques.
Qui sommes nous?
Christophe Audebert [@]
 En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
En charge de la plateforme génomique
du département recherche et développement
de la société Gènes Diffusion .
Renaud Blervaque [@]
 Biologiste moléculaire, chargé d'études génomiques.
Biologiste moléculaire, chargé d'études génomiques.
Gaël Even [@]
 Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Responsable bioinformatique au sein
du département recherche et développement de la société Gènes Diffusion.
Catégories
- Analyse de données (14)
- Automatisation (5)
- Bioinformatique (27)
- Biologie (56)
- biologie transverse (35)
- Biotechnologie (30)
- Chronique littéraire (8)
- Comparatif (6)
- Diagnostic (8)
- Economie (17)
- Epidemiologie (2)
- Evénement (17)
- Formation (3)
- Gestion de projet (5)
- Grille de calcul (1)
- Intégration (5)
- Logiciels (8)
- Médecine (14)
- politique de la recherche (17)
- Recherche (21)
- Séquençage (70)
- Séquenceur (39)
- Uncategorized (25)
- Workflow (4)
Accès rapide aux articles
- Covid-19 : zoom sur les vaccins
- Comment l’ADN pourrait être le stockage de données du futur
- COVID-19 : la data visualisation au service de la science
- COVID-19 : des explications et un point d’étape au 20 février 2020
- Pour mettre du vin dans son eau
- Des petits trous, toujours des petits trous…
- Qui serait candidat ?
- Un robot pour analyser vos données…
- Monde de dingue(s)
- L’art et la manière de développer une qPCR
- Un MOOC Coursera sur le WGS bactérien
- Chercheurs & enseignants-chercheurs, l’art du multitâche.
- Un jeu de données métagénomiques
- Facteur d’impact
- Microbiote & smart city : juxtaposition de tendances
Accès mensuels
- février 2021 (1)
- décembre 2020 (1)
- février 2020 (2)
- septembre 2019 (1)
- avril 2018 (2)
- décembre 2017 (1)
- novembre 2017 (2)
- juillet 2017 (2)
- juin 2017 (5)
- mai 2017 (4)
- avril 2017 (3)
- mars 2017 (1)
- janvier 2017 (2)
- décembre 2016 (3)
- novembre 2016 (4)
- octobre 2016 (2)
- septembre 2016 (2)
- août 2016 (3)
- juillet 2016 (2)
- juin 2016 (4)
- mai 2016 (3)
- mars 2016 (1)
- novembre 2015 (2)
- avril 2015 (1)
- novembre 2014 (1)
- septembre 2014 (1)
- juillet 2014 (1)
- juin 2014 (1)
- mai 2014 (1)
- avril 2014 (1)
- mars 2014 (1)
- février 2014 (3)
- janvier 2014 (1)
- décembre 2013 (5)
- novembre 2013 (2)
- octobre 2013 (2)
- septembre 2013 (1)
- juillet 2013 (2)
- juin 2013 (2)
- mai 2013 (4)
- avril 2013 (2)
- mars 2013 (1)
- février 2013 (3)
- janvier 2013 (2)
- décembre 2012 (2)
- novembre 2012 (2)
- octobre 2012 (2)
- septembre 2012 (2)
- août 2012 (1)
- juillet 2012 (3)
- juin 2012 (5)
- mai 2012 (5)
- avril 2012 (6)
- mars 2012 (6)
- février 2012 (8)
- janvier 2012 (6)
- décembre 2011 (5)
- novembre 2011 (6)
- octobre 2011 (6)
- septembre 2011 (7)
- août 2011 (5)
- juillet 2011 (8)
Pages